Rastreio virtual guiado por farmacóforo para reaproveitamento de medicamentos
O reaproveitamento de medicamentos é uma estratégia estabelecida há muito tempo para identificar novas aplicações para terapêuticas aprovadas ou em investigação que estão além do escopo da indicação médica original. 1 Desde o reposicionamento fortuito de um medicamento projetado para hipertensão e angina para se tornar a infame "pílula azul" da Pfizer, comumente referida como Viagra, o reaproveitamento de medicamentos não tem sido tão proeminente aos olhos do público. A urgência global para a terapêutica para tratar COVID-19 trouxe esta estratégia de drogas mais uma vez à atenção da massa.
A modelagem de proteínas 3D inclui vários métodos de modelagem e simulação molecular para reaproveitamento de drogas. Um post anterior desta série explorou o docking molecular como um método para identificar possíveis inibidores que se ligam à protease principal do SARS-CoV-2. Pode uma alternativa in silico método, como a modelagem farmacóforo, consegue resultados comparáveis e fornece evidências adicionais que apóiam a seleção de um candidato em detrimento de outro?
Tomando uma rota alternativa
A modelagem de farmacóforo fornece uma abstração das características moleculares que são necessárias para o reconhecimento de um ligante por uma proteína alvo. Sua representação de interações moleculares e ligação fornece uma perspectiva contrastante para métodos de simulação clássicos.
Obtivemos um conjunto de dados inicial de várias estruturas de proteína protease principal SARS-CoV-2 da Diamond Light Source. 2 Alinhamos essas estruturas e, em seguida, usamos o protocolo de ‘Geração de Farmacóforo de Interação’ disponível no BIOVIA Discovery Studio para gerar farmacóforos que representam as interações de não ligação de cada complexo receptor-ligante. O número total de recursos nos farmacóforos variou de dois recursos para as estruturas cristalinas 5R80 e 5R7Y a um modelo de nove recursos para 6LU7. Nós mesclamos os farmacóforos dos complexos individuais em um único modelo e editamos recursos intimamente agrupados. O modelo final incluiu 14 recursos que representam contatos intermoleculares entre a protease e possíveis ligantes de pequenas moléculas.
Posteriormente, executamos in silico mutagênese de varredura de alanina nos resíduos do sítio ativo de todos os complexos para identificar quais resíduos reduziram a afinidade de ligação (ponto de acesso) desse complexo proteína-ligante quando mutado. De todos os complexos, identificamos oito resíduos (HIS41, MET49, ASN142, HIS163, MET165, PRO168, GLN189 e GLN192) como pontos ativos em pelo menos um complexo e três resíduos que eram pontos ativos em pelo menos quatro complexos. No modelo de farmacóforo de 14 recursos, seis recursos corresponderam a interações com um dos oito resíduos de ponto de acesso. Um segundo conjunto de dados de M PRO não covalente complexos de ligantes lançados pela Diamond Light Source 2 revelou uma série de modos de ligação diferentes, levando-nos a separar os seis principais recursos do farmacóforo em dois grupos para uso na próxima etapa da triagem virtual.
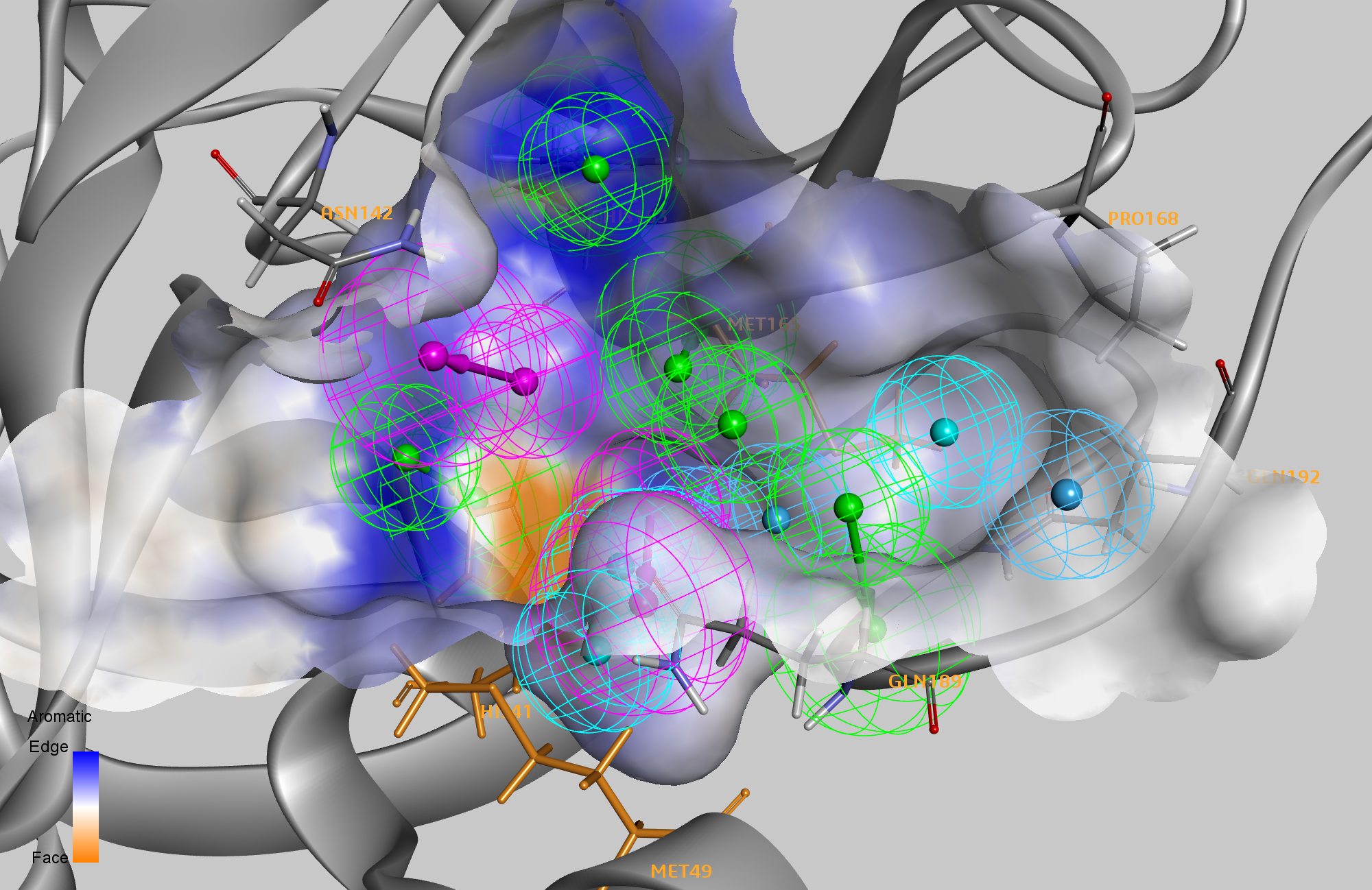
Agora usamos este modelo de farmacóforo para realizar a triagem virtual de uma biblioteca selecionada contendo 2.650 drogas aprovadas pela FDA que têm várias confirmações de ligante pré-geradas para pesquisa rápida. Nós rastreamos esses ligantes contra o farmacóforo de 14 recursos, com um requisito de que pelo menos um recurso de cada um dos dois grupos necessários precisava corresponder, bem como pelo menos dois recursos dos oito recursos restantes, para identificar um acerto. Isso é para permitir a exploração diversa de diferentes modos de ligação dos ligantes, ao mesmo tempo que garante que apenas as poses que podem formar pelo menos duas interações com resíduos significativos identificados na mutagênese de varredura de alanina sejam retidas. O algoritmo explora essencialmente mais de 12.000 combinações possíveis de farmacóforo. Em seguida, minimizamos os resultados de melhor ajuste para cada combinação de farmacóforo dentro da proteína. Finalmente, determinamos as energias de ligação das poses derivadas de farmacóforo otimizadas usando CHARMm com um modelo de solvente implícito de Born generalizado usando Volume Molecular (GBMV). Este método é efetivamente um cálculo de encaixe molecular (como em nosso blog anterior), mas aqui usamos nosso modelo de farmacóforo otimizado para restringir e filtrar os resultados encaixados.
Calculamos as interações não vinculadas de cada pose e filtradas para focar apenas nas poses com interações com quatro resíduos-chave - HIS41, MET49, CYS145 e MET165. HIS41, MET49 e MET165 foram os três resíduos comuns a pelo menos quatro complexos identificados anteriormente, e CYS145 é o segundo resíduo na díade catalítica HIS41 / CYS145 significativa encontrada em cada subunidade do homodímero de protease principal. Classificamos os resultados com a energia livre de ligação calculada para identificar os dez principais candidatos plausíveis para exploração posterior. Ritonavir foi o único ligante comum identificado aqui e no blog anterior. O ritonavir teve a sétima melhor energia livre de ligação e também foi anteriormente classificado em sétimo no estudo de docking. Ritonavir está atualmente passando por uma série de ensaios clínicos para COVID-19. 3
Vídeo 1 :Melhor pontuação de pose para Ritonavir interagindo com HIS41, MET49, SER144, CYS145, MET165, GLU166 e GLN189.
O ligante de melhor pontuação no método apresentado aqui foi Montelukast, um antagonista do receptor de leucotrieno cisteinil. Um artigo publicado recentemente levantou a hipótese de seu uso para limitar a progressão da doença na infecção por COVID-19. 4
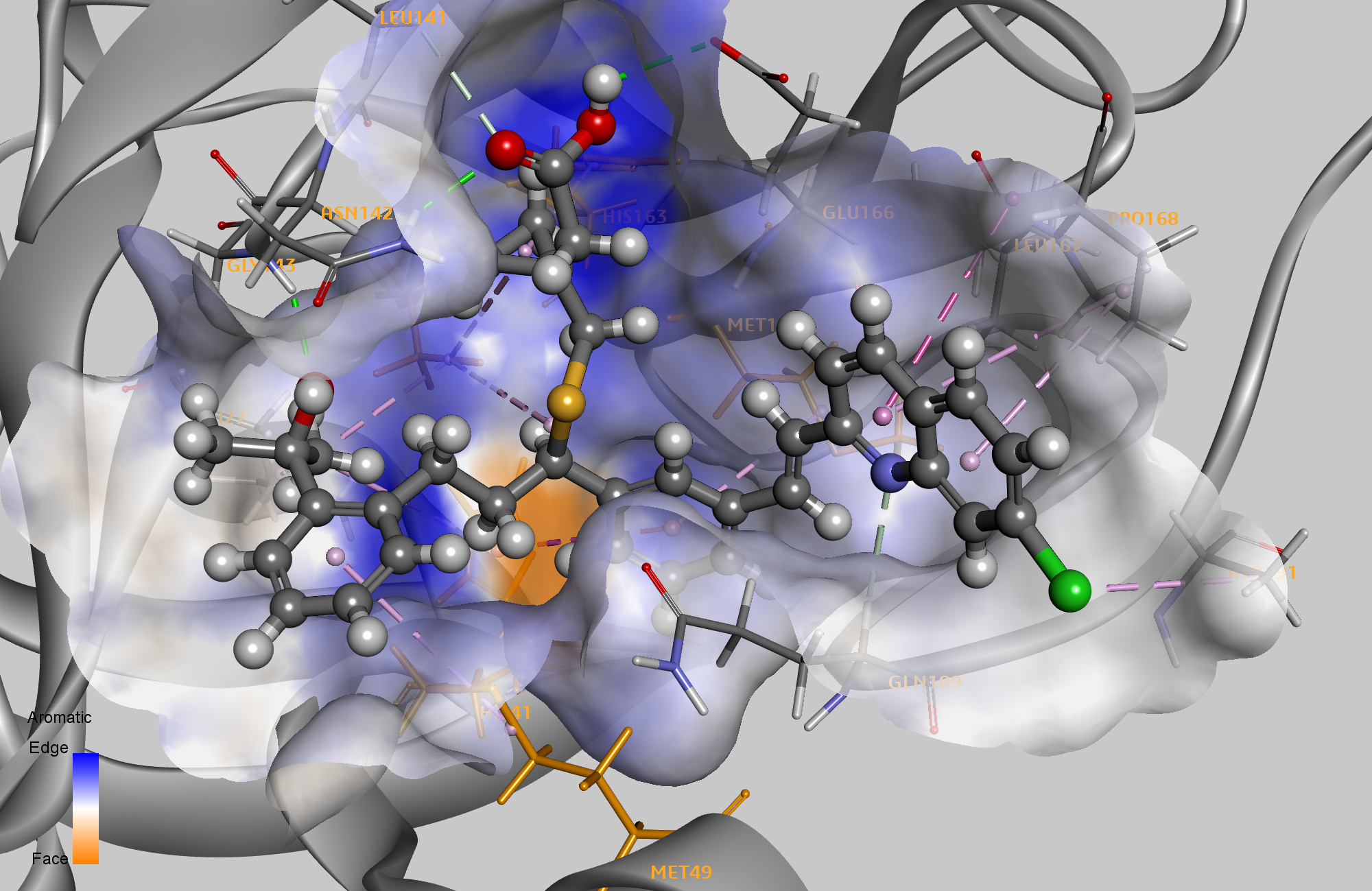
Três compostos adicionais com maior pontuação incluíram os medicamentos Telmisartan, Moexipril e hidroxicloroquina. O potencial de todos esses compostos como drogas reaproveitadas para COVID-19 foi recentemente analisado, 5 com a hidroxicloroquina gerando mais polêmica. Uma diferença adicional entre as dez principais listas de acertos dos dois métodos é a diversidade dos alvos de drogas incluídos. Os dez principais resultados de docking incluem sete inibidores da protease do HCV ou HIV. A lista de acertos priorizados por farmacóforo inclui medicamentos com sete classes-alvo diferentes.
Nossa triagem virtual derivada de farmacóforo produziu uma lista de acertos priorizada incluindo vários novos medicamentos COVID-19 potenciais não identificados em nosso trabalho de acoplamento anterior. Com esta abordagem, demonstramos a utilização de in silico mutagênese de varredura de alanina como uma técnica útil para refinar o modelo de farmacóforo. Também impusemos a exigência de que certos recursos estejam presentes para um ajuste durante o processo de triagem. Os resultados desses dois estudos não são diretamente comparáveis - nem tanto devido às diferenças nos algoritmos básicos, mas sim devido à implementação de diferentes estratégias de suporte.
A breve comparação dos resultados entre os dois métodos mostrou pouca sobreposição de pistas para enfocar. Pode-se dizer que a semelhança do ritonavir identificada por ambos os métodos fornece evidências para apoiar a seleção desse candidato para um estudo posterior. Estudos anteriores demonstraram que o uso de um consenso de funções de pontuação de docking proteína-ligante melhora a identificação de candidatos a medicamentos putativos. 6 Por esse motivo, os pesquisadores poderiam usar os compostos de melhor pontuação exclusivos de cada método para priorizar os compostos para testes experimentais.
Em conclusão, a triagem virtual derivada de farmacóforo fornece um método suplementar e complementar para docking, contribuindo para um consenso e maior confiança na seleção de candidatos para validação experimental. Além da urgência de fornecer um candidato a medicamento para reaproveitamento, este método conduzido por farmacóforo também pode identificar ligantes que são mais diversos para otimização de lead subsequente.
biológico
- Adesivo de nicotina
- Inventário virtual e impressão 3D:a necessidade de segurança
- C# para loop
- C para Loop
- Nanofibras e filamentos para entrega aprimorada de drogas
- Projeto terapêutico generativo
- Reaproveitamento de drogas
- Universal Robots lança a 'maior conferência virtual do mundo' para robôs colaborativos
- Fabricação de PCB para 5G
- Micro-robôs impressos em 3D prometem entrega de medicamentos



