Controle da translocação do DNA por meio de nanoporos de estado sólido
Resumo
Em comparação com o status dos bio-nanoporos, ainda existem vários desafios que precisam ser superados antes que os nanoporos de estado sólido possam ser aplicados no sequenciamento de DNA comercial. Baixa resolução espacial e baixa resolução temporal são os dois maiores desafios. Devido às restrições no comprimento dos nanoporos e nas propriedades da superfície dos nanoporos de estado sólido, ainda há espaço para melhorar a resolução espacial. Enquanto isso, a translocação do DNA é muito rápida sob uma força elétrica, o que resulta na aquisição de poucos pontos de dados válidos. A resolução temporal de nanoporos de estado sólido pode, portanto, ser aumentada se a velocidade de translocação do DNA for bem controlada. Nesta mini-revisão, resumimos brevemente os métodos de melhoria da resolução espacial e nos concentramos em métodos controláveis para promover a resolução da detecção de nanoporos. Além disso, fornecemos uma perspectiva sobre o desenvolvimento do sequenciamento de DNA por nanoporos.
Introdução
Nas últimas décadas, muito progresso foi feito na aplicação de sequenciamento de DNA para ler as sequências de bases no genoma [1, 2]. Para desenvolver medicamentos personalizados, os pesquisadores têm buscado um método de sequenciamento de DNA mais rápido e barato, onde os medicamentos alvo e os tratamentos médicos podem ser aplicados a um indivíduo especificamente [3, 4]. Como a tecnologia nanopore foi usada na detecção de DNA [5], ela foi considerada um método eficaz para o sequenciamento de última geração [6, 7]. A tecnologia Nanopore é uma plataforma promissora para identificar detectores de molécula única e altamente sensíveis para DNA [5] ou RNA [8]. No esquema básico de detecção, uma câmara eletroquímica é separada em dois reservatórios (compartimentos cis e trans) por uma membrana fina com um nanoporo [9], que conecta a solução condutora e o analito na câmara eletroquímica. Ao aplicar uma voltagem através da membrana, os íons de eletrólito fluem através do nanopore de estado sólido e formam uma corrente de poro, que é medida usando um patch clamp configurado com eletrônicos ultrassensíveis associados. Quando uma molécula ou complexo molecular passa pelo nanopore, o analito pode excluir alguns íons do volume definido pelo nanopore, o que pode ser detectado monitorando breves mudanças na corrente. Tanto a partir do tempo de residência (tempo de permanência) dentro do nanoporo quanto da assinatura de amplitude da corrente, podem ser obtidas informações sobre as moléculas. A resolução espacial do sequenciamento de nanopore é determinada pelas dimensões do nanopore, o que sugere que ele pode ser usado como um único sensor para pequenos objetos moleculares, resultando em uma assinatura de corrente detectável. Além disso, os sensores nanopore são fáceis de integrar em dispositivos lab-on-a-chip altamente portáteis e miniaturizar [10].
Um progresso significativo foi feito no sequenciamento de DNA por nanoporos, como nanoporos de estado sólido [2, 11] e nanoporos de proteínas [2, 7]. O sequenciamento de DNA por nanoporos de proteína foi alcançado [7]. No entanto, os sistemas de nanoporos de proteínas têm limites para estudar moléculas biológicas. Existem menos restrições com nanoporos de estado sólido em comparação com nanoporos biológicos / proteicos. Essas dificuldades podem ser superadas por nanoporos de estado sólido. Comparados com os nanoporos de proteína, eles são funcionais em faixas mais amplas de temperaturas, tensões e condições de solvente e podem ser ajustados em diâmetro com precisão sub-nanométrica. Eles são promissores para aplicação em tecnologia de próxima geração para sequenciamento de DNA.
Muitos nanoporos de estado sólido de diferentes materiais e estruturas foram feitos para sensores de DNA. No entanto, o sequenciamento de DNA não é alcançado por nanoporos de estado sólido. Para os sensores nanoporos de estado sólido, há a necessidade de superar dois grandes obstáculos, no que diz respeito à sua resolução espacial e resolução temporal, antes de sua aplicação comercial para sequenciamento de DNA. A dificuldade em relação à resolução espacial é que o nanoporo de estado sólido pode distinguir o espaçamento minúsculo entre dois nucleotídeos vizinhos para atingir a resolução de base única. O obstáculo da resolução temporal é que a translocação do DNA é muito rápida sob uma força elétrica, o que resulta na aquisição de muito poucos pontos de dados válidos por patch clamps existentes ou outros sistemas de aquisição de sinal. Esta mini-revisão apresenta uma visão geral dos vários métodos para melhorar a resolução espacial e a resolução temporal da detecção de DNA de nanoporos em estado sólido. Esta mini-revisão também se concentra mais em métodos de desaceleração da translocação de DNA através de nanoporos de estado sólido.
Resolução Espacial
Em 2001, nanoporos de estado sólido de nitreto de silício foram relatados pela primeira vez por Li et al. [12]. Vários nanoporos de estado sólido foram demonstrados para detecção molecular de DNA, como óxido de silício [13], silício [14], Al 2 O 3 [15], e HfO 2 [16]. Embora os nanoporos de estado sólido possam, em última análise, ser robustos às condições químicas e mecânicas, eles têm algumas limitações, como a baixa resolução espacial. Devido à espessura dos materiais, dezenas de bases podem passar por nanoporos de estado sólido por vez. Atualmente, o nanoporo de nitreto de silício mais fino é de 3 nm, o que ainda não distingue os quatro tipos de base [17].
Curiosamente, a espessura de uma única camada de material bidimensional (2D) é de aproximadamente 3,0-11,0 Å, que é comparável ao espaçamento entre dois nucleotídeos vizinhos ao longo do ssDNA (3,2-5,2 Å) [18]. Membranas bidimensionais, como grafeno (3,4 Å [19]), MoS 2 (6,5 Å [18]), WS 2 (7 Å [20]) e h-BN (11 Å [21]), demonstraram detectar translocações de DNA [21,22,23] por causa de sua alta razão sinal-ruído e resolução espacial. É claro pela sua resolução espacial que esses materiais podem ser usados para detecção de DNA. Além disso, técnicas de crescimento reproduzíveis e procedimentos de transferência em larga escala tornam possível fabricar poros sub-nanométricos em membranas 2D em grande escala.
O grafeno é uma folha atomicamente fina de átomos de carbono organizados em uma estrutura em favo de mel bidimensional [24]. Os pesquisadores demonstraram que moléculas de DNA individuais em solução podem ser detectadas e caracterizadas com nanoporos de grafeno [22, 25]. No entanto, existem fortes interações hidrofóbicas entre os nucleotídeos do DNA e o grafeno, e o DNA vai obstruir severamente e grudar nos nanoporos de grafeno, o que teria um impacto marcante na velocidade de translocação [26]. Modificado com os grupos hidrofílicos [26] ou revestido, um material hidrofílico [25] no grafeno poderia melhorar a hidrofilia do grafeno e evitar que o DNA grude em sua superfície. Infelizmente, tanto a modificação com grupos hidrofílicos quanto o revestimento com materiais hidrofílicos irão aumentar a espessura do filme suspenso, levando ao aumento da espessura dos nanoporos, diminuindo assim a resolução espacial do grafeno.
O dichalcogeneto de metal de transição em camadas é outro material 2D, que inclui MoS 2 [18, 27] e WS 2 [20]. Alta relação sinal-ruído (SNR> 10) e aumento de cinco vezes do sinal da corrente iônica foram detectados quando o DNA foi translocado através do MoS 2 membrana nanopore [18]. Enquanto isso, MoS 2 é hidrofílico, portanto, nenhum tratamento de superfície especial é necessário para evitar a interação hidrofóbica entre o DNA e sua superfície. O outro material, WS 2 tem um gap direto de 2,1 eV [28], e sua emissão de fotoluminescência (PL) é mais forte do que no bem estudado MoS 2 [29]. Danda et al. [20] fabricou um WS 2 nanopore e demonstrou a obtenção de tamanho de nanopore controlado atomicamente usando pulsos de luz curtos, o que pode ter efeitos positivos em nanoporos de estado sólido para detecção de DNA.
Além disso, Liu et al. [21] relataram o primeiro experimento de translocação de DNA através de nanoporos h-BN. Semelhante ao grafeno, o h-BN tem baixa hidrofilicidade, o que fará com que o DNA bloqueie os nanoporos. Posteriormente, Zhou et al. [23] melhorou com sucesso a hidrofilicidade dos nanoporos h-BN, aproveitando a antioxidação e integridade do material h-BN após o tratamento com ozônio UV. A natureza isolante de h-BN pode exibir durabilidade mais notável e propriedades isolantes em solução de alta força iônica do que em grafeno. É um candidato competitivo para obter resolução de base única em estruturas nanopore superfinas.
O uso de materiais bidimensionais pode aumentar potencialmente a resolução espacial do dispositivo para atingir a resolução de nucleotídeo único. Embora experimentos de detecção de DNA para vários materiais bidimensionais tenham sido relatados, ninguém relatou a realização de sequenciamento de DNA de nanoporos em estado sólido. A resolução temporal do sequenciamento de nanoporos também é um desafio.
Resolução Temporal
A velocidade de translocação do DNA através de um nanoporo de estado sólido é muito rápida, de até 0,01–1 μs / base [30], levando a poucos dados eficazes coletados por patch clamps comerciais. Como tal, não é possível distinguir todas as bases com base no sinal de corrente de bloqueio. As velocidades de translocação de DNA de nanoporos de estado sólido de materiais 2D, como grafeno, MoS 2 , WS 2 , e h-BN, são mostrados na Fig. 1. Idealmente, a velocidade de translocação do DNA em um nanopore deve ser de 1–100 bp / ms para permitir a gravação satisfatória do sinal de cada nucleotídeo [32].
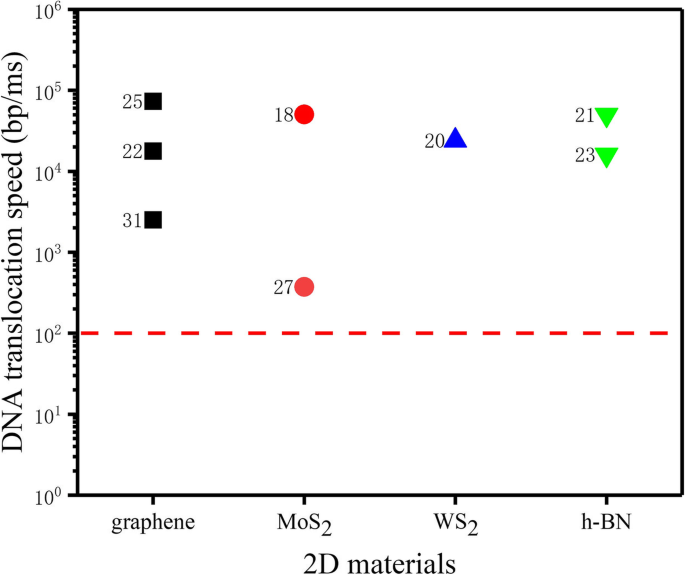
Velocidade de translocação de DNA de nanoporos de estado sólido 2D [18, 20,21,22,23, 25, 27, 31]. A linha vermelha pontilhada indica uma taxa de translocação de DNA de 100 bp / ms
Neste contexto, diminuir a velocidade de translocação do DNA é um objetivo importante perseguido por muitos pesquisadores. Vários métodos foram desenvolvidos para desacelerar a translocação de DNA para melhorar a resolução temporal da detecção de nanoporos em estado sólido. O método usual é alterar a influência de fatores experimentais, como temperatura [33], viscosidade do eletrólito [27], voltagem motriz [34], concentração de íons [35] e densidade de carga superficial de um nanoporo [36], alterando o DNA translocação através dele. Wanunu et al. [33] se concentrou em desacelerar a translocação de dsDNA através de nanoporos de estado sólido, alterando a temperatura, voltagem e comprimento do DNA. Além disso, Feng et al. [27] mostraram que um sistema de gradiente de viscosidade, baseado em líquidos iônicos à temperatura ambiente, pode ser usado para controlar a dinâmica da translocação de DNA através de MoS 2 nanoporos, e demonstraram que a alta viscosidade de líquidos iônicos à temperatura ambiente fornece velocidades de translocação de nucleotídeo único ideais de 373 bp / ms.
Embora muitas abordagens tenham o potencial de reduzir a velocidade de translocação de DNA e facilitar a detecção de corrente iônica, elas ainda não podem atender aos requisitos para o sequenciamento de DNA. Portanto, é necessário desenvolver métodos mais radicais para controlar a passagem do DNA pelos nanoporos. Aqui, discutimos os métodos de controle da estrutura dos nanoporos e do movimento quantitativo do DNA para melhorar a resolução temporal.
Sistema de dois nanoporos
Os pesquisadores usaram sistemas de dois nanoporos para manipular as translocações de moléculas de DNA, que podem detectar controladamente a mesma molécula muitas vezes. Dois nanoporos empilhados, separados por um compartimento de cavidade do tamanho de um micrômetro [37] (Fig. 2a), podem capturar as moléculas de DNA por um determinado período de tempo e liberá-las de maneira controlada. A dinâmica das moléculas de DNA pode ser deduzida dos sinais dos dois poros por análise de correlação, que fornece prova elétrica direta para a translocação. Além disso, por causa das barreiras de entropia do sistema de nanoporos de duas camadas, o movimento browniano das moléculas de DNA pode ser confinado, o que pode melhorar a precisão do sequenciamento de DNA com nanoporos. Em comparação com as técnicas de detecção de molécula única, as moléculas de DNA podem ser medidas várias vezes em sistemas de nanoporos de duas camadas, organizando em série vários poros, em vez de passá-los para frente e para trás através de um único poro [39].
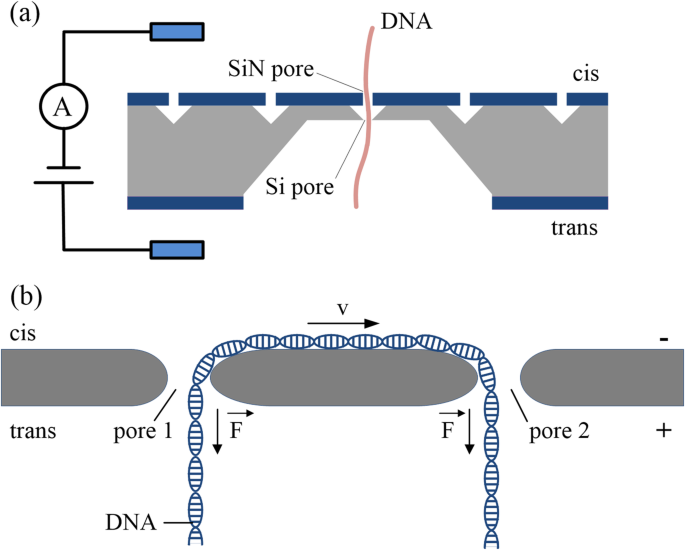
a Esquema do sistema de nanoporos de duas camadas usado para detecção de DNA de nanoporos [37]. b Esquema do sistema de nanopore duplo usado para detecção de DNA de nanopore [38]
Os sistemas de nanoporos duplos fornecem outro método para controlar o transporte molecular e conectar moléculas de forma eficiente entre dois poros; eles são uma abordagem mecanicista sem rótulo para manipulação de DNA [40]. Em um sistema de nanoporos duplos, existem dois nanoporos adjacentes e endereçáveis de forma independente dentro da mesma membrana de estado sólido. Durante a passagem eletroforética de uma molécula de DNA através de um dos nanoporos, uma única molécula de DNA pode ser capturada em ambos os poros, levando a um "cabo de guerra" entre os dois nanoporos [38] (Fig. 2b). Portanto, forças são aplicadas às diferentes extremidades de uma molécula de DNA, o que desacelera e até mesmo interrompe totalmente seu movimento. O sistema de nanoporos duplos abre um novo caminho para o aprisionamento mecânico de DNA em nanoporos de estado sólido, e é uma técnica promissora para medir uma ampla gama de biomoléculas com as vantagens de ser livre de marcadores e ter um alto sinal. relação ao ruído e baixo custo. Ele pode confinar e prender com eficiência as moléculas de DNA para retardar a translocação do DNA e também pode ser usado para estudar a física desse cabo de guerra em nanoescala sobre o DNA [41].
Nanopore de armadilha óptica
Os nanoporos de armadilhas ópticas permitem que as pinças ópticas prendam partículas com menos de dezenas de nanômetros de tamanho. Ele torna possível o aprisionamento óptico de proteínas [42], fragmentos de DNA [43] e outras biomoléculas [44], bem como pequenos vírus [45]. A teoria básica por trás dos nanoporos de armadilhas ópticas é uma armadilha óptica de ação traseira auto-induzida [46]. O feixe de laser focado na região do arranjo de nanoporos formará um campo de luz local de alta densidade de potência na borda da camada de metal no buraco. Conforme uma partícula se move entre o campo de luz local, ela pode causar uma grande mudança na transmissão óptica local, que por sua vez produzirá uma grande força óptica e dielétrica na partícula. Uma estrutura de nano-furo duplo é usada para quebrar o gargalo de tamanho das partículas capturadas. Muhammad et al. [47] demonstraram o uso potencial de nanoporos de armadilhas ópticas com sílica de 20 nm e nanopartículas de Au. A forma de haltere do nanohole duplo foi moída no filme Au, e um nanoporo de 25 nm foi perfurado através do Si x suspenso N y membrana no meio do nanohole duplo, como mostrado na Fig. 3a. Força eletroforética conduzindo as nanopartículas através do nanopore, ao passar pela borda do orifício, a força plasmônica de retro ação auto-induzida existente entre as pontas do nanoofuro duplo se opõe à força eletroforética, reduzindo a velocidade das nanopartículas. Os resultados mostraram que o aprisionamento óptico estendeu o tempo de translocação eletroforética em quatro ordens de magnitude. Kim et al. [43] realizaram a captura óptica de DNA de plasmídeo e DNA de lambda usando as estruturas nanoplasmônicas de nanoporos individuais. A tecnologia tem aplicações potenciais para detecção de DNA e vários campos de luz locais podem ser configurados para realizar a detecção paralela. No entanto, a oscilação da corrente de íons de alta frequência pode afetar os resultados de detecção atuais do DNA. Isso pode ser devido à competição entre forças eletroforéticas e auto-induzidas de ação reversa, fazendo com que as nanopartículas flutuem para cima e para baixo através da boca do nanoporo.
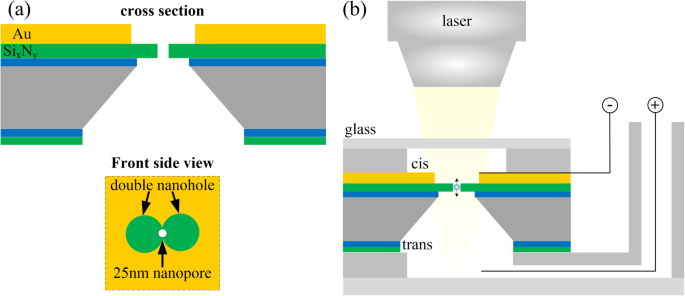
a Esquema do chip nanoporo de armadilha óptica [47]. b Esquema do sistema de ação traseira auto-induzida
Pinças ópticas
Pinças ópticas podem ser usadas para controlar a translocação molecular através de nanoporos e têm sido comumente usadas nos últimos anos. Em 2006, Keyser et al. [48] demonstrou pela primeira vez um cabo de guerra molecular entre as forças eletroforéticas e mecânicas, aplicando pinças ópticas para controlar a translocação de DNA através de SiN x nanoporos. Este sistema atua como uma simples mola Hooke e a força de tensão no cordão pode ser calculada com base na lei de Hooke: F ot =−k armadilha Z , onde F ot é a força óptica, k armadilha é a rigidez da armadilha ao longo da direção de deslocamento, e Z é a deformação linear do cordão [48]. O método de pinça óptica, que captura um grânulo de poliestireno preso por DNA no cruzamento de um feixe de laser focado, pode manipular o grânulo de poliestireno preso por DNA em três dimensões e tem uma faixa de pico newton de sensibilidade à força, como mostrado na Fig. 4a . Para prender o DNA em translocação dentro do nanoporo, a força de tensão foi ajustada para equilibrar a força do campo elétrico ( F el ) no DNA. Portanto, a tensão pode ser usada para reduzir a velocidade de translocação do DNA e puxar a molécula de DNA para fora de um nanoporo [52]. Este sistema permite a amostragem espacial simultânea e medições de força de alta resolução de ácidos nucléicos e proteínas, que alcançou um progresso significativo no sequenciamento de DNA. No entanto, a técnica das pinças ópticas sofre de várias dificuldades fundamentais. Primeiro, é difícil aumentar a escala para um grande número de nanoporos. Em segundo lugar, o aquecimento causado pelo laser em pinças ópticas impacta fortemente na corrente iônica através de um nanopore e no nível de ruído, exigindo que o cordão opticamente preso esteja a vários micrômetros de distância do nanopore [53].
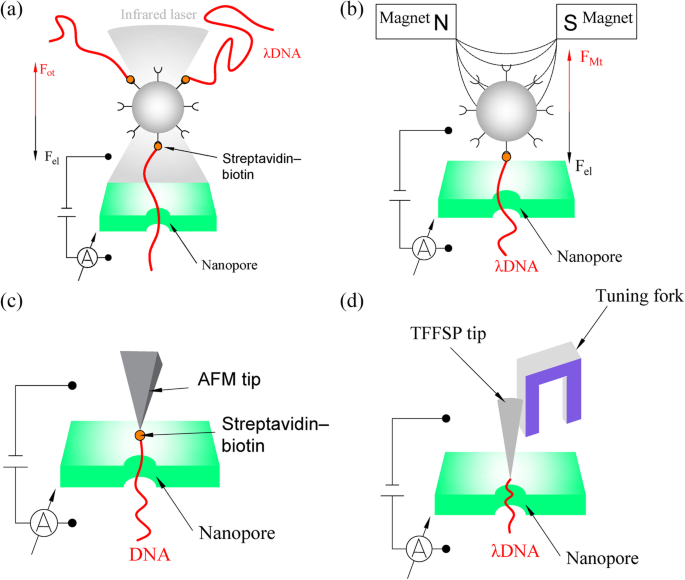
a Esquema de pinças ópticas usadas para detecção de DNA nanopore [48]. Quando o sistema de pinças ópticas está em equilíbrio, a força óptica (F ot ) é igual à força do campo elétrico (F el ) b Esquema de pinças magnéticas usadas para detecção de DNA nanopore [49]. Quando o sistema de pinças magnéticas está em equilíbrio, a força magnética (F Mt ) é igual à força do campo elétrico (F el ) c Esquema de AFM usado para detecção de DNA nanopore [50]. d Esquema de TFFS usado para detecção de DNA de nanoporos [51]
Pinça magnética
As pinças magnéticas fornecem outra maneira de controlar a translocação do DNA pela força de tensão, e foi demonstrado que a técnica das pinças magnéticas é eficaz em retardar a translocação do DNA [49]. Neste sistema, como mostrado na Fig. 4b, as moléculas de DNA podem ser anexadas a uma esfera magnética do tamanho de um micrômetro usando ouro forte-tiol [54] ou interação estreptavidina-biotina [49]. Então, a extremidade livre do DNA pode ser capturada no nanopore por um campo elétrico aplicado. Posteriormente, dois ímãs com uma pequena lacuna podem ser usados para criar um gradiente de campo magnético. Esta técnica pode equilibrar a força elétrica no DNA preso para reduzir as velocidades de translocação e até mesmo reverter a eletroforese. Em comparação com as pinças ópticas, as pinças magnéticas são um candidato promissor para a espectroscopia de força massivamente paralela. Nesse sistema, centenas de grânulos e, portanto, moléculas de DNA podem ser controlados simultaneamente em centenas de nanoporos, que são facilmente escaláveis para muitos nanoporos endereçáveis. Isso pode acelerar o processo analítico em ordens de magnitude. No entanto, em comparação com as pinças ópticas, uma desvantagem óbvia da abordagem das pinças magnéticas é a falta de controle tridimensional das moléculas [55].
Sonda de detecção de força
Embora um progresso significativo tenha sido feito no controle da velocidade de translocação do DNA por pinças ópticas e magnéticas, os métodos de captura de contas têm um problema com o movimento browniano que torna difícil controlar o movimento da conta com resolução inferior a 10 nm [51] . Para superar isso, o AFM tem sido usado para controlar a velocidade de translocação do DNA [50], e também pode medir simultaneamente a força e a corrente de bloqueio. Em um estudo usando este sistema, como mostrado na Fig. 4c, o DNA foi amarrado à ponta de uma sonda de AFM e, em seguida, foi preso no suporte da sonda. Ao controlar o movimento da sonda, a translocação do DNA poderia ser controlada para reduzir sua velocidade e até mesmo reverter a eletroforese. Além disso, ao retrair a ponta a uma altura acima da superfície correspondente ao comprimento da molécula, as medições podem ser repetidas. Com a ajuda do AFM, a detecção de DNA avançou na prática e na teoria porque a resolução da detecção pode ser muito melhorada pelos sinais combinados de corrente de bloqueio e medições de força de AFM [56]. No entanto, ainda existe um obstáculo na aplicação de técnicas de nanopore na detecção de DNA, a saber, a ocorrência intermitente de flutuações regulares na força (e na corrente) a cada 0,35-0,72 nm quando uma molécula de DNA desliza de maneira relativamente sem atrito através de um nanopore. Essas flutuações são atribuídas a nucleotídeos individuais traduzindo através do nanopore em um movimento semelhante a uma catraca [50].
Estudos demonstraram que um diapasão, que pode ser usado como um sensor de detecção de força, pode controlar o DNA para passar por um nanopore a uma taxa sub-nanométrica [51, 57]. Em um estudo usando este aparelho integrado, como mostrado na Fig. 4d, uma molécula de DNA foi anexada a uma ponta de sonda que foi colada a um pino de um diapasão. Um sistema de nanoposicionamento, que possui uma precisão sub-nanométrica, foi usado para segurar o diapasão [51]. A posição da ponta da sonda pode ser detectada pelo sensor de força de feedback com base em diapasão e controlada pela manipulação do sistema de posicionamento nanométrico. Essa velocidade de movimento é 10 vezes mais lenta do que a do DNA manipulado por pinças ópticas e 1000 vezes mais lenta do que o DNA que passa livremente por nanoporos de estado sólido [57]. Comparado com o AFM convencional, um diapasão pode fornecer um movimento de varredura mais rápido e possuir alta sensibilidade à força quando imerso no líquido. Ao incorporar TFFS com um nanopore, a corrente iônica através de um nanopore, a posição da ponta e a amplitude vibracional da ponta podem ser medidas simultaneamente durante a passagem de uma molécula de DNA através do nanopore.
Sonda Si
Todos os métodos acima, ou seja, pinças magnéticas, pinças ópticas, AFM e TFFS, precisam fazer a varredura da posição nanopore. Eles precisam localizar o nanoporo dentro do comprimento efetivo do DNA para garantir que o DNA possa passar pelo nanoporo. O endereçamento de nanopore é uma parte importante desses métodos, mas é difícil [32] conectar o DNA a uma grande superfície de uma sonda de silício (Fig. 5), maior do que a área do chip, o que significava que eles poderiam inserir facilmente o DNA imobilizado em um nanopore sem procurar a localização do nanopore na membrana. A viabilidade de usar uma sonda de Si imobilizada por DNA e um controlador de posição para controlar o movimento do DNA para dentro e para fora dos nanoporos foi demonstrada. A dificuldade desse método é que a sonda de Si fica imersa na solução e conectada ao DNA pelo acoplamento do peptídeo. A densidade do DNA na superfície da sonda é difícil de controlar, então vários DNAs passando por um nanoporo ao mesmo tempo podem ocorrer, afetando a corrente de detecção.
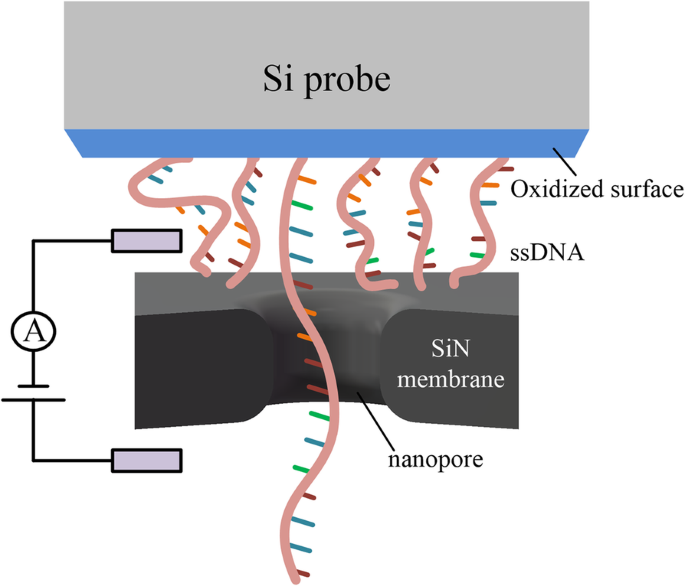
Esquema do sistema de sonda de Si imobilizado por DNA usado para detecção de DNA nanopore [32]
Pinças ópticas, pinças magnéticas, microscopia de força atômica (AFM) e detecção de força baseada em diapasão (TFFS) podem detectar as forças reais e a posição da molécula no nanoporo, que é promissor para controlar a passagem do DNA através dos nanoporos com velocidade apropriada. A dificuldade de endereçar nanoporos é evitada usando uma sonda de Si. Além disso, o uso de um sistema de dois nanoporos é um método viável para controlar e retardar a passagem do DNA através dos nanoporos. Além disso, um nanoporo de armadilha óptica tem potencial para detecção de DNA no futuro. Aqui, resumimos a velocidade de translocação de DNA de um nanopore de estado sólido integrado com alguns métodos de controle de DNA, como um sistema de dois nanopore, nanopore de armadilha óptica, pinças ópticas, pinças magnéticas, AFM, TFFS e sonda de Si (Tabela 1) .
Conclusão
Materiais 2D monocamada, como grafeno, MoS 2 , WS 2 , e h-BN, são possivelmente os materiais mais finos possíveis, pois são tão espessos quanto o espaçamento entre os nucleotídeos. Em comparação com as membranas nanopore de estado sólido tradicionais, as membranas 2D de monocamada são ideais para dispositivos nanopore, pois exibem uma alta relação sinal-ruído de corrente iônica e regiões de detecção relativamente grandes. Eles são potencialmente elegíveis para realizar o sequenciamento de DNA combinando com pinças ópticas, pinças magnéticas, AFM, TFFS, sonda de Si, sistema de dois nanoporos ou nanopore de armadilha óptica. Porém, com essas técnicas, vários desafios surgiram, os quais precisam ser resolvidos antes da comercialização do sequenciamento de DNA de nanoporos. A primeira delas ocorre quando os grânulos ou pontas de sonda estão perto de um nanoporo, quando é mais difícil discriminar os nucleotídeos do DNA com os sinais de corrente iônica. Alças moleculares ou outras moléculas mais longas devem ser usadas para adicionar o comprimento de uma fita de DNA que pode compensar o efeito no sinal de corrente provocado por grânulos ou pontas. Em segundo lugar, os arranjos de nanopore devem ser usados para realizar alta taxa de transferência e detecção paralela, mas a tecnologia de detecção paralela atualmente não está madura o suficiente. Terceiro, de acordo com os métodos de fabricação atuais, é difícil fabricar um sistema de dois nanoporos e um sistema de captura óptica de nanoporos com alta precisão e reprodutibilidade, o que é muito significativo para a detecção de DNA de nanoporos. Um feixe de íons de hélio pode ser a tecnologia chave para resolver este problema [11, 58]. Assim, esperamos que o sequenciamento de nanoporos de DNA continue a ser um foco de pesquisa e possa ser integrado a mais novas ideias e abordagens inovadoras para obter baixas taxas de erro, gravação paralela rápida e alta e comprimentos de leitura longos de até 100 quilobases.
Disponibilidade de dados e materiais
Todos os dados gerados ou analisados durante este estudo estão incluídos neste artigo publicado.
Abreviações
- 2D:
-
Bidimensional
- 3D:
-
Materiais tridimensionais
- TMDs:
-
Dichalcogeneto de metal de transição em camadas
- SNR:
-
A relação sinal-ruído
- PL:
-
Fotoluminescência
- SIBA:
-
Retrocesso auto-induzida
- AFM:
-
Força atômica microscópica
- TFFS:
-
Sensor de força baseado em diapasão
Nanomateriais
- Relés de estado sólido
- Síntese de DNA
- Amazon Alexa Controlando um Chromecast
- Melhorar por meio do monitoramento sem fio
- Conduza ouvindo
- Blog:Detecção de genes através de microarray
- Cientistas da IBM medem transferência de calor por meio de átomos individuais
- Protegendo a IoT por meio de engano
- Chapeamento de furos
- Sensor de dióxido de carbono de estado sólido



