Reaproveitamento de drogas
Inibição da protease principal SARS-CoV-2
O desenvolvimento de um novo medicamento é um processo caro e demorado. Hoje, enquanto o mundo enfrenta uma pandemia, há uma necessidade urgente de identificar rapidamente os medicamentos que impedem a proliferação do vírus. O reaproveitamento de medicamentos oferece uma alternativa atraente para esse processo demorado, tentando identificar se um medicamento sabidamente seguro em humanos poderia ser usado para tratar novas doenças.
Embora o uso de tais drogas reposicionadas individualmente possa eventualmente não resultar em um benefício clínico significativo, a combinação cuidadosa de drogas direcionadas a várias proteínas cruciais para a replicação e proliferação do vírus pode ser muito eficaz, como foi o caso do HIV na década de 1990. A questão urgente é qual combinação seria mais eficaz?
Aqui, tentamos compreender a estrutura do sítio ativo da protease SARS-CoV-2, comparando-a com as estruturas existentes da protease SARS-CoV complexada com inibidores micromolares, para que possamos compreender melhor as principais interações necessárias para criar um bom inibidor para a protease SARS-CoV-2.
Em seguida, conduzimos um experimento de triagem virtual usando uma biblioteca de medicamentos aprovados pelo FDA para ver se alguns deles se ligam à protease. Examinamos como se prevê que eles se liguem à protease SARS-CoV-2 e, portanto, poderiam ser usados em uma terapia combinada.
Proteínas SARS-CoV – 2
O genoma SARS-CoV – 2 de pacientes doentes foi rapidamente isolado e sequenciado, fornecendo as sequências de possíveis alvos proteicos. Essas proteínas compartilham alta similaridade de sequência com as proteínas SARS-CoV e, inicialmente, grupos de pesquisa começaram a construir modelos de homologia. Agora estamos vendo mais e mais dessas estruturas derivadas experimentalmente (raios-X e Cryo-EM) se tornando disponíveis.
Um dos alvos de drogas mais bem caracterizados entre os coronavírus é a protease principal:Mpro, também chamada de 3CL Protease. 1 Junto com as proteases semelhantes à papaína, esta enzima é essencial para processar as poliproteínas que são traduzidas do RNA viral. 2 Ele cliva a estrutura de aminoácidos em 11 locais de clivagem na grande poliproteína 1ab (Fig. 1).
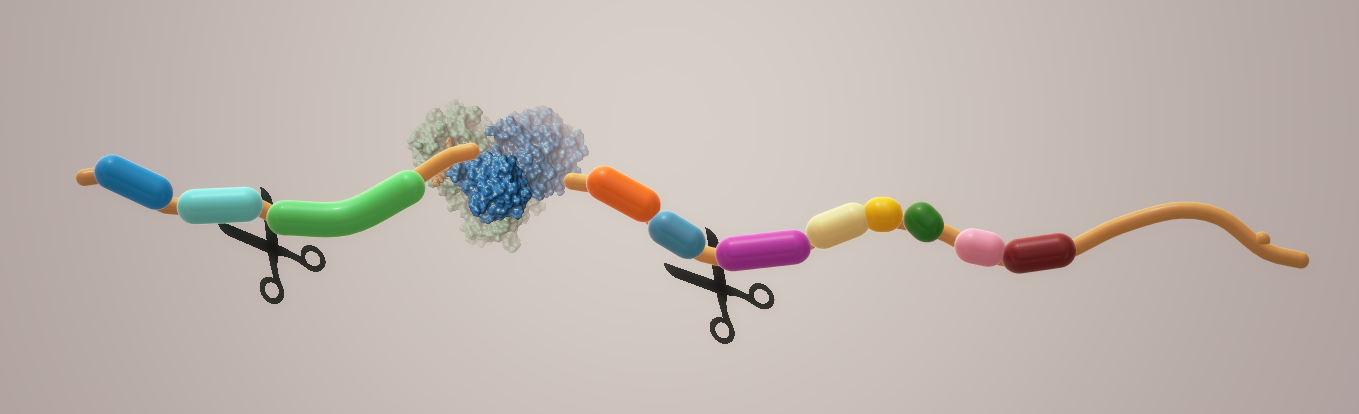
A inibição da atividade dessa enzima bloquearia a replicação viral. Uma vez que nenhuma protease humana com especificidade de clivagem semelhante é conhecida, os inibidores para este alvo são muito menos propensos a serem tóxicos e causar efeitos colaterais.
Projeto de medicamento baseado em estrutura para reaproveitamento de medicamentos:protease principal
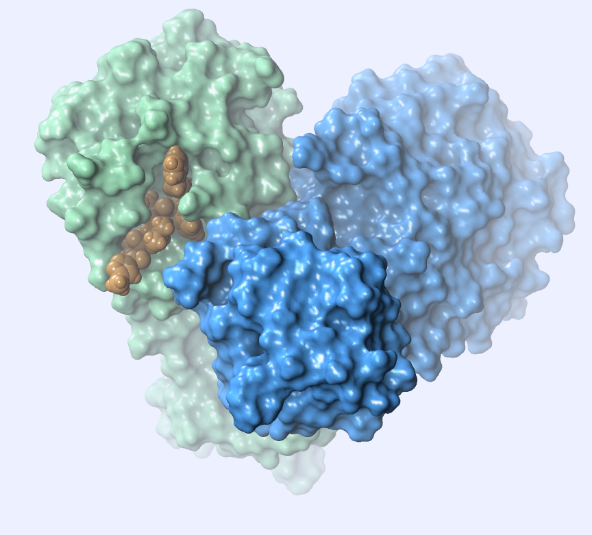
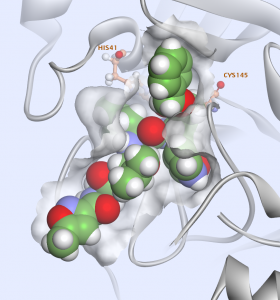
Esta protease principal é um homodímero (Fig. 2) e cada subunidade contém uma díade catalítica His41 / Cys145.
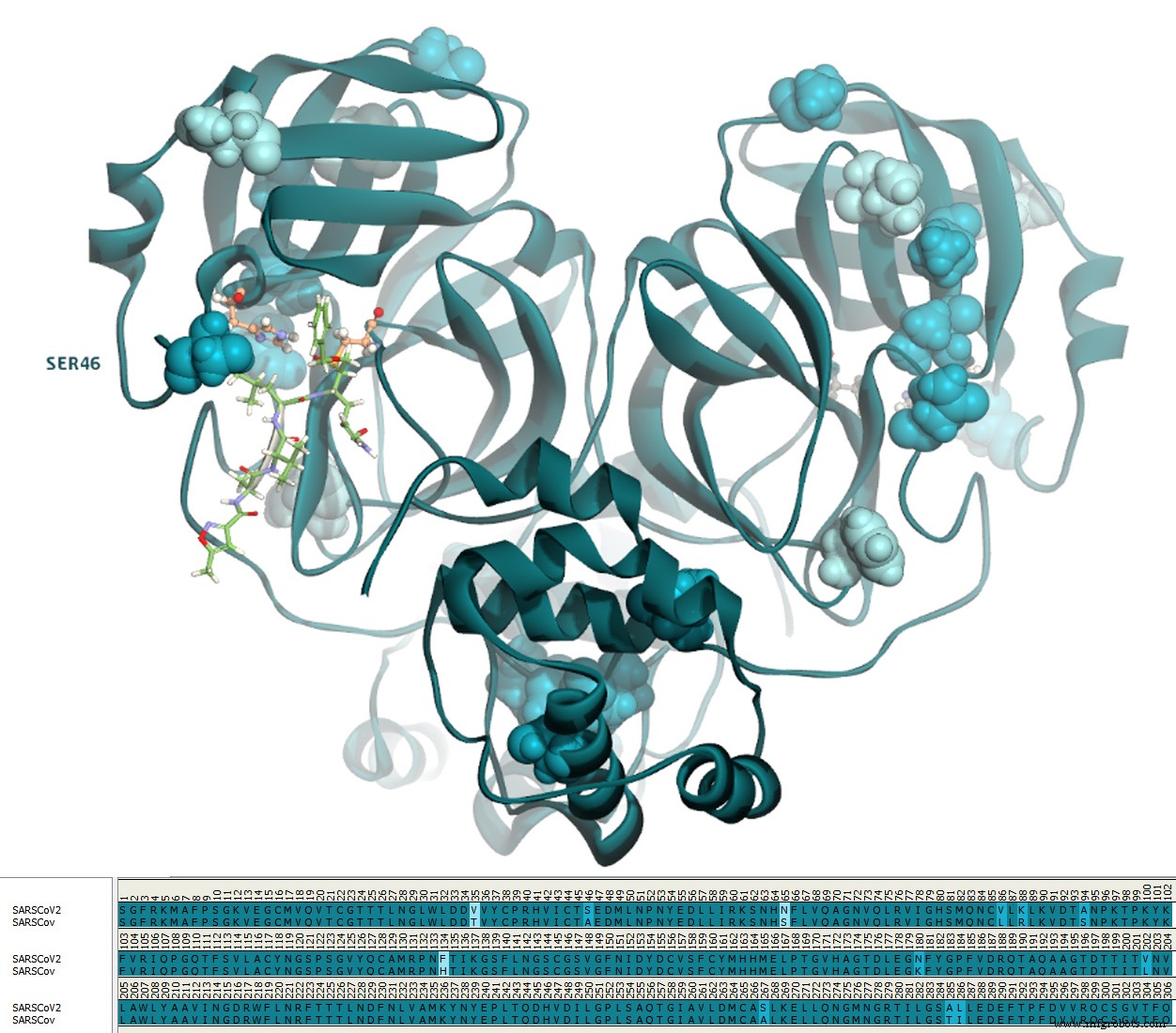
O SARS-CoV-2 compartilha 96,1% de identidade e 99% de similaridade com a protease SARS-CoV. (Fig. 3) Há apenas uma diferença de aminoácidos no sítio ativo:o resíduo 46 no SARS-CoV-2 é uma serina em vez de uma alanina no SARS-CoV.
Existem muitas estruturas de SARS-CoV Mpro complexadas com ligantes disponíveis no PDB e os ligantes que têm uma afinidade de ligação micro molar são todos ligantes covalentes.
Os sítios ativos da protease principal são altamente conservados entre os coronavírus e geralmente são compostos por quatro sítios (S1 ′, S1, S2 e S4) 3 (Fig. 4).
No caso de estruturas PDB 2ZU4 e 2GX4 para as quais os ligantes têm valores Ki respectivamente de 0,038 μM e 0,053 μM, o tiol de cisteína 145 no local S1 ′ contata inibidores com uma ligação covalente. Quando comparado com outros inibidores com menor afinidade, isso parece ser importante para maior afinidade.
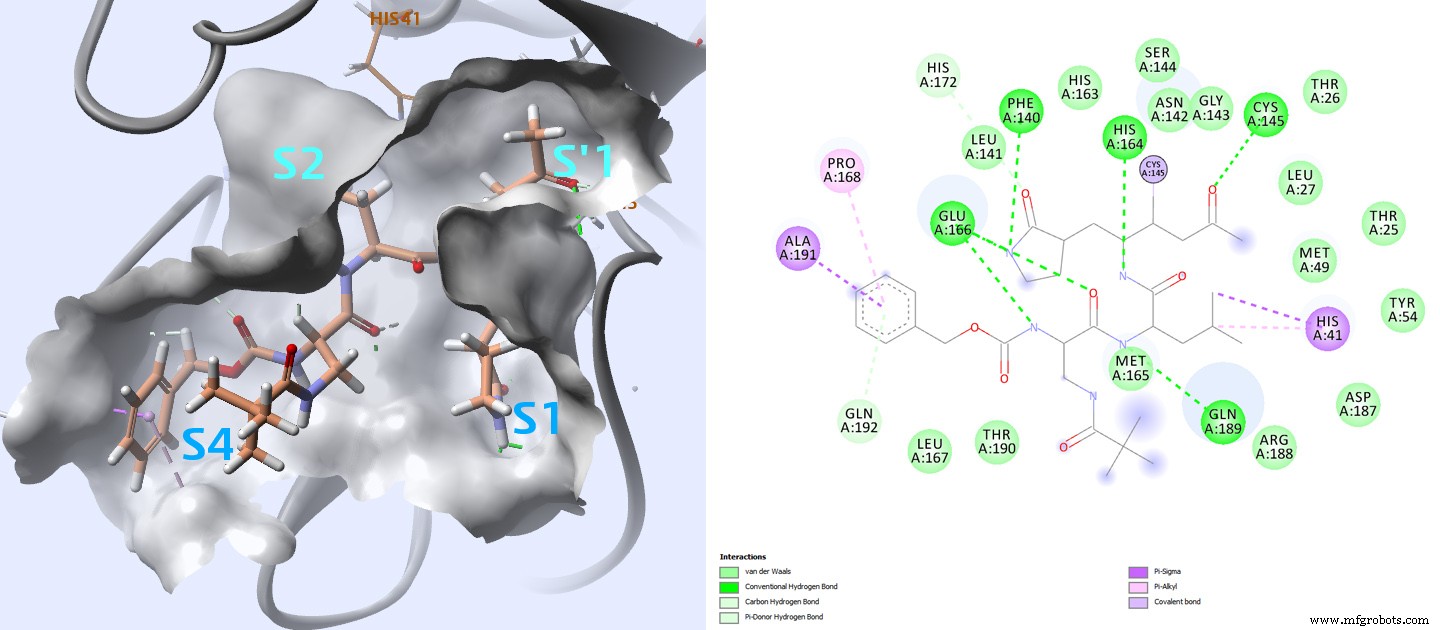
O exame cuidadoso de como todos esses ligantes interagem com esta protease pode fornecer informações sobre as principais interações a serem monitoradas ao analisar os resultados de docking.
Triagem Virtual
Tirando vantagem de uma estrutura de cristal de alta resolução do dímero de protease SARS-COV-2 em complexo com um inibidor semelhante a um peptídeo ligado covalentemente N3 lançado em fevereiro (6LU7) 4 , usamos essa estrutura para conduzir um experimento de triagem virtual. O inibidor N3 que é cocristalizado se liga na bolsa de ligação ao substrato em uma conformação estendida. (Fig. 5)
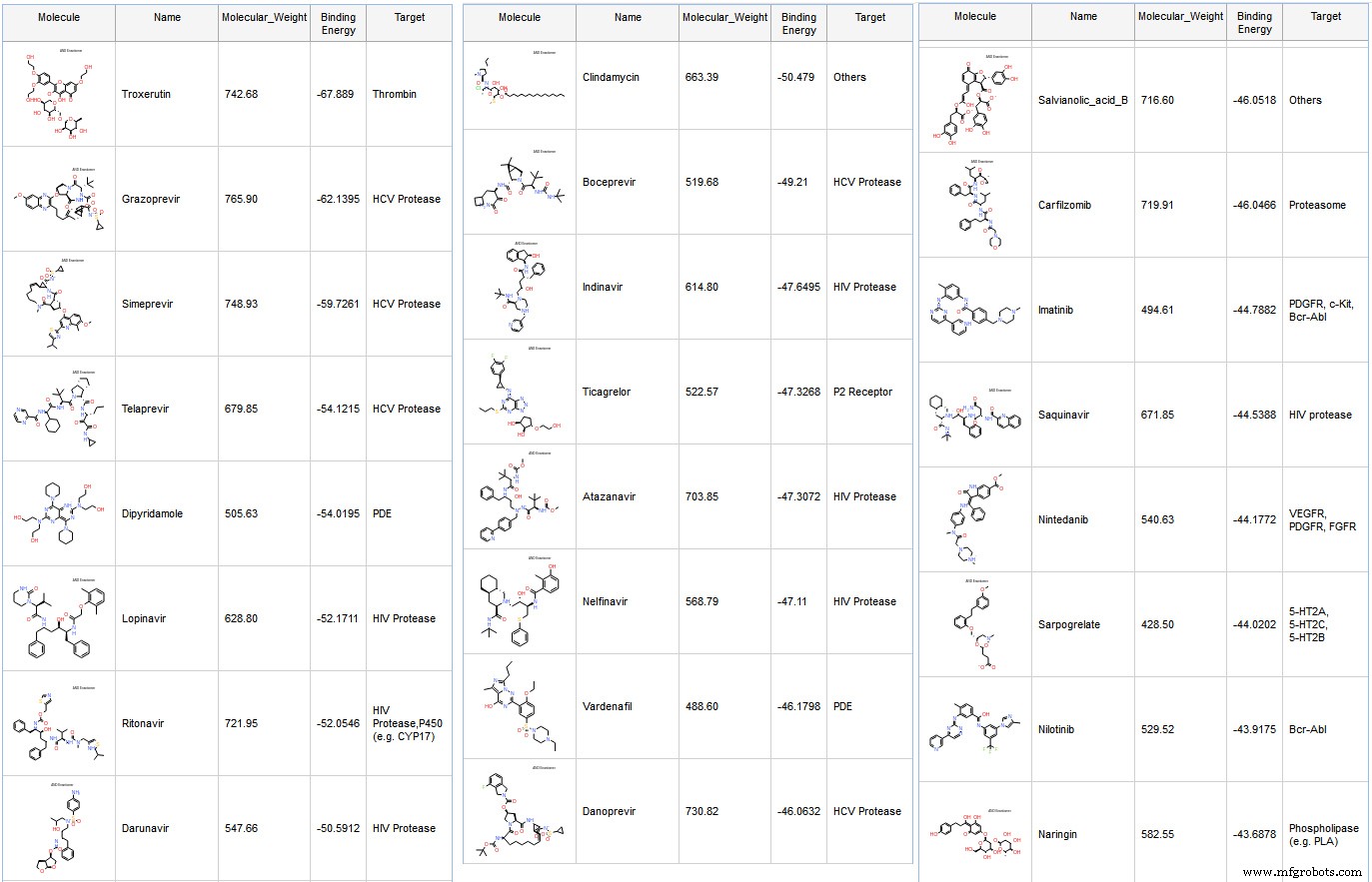
Começamos com uma biblioteca contendo medicamentos aprovados pela FDA com 2.684 compostos. Retivemos compostos com Peso Molecular inferior a 800 kDa e realizamos docking com GOLD do CCDC. Posteriormente, calculamos as Energias Livres de Ligação com o solvente implícito CHARMM e GBMV. Para cada postura, primeiro executamos a minimização do ligante in situ com uma esfera de 14 Å de diâmetro para a flexibilidade do resíduo e estimamos a entropia do ligante ao calcular a energia livre de ligação. A maioria dos compostos acoplados são drogas conhecidas de protease de HCV e HIV. (Fig. 6)
Muitos compostos fazem contatos nos 4 subsites:S1, S’1, S2, S4 e alguns com HIS 41 e CYS 145, especialmente os inibidores de protease de HIV.
O composto com melhor pontuação é a Troxerutina, que é um flavonóide.
- Foi demonstrado que os flavonóides inibem algumas proteases 5 e recentemente IC 50 os valores foram calculados a partir das curvas inibitórias dependentes da dose de herbacetina, rhoifolina e pectolinarina em SARS-CoV. Os valores medidos foram 33,17, 27,45 e 37,78 μM, respectivamente.
O dipiridamol (Fig. 7), que é o quinto composto classificado, foi mencionado recentemente em uma pré-impressão. Efeitos terapêuticos do dipiridamol em pacientes COVID-19 com disfunção de coagulação e mostrou suprimir a replicação do HCoV-19 a uma EC50 de 100 nM in vitro.
Figura 7 :Renderização 3D e mapa de interação 2D do dipiridamol na protease principal. Interações com resíduos catalíticos estão presentes, e com alguns dos resíduos mencionados anteriormente para inibidores de SARS-CoV.
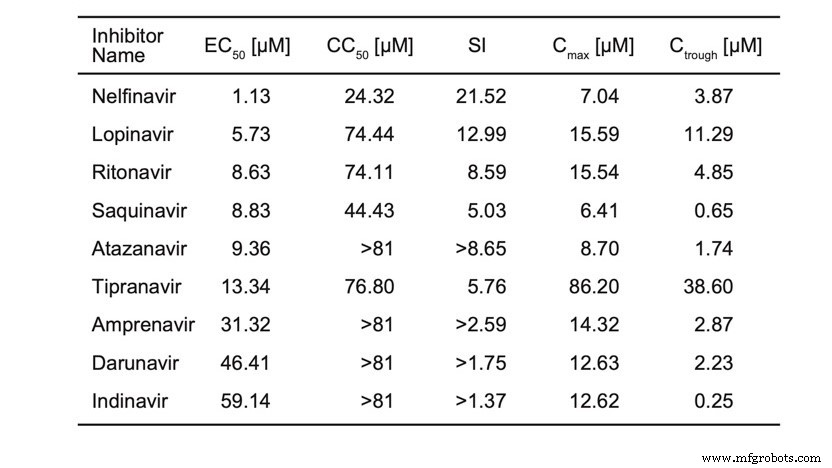
Uma pré-impressão recente relatou ensaios celulares de vários inibidores da protease do HIV:“Nelfinavir inibe a replicação do coronavírus 2 da síndrome respiratória aguda grave in vitro . ”
Estamos relatando a tabela da pré-impressão (Tabela 1). Estes não são IC50, mas EC50, portanto, não diz o quão bem essas moléculas se ligam à protease. No entanto, isso mostra que todos esses compostos estão inibindo a replicação do SARS-CoV-2 e alguns são mais ativos do que outros.
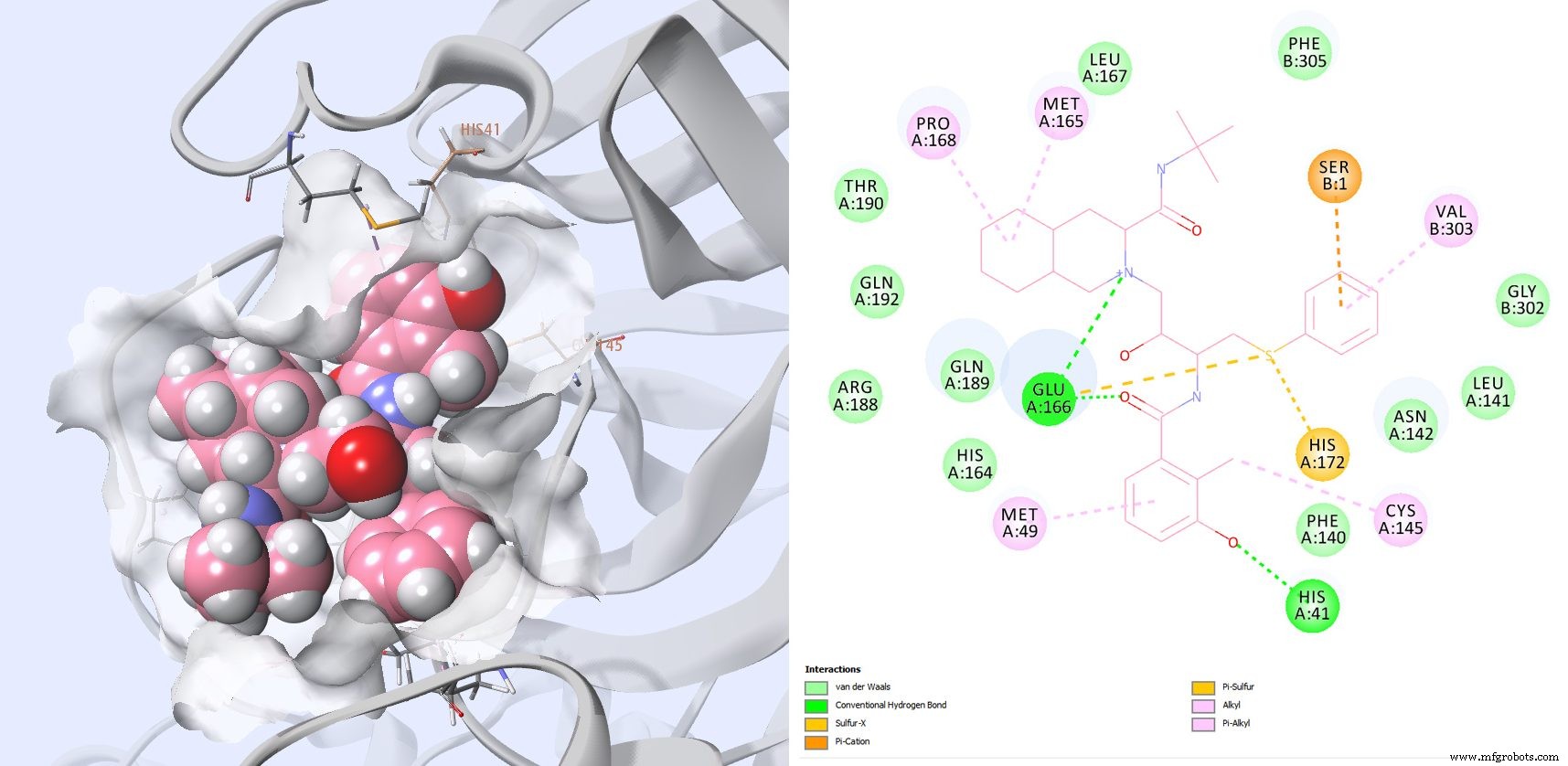
O ritonavir e o lopinavir estão interagindo com muitos resíduos previamente observados em outros compostos que têm como alvo a protease SARS-CoV (Fig. 8 e 9). Embora vários ensaios clínicos estejam em andamento com uma combinação dessas duas moléculas, infelizmente não há dados de IC50 disponíveis para Ritonavir ou Lopinavir em SARS-CoV-2.
Figura 8 :Melhor pontuação para o Ritonavir:e diagrama de interação 2D mostrando que o Ritonavir está interagindo com os dois resíduos catalíticos:CYS145 e HIS41, mas também GLU166, PRO168 e GLN189 conforme observado com outros inibidores conhecidos da SARS-CoV.
Figura 9 :Melhor pontuação para Lopinavir:e diagrama de interação 2D mostrando que Lopinavir está interagindo com os dois resíduos catalíticos:CYS145 e HIS41, mas também GLU166, PRO168 e GLN189 como visto com outros inibidores conhecidos de SARS-Co V.
O nelfinavir (Fig. 10) tem uma alta afinidade de ligação prevista em nossos cálculos e também foi caracterizado contra COVID19 em um preprint recente:“O nelfinavir é ativo contra SARS-CoV-2 em células Vero E6”.
Uma segunda pré-impressão mostra que o Atazanavir é ativo nas células infectadas com SARS-CoV-2:“O atazanavir inibe a replicação do SARS-CoV-2 e a produção de citocinas pró-inflamatórias”.
Uma estrutura PDB 6W63 que contém um novo inibidor para SARS-CoV-2 foi publicada após iniciarmos este trabalho (Fig. 11). Não há informações publicadas sobre a afinidade de ligação deste composto ao SARS-CoV-2 Mpro. As cadeias laterais no local de ligação estão quase na mesma orientação que para 6LU7. Encaixamos nossos compostos e os recriamos com abordagens MMGBSA como anteriormente e encontramos resultados muito semelhantes.
Figura 11 :Estrutura do PDB 6W63:Protease principal do SARS-CoV-2 em complexo com o inibidor X77.
Perspectivas
O sítio de ligação da protease SARS-CoV-2 é muito grande, contendo quatro sub-sítios, e pode acomodar muitos ligantes diferentes com uma afinidade de ligação moderada. Conforme explicado aqui, a triagem virtual é uma ferramenta útil para identificar possíveis inibidores e foi capaz de nos dar alguma explicação estrutural sobre como os possíveis inibidores podem estar interagindo com a protease.
No entanto, todas essas hipóteses requerem confirmação por evidências experimentais, como medição de IC50, para construir modelos mais robustos.
Aqui não realizamos docking covalente, mas como os compostos mais ativos contra a protease SARS-CoV eram covalentes, isso pode ser um requerimento de inibidores fortes. Os inibidores covalentes também apresentam vantagens em comparação com inibidores reversíveis, como ter forte afinidade ao alvo e vida de ação prolongada em pacientes.
Para que um medicamento seja ativo em um paciente, precisamos considerar outros aspectos farmacológicos, como a farmacocinética. Será necessário avaliar a capacidade dessas drogas para atingir as concentrações plasmáticas e pulmonares alvo após a dosagem aprovada em humanos.
Não estava no escopo deste trabalho comentar sobre outras pesquisas de design baseado em estrutura, uma vez que isso não seria oportuno para a epidemia atual. Nosso objetivo foi avaliar a terapêutica prontamente utilizável para drogas candidatas em potencial contra COVID19.
Gostaríamos de agradecer ao Cambridge Crystallographic Data Center (CCDC) pela permissão para usar o GOLD para a exibição virtual neste trabalho. O GOLD tem sucesso comprovado em triagem virtual, otimização de leads e identificação do modo de ligação correto de moléculas ativas. Uma interface para GOLD está disponível no Discovery Studio.
TREINAMENTO DO PILOTO DO PIPELINE ONLINE
Modelagem e simulação propõe novos insights para SARS-CoV-2
biológico
- China estabelecerá vínculo de patente
- A cadeia de suprimentos de drogas dos EUA em crise:Soluções para a escassez
- COVID-19 revelou falhas fatais na cadeia de suprimentos de medicamentos dos EUA
- Como a Blockchain pode ajudar a combater drogas falsificadas
- Aplicando Blockchain e Aprendizado de Máquina para Redes de Suprimento de Drogas
- Teste completo para monitoramento de COVID-19
- Novo efeito fotônico pode acelerar o desenvolvimento de medicamentos
- Smartwatch rastreia os níveis de medicação
- 5 Ws do sensor SARS-CoV-2 RapidPlex
- Micro-robôs impressos em 3D prometem entrega de medicamentos



