Decodificando os Genomas SARS-CoV-2 - Origem
A atual pandemia de COVID-19 é uma ameaça global aos sistemas de saúde pública e atingiu duramente a economia mundial. O culpado desta pandemia, SARS-CoV-2, não é a gripe típica. Este vírus afeta as vias respiratórias superiores e inferiores, interferindo no processo básico da vida, a respiração e, portanto, é mortal. Em 6 de abril de 2020, o Worldometer relatou 1.337.166 casos com 74.176 mortes em todo o mundo .
O exame do SARS-CoV-2 no nível do genoma fornecerá informações sobre a compreensão das origens desse vírus. Também ajudará os cientistas a projetar ferramentas de diagnóstico para detectar esse patógeno invisível e facilitar a invenção de terapêuticas para minimizar a perda de vidas.
Compreendendo o Genoma SARS-CoV-2
Um vírus é um agente infeccioso que requer um hospedeiro vivo para se desenvolver e se replicar. Além disso, o SARS-COV-2 é um vírus de RNA de fita simples com um genoma de bases de nucleotídeos de quase 30 kb com 12 quadros de leitura abertos putativos. Logo após o início da epidemia em dezembro de 2019, cientistas chineses sequenciaram o genoma SARS-CoV-2. Vários grupos científicos lançaram sequências genômicas completas do SARS-CoV-2 nas últimas semanas. Estes estão publicamente disponíveis no Genbank e no Coronavirus Database .
Origem do vírus SARS-CoV-2
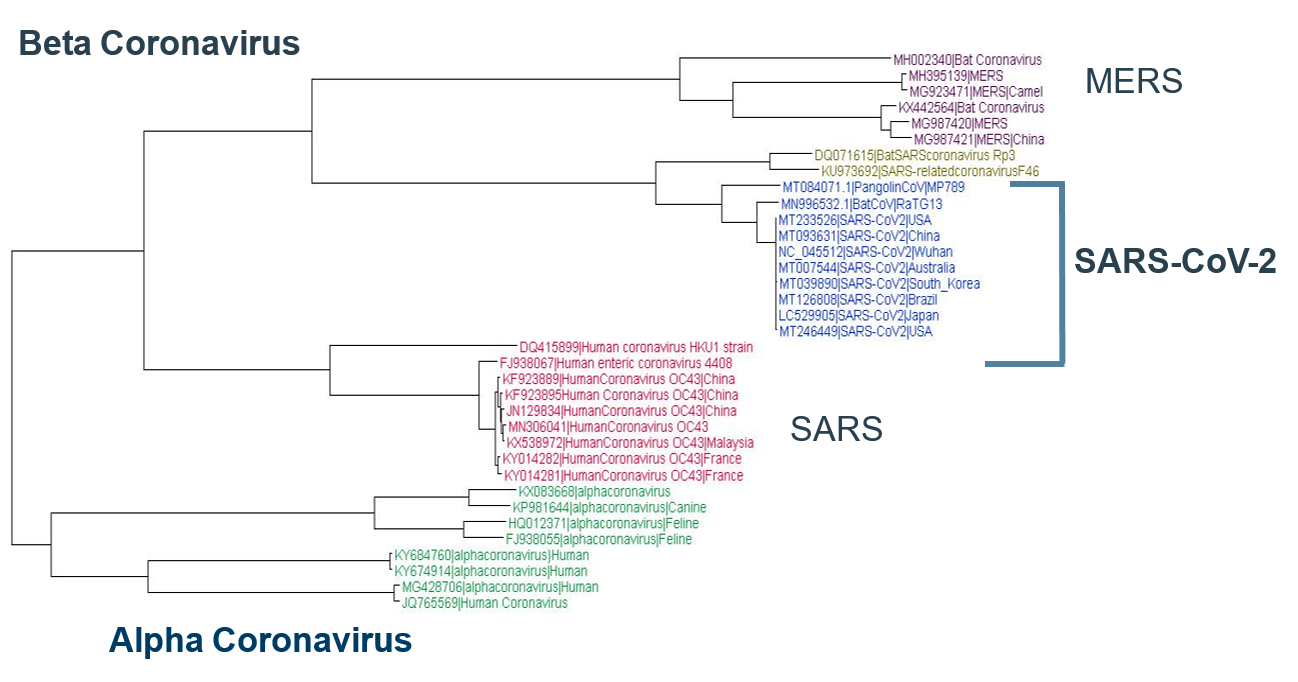
Durante surtos como este, teorias de conspiração não científicas podem resultar em preconceitos desnecessários contra países, comunidades e culturas. O SARS-CoV-2 não é exceção, e a situação só é agravada pelas plataformas de mídia social em expansão de hoje. É nossa responsabilidade ver esse inimigo invisível por meio de lentes científicas racionais. Com base nas análises do genoma, o SARS-CoV-2 é um vírus que evoluiu naturalmente e não é uma cepa sintética de laboratório 1,2 . Os cientistas sequenciaram os genomas completos de mais de 100 cepas de SARS-CoV-2 coletadas em diferentes regiões do mundo. Acontece que essas cepas são mais de 99,5% idênticas em um nível de nucleotídeo. Isso indica que as cepas não sofreram grandes mutações em diferentes regiões, aparentemente porque o vírus já tem uma alta taxa de infecção e virulência.
No passado recente, dois outros coronavírus receberam atenção global. Esses foram o SARS-CoV, China, 2002, e o MERS-CoV, Arábia Saudita, 2012. Ambos os vírus anteriores mostraram ter se originado em morcegos. Com base nesse conhecimento histórico, os cientistas sequenciaram o coronavírus dos morcegos e mostraram que Bat CoV ( RaTG13 ) foi 96,2% idêntico ao SARS-COV-2, confirmando assim a origem zoonótica do último. 2 O coronavírus costuma usar um portador intermediário antes de infestar humanos. Curiosamente, por volta de outubro de 2019, relatos de pangolins malaios mortos com pulmões e sintomas de fibrose pulmonar espumosa no centro de resgate de animais selvagens de Guangdong, na China, levaram os cientistas a isolar seu metagenoma. Na verdade, os dados do metagenoma dos pangolins mortos continham o coronavírus! 3
Curiosamente, em todo o nível do genoma, o SARS-CoV-2 é quase 91% idêntico ao Pangolin CoV malaio, indicando que os pangolins podem ser um hospedeiro intermediário.
O que são Pangolins? Eles são mamíferos comedores de formigas muito procurados na Ásia para uso na medicina tradicional chinesa, bem como para sua carne, que muitos consideram uma iguaria. Eles também são os mamíferos mais traficados atualmente no comércio ilegal de vida selvagem.
O SARS-CoV-2 é diferente de outros coronavírus conhecidos, com 88% ou menos de identidade de sequência. Com base em análises filogenéticas, o SARS-CoV-2 observado em humanos, morcegos (RaTG13) e pangolins malaios é uma nova classe de beta coronavírus. Quase 35 tipos diferentes de cepas de coronavírus de diferentes partes do mundo e de diferentes organismos foram analisados em todo o genoma. SARS-CoV-2, mostrado em azul abaixo, é uma nova classe de beta coronavírus (Figura 1).
Como o Coronavírus entra no hospedeiro?
Uma das proteínas do coronavírus, chamada proteína Spike, parece desempenhar um papel importante nesse processo. A proteína Spike é uma máquina molecular multifuncional que consiste em duas subunidades principais, S1 e S2. A proteína Spike primeiro se liga a um receptor na superfície da célula hospedeira por meio de sua subunidade S1 e, em seguida, funde as membranas virais e do hospedeiro por meio de sua subunidade S2. O domínio em S1 de diferentes coronavírus reconhece uma variedade de receptores do hospedeiro, levando à ligação viral. O domínio de ligação ao receptor (RBD), que tem 193 aminoácidos, liga-se e liga-se à célula hospedeira. O receptor do SARS-CoV-2 em humanos é a Enzima Conversora da Angiotensina 2 (ACE2). ACE2 está ligado à superfície externa das membranas celulares nos pulmões, artérias, coração, rins e intestinos. O ACE2 reduz a pressão sanguínea ao catalisar a clivagem da angiotensina II, um peptídeo vasoconstritor, em angiotensina 1-7, um vasodilatador. Infelizmente, o ACE2 também parece ser um ponto de entrada popular para coronavírus.
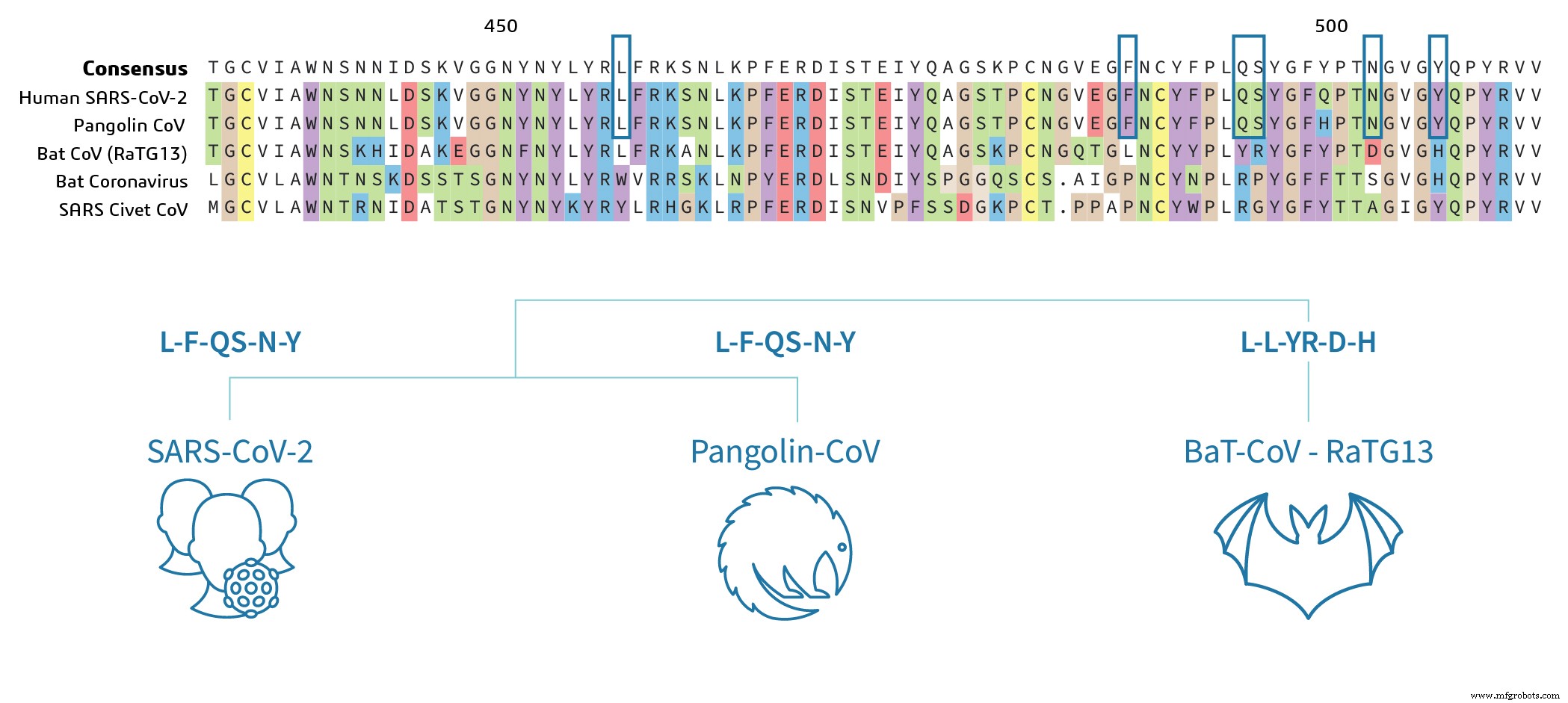
As sequências Pangolin CoV e SARS-CoV-2 são altamente conservadas na região RBD, indicando que o potencial patogênico do vírus é muito semelhante entre Pangolin CoV e SARS-CoV-2. Os principais resíduos de aminoácidos, que determinam a ligação, são idênticos entre Pangolin CoV e SARS-CoV-2 no alinhamento de sequência (marcados com caixas azuis na Figura 2a) e os aminoácidos principais (LFQSNY) mostrados acima dos desenhos na Figura 2b . Curiosamente, o morcego SARS-CoV-2 RBD difere em 17 resíduos de aminoácidos, que incluem cinco resíduos críticos para a ligação 3 . Com base na análise dos dados da sequência, pode-se especular que o morcego-SARS-CoV-2 pode não ter os resíduos-chave para se ligar à proteína ACE2 da célula hospedeira para desencadear a infecção. Isso exigirá experimentação para confirmar.
Como observado anteriormente, a proteína Spike contém dois domínios funcionais:um domínio de ligação ao receptor e um segundo domínio que contém sequências que medeiam a fusão das membranas viral e celular. A glicoproteína de Spike deve ser clivada por proteases celulares para permitir a exposição das sequências de fusão e, portanto, é necessária para a entrada na célula. A comparação da sequência do local de clivagem S1 / S2 de Pangolin CoV e morcego-SARS-CoV-2 mostra uma inserção do motivo de reconhecimento de furina. Isso indica um mecanismo distinto para a entrada do genoma viral no citoplasma do hospedeiro para replicação, conforme mostrado na Figura 3.
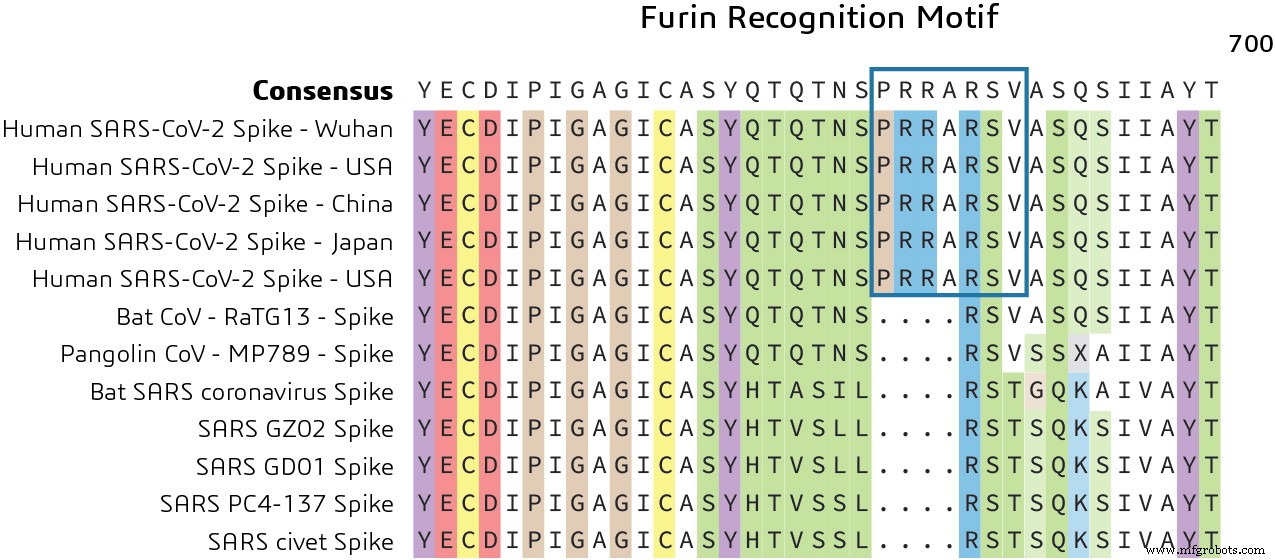
Qual é o papel do motivo de reconhecimento de furin? Em humanos, o motivo de reconhecimento da furina (PRRARSV) é reconhecido pela proteína FURIN, um membro da família S8 de peptidases semelhantes à subtilisina que ajuda a remover seções da proteína para alterar sua conformação de um estado inativo para um ativo.
Foi sugerido que a aquisição deste local de clivagem de furin pode ser um "ganho de função" que permitiu a um morcego CoV saltar para os humanos e iniciar sua atual disseminação epidêmica. Esta pode ser uma via potencial para explorar novos medicamentos que visam o bloqueio deste motivo para evitar a replicação do vírus dentro do hospedeiro.
Assim, um exame cuidadoso da proteína Spike no SARS-CoV-2 mostra o RBD otimizado, um motivo de reconhecimento de furina, como alguns coronavírus MERS, e sua capacidade de se ligar fortemente à proteína ACE2. Isso sugere um processo de seleção natural em jogo. Demonstrou-se que os eventos de recombinação natural em vírus que co-infectam um hospedeiro melhoram sua gama de hospedeiros, ao mesmo tempo que aumentam a virulência e a adaptação do vírus. Os dados do genoma SARS-CoV-2 com espinha dorsal do morcego (RaTG13) e pangolim CoV indicam novamente que este é um vírus gerado por recombinação natural .
Qual é o doador imediato de SARS-CoV-2 para humanos?
A sequência SARS-CoV-2 possui uma mistura de morcegos SARS-CoV (RaTG13) e também regiões de Pangolin CoV conservadas que só podem ocorrer durante a recombinação desses genomas virais. Além disso, um ganho de função, como visto com o motivo de reconhecimento de furina, envolve outra recombinação de vírus. Para que a recombinação ocorra, é lógico que haja um hospedeiro natural que abrigue esses genomas virais. É outro pangolim? Ou outro animal selvagem no mercado de frutos do mar de Wuhan? Isso ainda é desconhecido. Compreender a origem pode ajudar a prevenir futuros surtos de cepas virais e pandemias globais.
Para obter mais informações, entre em contato conosco:https://www.3dsbiovia.com/about/contact/.
biológico
- O 555 IC
- A Fórmula Quadrática
- Decodificando ‘Indústria 4.0’
- Uma abordagem da cadeia de suprimentos para resolver o desafio do Coronavírus
- A epidemia de Coronavirus servirá como um alerta para cadeias de suprimentos globais?
- O Coronavirus está destruindo as cadeias de suprimentos tradicionais
- Seis Estratégias de Cadeia de Suprimentos para Petróleo e Gás na Era do Coronavírus
- O Coronavirus pode estimular o fim de dados inválidos de envio
- Como a ciência de dados ajudou a combater o surto do Coronavírus
- 5 Ws do sensor SARS-CoV-2 RapidPlex



