Identificação de macromoléculas características de genótipos de Escherichia coli por microscópio de força atômica Mapeamento mecânico em nanoescala
Resumo
A categorização das cepas microbianas é convencionalmente baseada no método molecular e raramente são as características morfológicas nas cepas bacterianas estudadas. Nesta pesquisa, revelamos as estruturas macromoleculares da superfície bacteriana via mapeamento mecânico de AFM, cuja resolução foi determinada não apenas pelo tamanho da ponta em nanoescala, mas também pelas propriedades mecânicas do espécime. Esta técnica possibilitou o estudo em nanoescala de estruturas membranosas de cepas microbianas com preparação de espécimes simples e ambientes de trabalho flexíveis, que superaram as múltiplas restrições da microscopia eletrônica e possibilitaram a marcação de métodos analíticos bioquímicos. As macromoléculas características localizadas entre a superfície celular foram consideradas proteínas da camada superficial e foram consideradas específicas da Escherichia coli genótipos, a partir dos quais os tamanhos moleculares médios foram caracterizados com diâmetros variando de 38 a 66 nm, e as formas moleculares eram semelhantes a rins ou redondas. Em conclusão, as estruturas macromoleculares de superfície têm características únicas que se ligam à E. coli genótipo, o que sugere que os efeitos genômicos nas morfologias celulares podem ser rapidamente identificados usando mapeamento mecânico AFM.
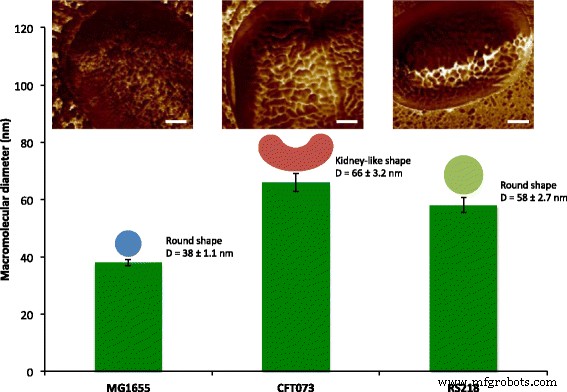
Quantificação de macromoléculas de superfície de E. coli células usando mapeamento mecânico AFM. Macromoléculas de superfície da superfície celular de três E. coli genótipos, MG1655, CFT073 e RS218, foram caracterizados com os tamanhos variando de 38 a 66 nm e com formas redondas ou semelhantes a rins. As imagens da topografia foram coloridas com mapeamento de adesão com as barras de escala =200 nm.
Histórico
Devido à resolução inferior da microscopia óptica e ao ambiente de trabalho restrito da microscopia eletrônica, os pesquisadores no campo microbiano raramente consideram usar a aparência de células bacterianas, mas em vez disso, adotam métodos analíticos moleculares ou químicos para a identificação de fragmentos genômicos, expressão de proteínas, etc. Não é de surpreender que esses métodos tenham uma série de desvantagens, incluindo trabalho intensivo e demorado e, portanto, abordagens mais simples, eficientes e flexíveis são necessárias. Inventado em 1982 por Binnig et al., O microscópio de força atômica (AFM) é projetado para usar uma sonda em nanoescala monitorada por um feixe de laser para a observação da superfície de um espécime com resolução nanoscópica ou mesmo atômica [1]. Imaginando a morfologia por meio de uma ponta de sonda física, esta técnica supera o limite de resolução e as restrições ambientais da microscopia óptica e eletrônica e tem uma série de vantagens, como preparação simples da amostra e ambientes de trabalho flexíveis no ar ambiente ou condições de fluido [2, 3]. Ainda assim, no que diz respeito aos estudos microbianos, as aplicações atuais de AFM envolvem principalmente a imagem qualitativa da morfologia estática ou dinâmica de células bacterianas ou a expressão de flagelos e pili [4,5,6], enquanto poucos estudos têm enfocado a ultraestrutura de superfície de células microbianas e a análise quantitativa das propriedades celulares.
Nesta pesquisa, AFM foi selecionado para o estudo de superfície de Escherichia coli ( E. coli ) células, e as formas e dimensões da bactéria individual foram observadas pela topografia AFM e imagens de fase. Além disso, o mapeamento mecânico simultâneo revelou informações biomecânicas adicionais sobre os componentes da superfície, onde pequenas diferenças nas características adesivas entre as macromoléculas e a matriz circundante puderam ser detectadas durante cada contato físico entre a ponta e a amostra. Aplicando essas técnicas avançadas aos campos microbianos, examinamos três E. coli genótipos que continham uma cepa de laboratório e duas cepas patogênicas humanas para a identificação de macromoléculas de superfície. Os resultados mostraram que a técnica pode oferecer uma resolução de imagem superior à escala da ponta do AFM ao detectar a distribuição mecânica do corpo de prova. Em conclusão, sugerimos que tal desenvolvimento na ciência de superfície não só forneceria aos pesquisadores que trabalham em campos microbianos detalhes da aparência celular, mas também contribuiria para o nosso conhecimento das características da superfície de outros sistemas bio ou nanomateriais.
Métodos
Amostras microbianas
Os três E. coli cepas testadas nesta pesquisa foram clinicamente isoladas e fornecidas pelo laboratório do Prof. Ching-Hao Teng no Instituto de Medicina Molecular da Universidade Nacional de Cheng Kung. MG1655 é a cepa de laboratório de tipo selvagem e intestinal de E. coli K-12 e as duas outras cepas são patógenos humanos - CFT073, a principal causa de infecções do trato urinário, e RS218, associada à meningite neonatal entre bebês [7].
Substrato funcionalizado
Duas etapas de modificações de superfície foram aplicadas para a ligação covalente entre a superfície sólida e as células microbianas. Primeiro, a solução 3-aminopropiltrietoxissilano (APTES, Sigma-Aldrich Co. LLC, EUA) foi usada para formar um NH 2 inicial - camada funcionalizada na superfície, onde o revestimento APTES estável contribuiu para as ligações de Si-O estáveis - Si fornecido por APTES e O da superfície oxidada. Possuindo duas funções COOH, o glutaraldeído se combina ao NH 2 função do APTES com um COOH e funciona com o NH 2 na superfície bacteriana com o outro COOH.
Os substratos limpos foram imersos em solução de APTES, com uma mistura de 5% de APTES em etanol, por uma hora e enxaguados com etanol e DDH 2 O. As lâminas foram secas com corrente de nitrogênio e, em seguida, colocadas em solução de glutaraldeído, 2% em PBS, durante a noite, e lavadas com PBS.
Preparação da amostra
Colônias únicas de E. coli as cepas foram selecionadas das placas de ágar com caldo de lisogenia (LB) e incubadas em caldo LB Após os tempos de cultivo de 12 h, a solução bacteriana foi então diluída 1:100 em caldo LB pré-equilibrado fresco. Após mais 12 h para cultivo microbiano, a solução bacteriana foi submetida a centrifugação a 1500 × g (4000 rpm) por 3 min e ressuspenso em caldo LB, com esse processo repetido duas vezes. Duzentos microlitros da solução bacteriana foram colocados no substrato funcionalizado e deixados em repouso por 30 min. A amostra foi então submersa em água destilada duas vezes para remover as células não anexadas e imediatamente fotografada sob AFM no ar ambiente.
Caracterização AFM
Um instrumento AFM (Bruker Nano, Santa Bárbara, CA, EUA) e uma sonda de nitreto de silício com a constante de mola calibrada de 0,7 N / me raio de ponta de 10 nm foram selecionados para o exame de superfície de espécimes microbianos. A taxa de varredura AFM e os pixels de linha foram de 0,5 Hz e 256 linhas, respectivamente, para o tamanho de varredura de 10 μm para a primeira detecção de topografia, e os parâmetros foram então definidos como 0,3 Hz e 512 linhas para o tamanho de varredura de 2 μm para a observação detalhada. O modo quantitativo nanomecânico PeakForce (QNM) foi utilizado para o mapeamento nanomecânico, onde as propriedades adesivas da superfície foram calculadas a partir da força de atração máxima entre as curvas força-distância de retirada.
Nosso estudo anterior sobre Streptococcus mutans mostraram a evolução mecânica na superfície bacteriana por 2 h, monitoradas por mapeamento mecânico contínuo de AFM, e as amostras microbianas foram verificadas permanecer vivas durante esse período [8,9,10]. Para garantir a viabilidade do E. coli amostras utilizadas neste trabalho, os pré-estudos nas amostras bacterianas foram conduzidos, e a mudança contínua na adesão da superfície por 4 h implicou que as células permanecem vivas por pelo menos 4 h após o preparo da amostra (mostrado no arquivo adicional 1). Consequentemente, as amostras microbianas testadas neste trabalho foram medidas por AFM dentro de 2 h após a preparação da amostra. Todos os E. coli os genótipos foram cultivados individualmente e em épocas diferentes, e as medições de AFM foram realizadas imediatamente após o preparo dos espécimes. Em outras palavras, as amostras bacterianas não estavam alinhadas esperando o exame, então os efeitos do tempo de espera nas diferenças entre E. coli as cepas foram minimizadas. Os dados quantitativos para cada E. coli cepas foram coletadas a partir das medições feitas dentro de 2 h neste estudo.
Análise estatística
Prism (GraphPad Software, USA) foi utilizado para a análise estatística neste trabalho. Os comprimentos celulares e macromoleculares foram apresentados como valores médios juntamente com o erro padrão da média (SEM). As múltiplas comparações entre E. coli os genótipos foram processados usando a análise de variância comum (ANOVA). O nível de confiança de 95% ( p <0,05) foi selecionado, e os asteriscos indicam o grau de diferença significativa que foi encontrado. O número de amostra n de cada cepa foi> 40.
Resultados e discussão
Ultraestrutura de superfície por mapeamento múltiplo
Ao digitalizar com AFM em uma escala de observação de 10 μm em uma amostra bacteriana, vários E. coli Células MG1655 podiam ser vistas, e a forma celular em três dimensões podia ser observada na imagem topográfica (Fig. 1a); os contornos nítidos das células mostradas em duas dimensões foram obtidos pela imagem do erro de deflexão (Fig. 1b), e vários túbulos além das células bacterianas puderam ser encontrados. Os filamentos ondulados (fig. 1c) foram consistentes com o aparecimento de flagelos microbianos relatados em outro trabalho, que confirmou o achado como flagelos, e os pelos mais curtos e semelhantes a pêlos também puderam ser vistos [11]. Ao diminuir a área de observação da sonda para o estudo detalhado de células microbianas individuais, a topografia mostrou uma ligeira diferença na direção vertical entre as superfícies celulares, como mostrado na Fig. 1d, e a imagem de erro de deflexão parecia fornecer mais informações morfológicas enquanto o ruído ambiental coletado foi muito para investigar a ultraestrutura da superfície celular (Fig. 1e). Quando as propriedades biomecânicas simultâneas da amostra foram medidas durante o contato entre a ponta e o objeto testado, verificou-se que a superfície bacteriana era na verdade composta por uma grande quantidade de macromoléculas com forma e tamanho específicos, conforme foi revelado pelo mapeamento da força adesiva ( Fig. 1f); assim, a resolução morfológica em imagens topográficas e de erro de deflexão foi melhorada ainda mais com informações biomecânicas.
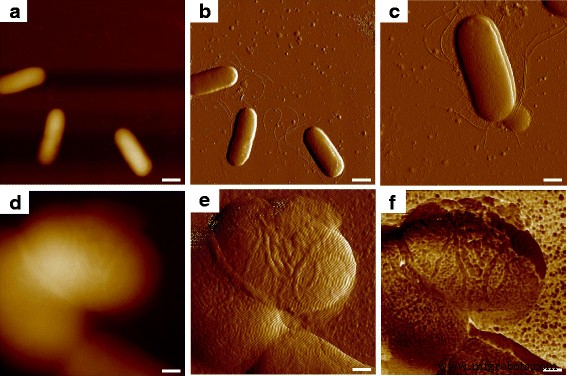
Ultraestrutura de superfície de E. coli MG1655 usando mapeamento múltiplo AFM. a e b foram as imagens topográficas e de erro de deflexão, e c foi a imagem detalhada do erro de deflexão da célula bacteriana com a expressão de flagelos e pili. Uma única célula foi então focada, onde d e e foram as imagens topográficas e de erro de deflexão, e f foi o mapeamento de adesão correspondente. As barras de escala =1 μm em a e b , 500 nm em c e 200 nm em d - f
Na Fig. 2a, a expressão de flagelos por E. coli MG1655 podia ser visto evidentemente, onde o tamanho dos filamentos era semelhante aos da Fig. 1b, com as propriedades de adesão relativamente mais baixas do que o substrato. Além disso, constatou-se que a superfície bacteriana era composta por componentes circulares que foram caracterizados por serem menos adesivos quando comparados à matriz circundante. Esta observação é semelhante aos nossos achados anteriores nas camadas de tecido da pele de camundongos, e os grãos recorrentes e comparáveis foram considerados macromoléculas, cuja estrutura é mais densa e consistente do que aquelas vistas na região intermolecular, de modo que as diferenças no desempenho de adesão poderiam ser facilmente detectado [12]. A camada mais externa do envelope celular em bactérias Gram-negativas é uma camada de proteínas de automontagem, conforme ilustrado na Fig. 2b, que é conhecida como proteína da camada superficial (camada S) [13]. As estruturas da camada S eram tradicionalmente medidas por microscopia eletrônica, enquanto os requisitos dos ambientes de vácuo e do revestimento condutor perdiam as informações nativas e em tempo real sobre as proteínas. Embora alguns pesquisadores tenham extraído as proteínas da camada S e as remontado em substrato de mica para a varredura AFM, os resultados faltaram para o desempenho in situ e em tempo real da estrutura da camada S [14, 15]. Com base na arquitetura celular e em algumas imagens anteriores da superfície microbiana por microscopia eletrônica, consideramos as macromoléculas observadas como proteínas da camada S [16].
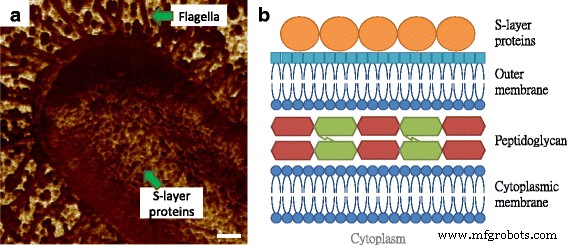
Ilustração das estruturas verticais e superficiais de E. coli células. a As macromoléculas de superfície em E. coli Célula MG1655 visualizada por mapeamento de adesão AFM. b A arquitetura molecular do envelope celular em micróbios Gram-negativos, que consiste em membrana citoplasmática, peptidoglicano, membrana externa e camada S. A barra de escala =200 nm
Comparando as medições de AFM e microscopia eletrônica de transmissão (TEM), o primeiro tem várias vantagens sobre o último, como preparação de espécime mais simples, requisitos experimentais menos restritos e aplicações de imagem mais ecologicamente corretas. A seleção de TEM no campo biológico é geralmente selecionada devido à sua capacidade de ver as organelas intercelulares e a imagem de ultra-resolução (normalmente nanômetro ou sub-nanômetro) que pode ser obtida. O mapeamento de adesão AFM no trabalho atual melhorou a resolução de características e apresentou os arranjos das macromoléculas de superfície de uma forma que não dependia do tamanho da ponta, mas sim da estrutura intrínseca da própria amostra. Além disso, essa abordagem permite a resolução em nanoescala da superfície microbiana ao longo de uma faixa de dezenas de micrômetros de área quadrada.
Diferenças de manipulação genômica em características morfológicas
Depois de observar a ultraestrutura da superfície de E. coli Células MG1655, uma questão de interesse é como as macromoléculas são organizadas em outras cepas. Os patógenos humanos E. coli CFT073 e RS218 foram examinados por AFM com os mesmos parâmetros experimentais, e não houve diferenças significativas nas formas e dimensões celulares entre esses três genótipos no tamanho de característica de 10 μm, visto na Fig. 3a-c. O mapeamento de força adesiva foi usado para identificar as proteínas da camada S, e estruturas diferentes com várias formas e tamanhos foram detectadas entre os diferentes E. coli cepas, como mostrado na Fig. 3d – f. Para facilitar a comparação, as imagens detalhadas do mapeamento de adesão do E. coli as cepas foram exibidas na Fig. 3g – i. As macromoléculas de superfície foram caracterizadas como tendo forma redonda nas células MG1655 e RS218, embora com diâmetros moleculares diferentes, que eram 38 ± 1,1 nm ( n =80) para MG1655 e 58 ± 2,7 nm ( n =46) para RS218. Por outro lado, as células CFT073 possuíam uma forma única de proteínas da camada S, que eram semelhantes a rins, com a diferença de comprimento entre dois pontos finais de 66 ± 3,2 nm ( n =44). Depois de analisar os tamanhos das proteínas da camada S desses três genótipos para comparações múltiplas, os resultados demonstraram as diferenças significativas entre essas cepas (Fig. 4).
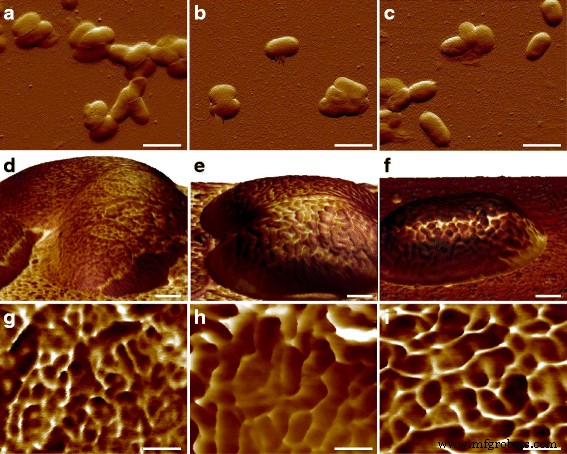
Características morfológicas do E. coli genótipos. A linha superior exibia as imagens de erro de deflexão de a MG1655, b CFT073 e c RS218. A linha do meio mostrou as topografias 3D coloridas com mapeamento de adesão em d MG1655, e CFT073 e f Células RS218. A linha inferior era o mapeamento de adesão detalhado em g MG1655, h CFT073 e i RS218. No mapeamento de adesão, a cor mais escura se refere a menos desempenhos de adesão e vice-versa. As barras de escala eram de 2 μm para a - c , 200 nm para d - f e 100 nm para g - i
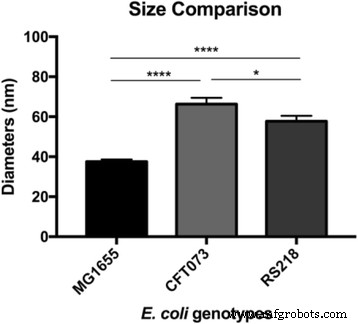
Tamanhos moleculares de E. coli genótipos. Os diâmetros das proteínas de superfície foram detectados por AFM e processados via ANOVA unilateral para as comparações múltiplas. **** p <0,001 e * p <0,05
As camadas S microbianas desempenham papéis importantes em muitas funções, que contêm células protetoras de ambientes severos, ataques de fagocitose e bactérias predatórias. Além disso, as camadas em S também servem como adesina que permite a colonização efetiva [17]. As estruturas da camada S foram bem estudadas por TEM e categorizadas em vários tipos de rede com o espaço entre centro a centro na faixa de 4-35 nm [16]. A variação entre nossos resultados de AFM e relatórios de TEM da literatura foi pensada como as diferentes metodologias de imagem, onde TEM fornece a morfologia 2D da estrutura da camada S e AFM captura a topografia 3D que inclui as múltiplas influências contribuídas por radiano celular e rugosidade e o geometria da sonda AFM.
Diferentes tipos de estruturas de camada S foram originalmente considerados possíveis para usar suas várias características taxonômicas para distinguir entre as espécies bacterianas, embora tenha sido descoberto que mesmo para uma única espécie, as manchas microbianas poderiam possuir diferentes redes de proteínas [13, 16, 18, 19]. Enquanto alguns estudos investigaram o papel da camada S na formação de filamentos, os tipos de proteínas nas membranas celulares e as diversidades genômicas de tamanho entre E. coli genótipos, as diferenças nas proteínas da camada S raramente foram observadas [20,21,22]. Os resultados do presente estudo revelaram as diferenças nas características morfológicas entre E. coli MG1655, CFT073 e RS218 e sugerem que as aparências das macromoléculas de superfície eram provavelmente específicas do indivíduo E. coli genótipo.
Conclusões
Neste trabalho, informações nanoestruturais genômicas específicas sobre a superfície bacteriana foram detectadas por mapeamento mecânico de AFM, que distinguiu as diferenças adesivas entre as macromoléculas e a matriz circundante. As macromoléculas de superfície das células microbianas foram consideradas proteínas da camada superficial, de acordo com a arquitetura molecular dos micróbios Gram-negativos. Os arranjos e tamanhos dessas macromoléculas foram considerados específicos para o E. testado. coli genótipos com formas e tamanhos distintos, sendo essas diferenças significativas pela análise estatística. Em conclusão, consideramos que a estrutura da camada S bacteriana é dependente do genoma e pode ser o método potencial para o diagnóstico rápido de doenças ou cepas microbianas associadas a micróbios. Para implementar a aplicação prática da característica da camada S, o exame de mais genótipos bacterianos é necessário para o catálogo do complemento. No momento, estamos estabelecendo o banco de dados que conecta as características morfológicas bacterianas e os desempenhos fisiológicos / patológicos e acreditamos que será um progresso promissor para a aplicação prática do exame de AFM.
Abreviações
- AFM:
-
Microscópio de força atômica
- ANOVA:
-
Análise de variância unilateral
- APTES:
-
3-aminopropiltrietoxissilano
- LB:
-
Caldo de lisogenia
- QNM:
-
Quantitativo nanomecânico
- S-layer:
-
Camada superficial
- TEM:
-
Microscopia eletrônica de transmissão
Nanomateriais
- Cientistas da IBM inventam um termômetro para nanoescala
- Pioneiros do AFM reconhecidos com o Prêmio Kavli
- O próximo jato de óleo grande está em nanoescala
- Não condutores conduzem corrente em nanoescala
- Síntese biogênica, caracterização e avaliação do potencial antibacteriano de nanopartículas de óxido de cobre contra Escherichia coli
- Efeito de superfície no transporte de petróleo em nanocanais:um estudo de dinâmica molecular
- Dispersão em pequeno ângulo de fractais gordos em nanoescala
- Dependência de ressonância de plasma de superfície localizada no dímero de nanoprisma Ag truncado desalinhado
- Fabricação de alto rendimento de nanofibras de qualidade usando eletrofiação de superfície livre modificada
- Fabricação de fossos em nanoescala com alto rendimento em filme fino de polímero usando litografia de aragem dinâmica baseada em ponta de AFM



