Análise de Actina e Organização de Adesão Focal em Células U2OS em Nanoestruturas Poliméricas
Resumo
Histórico
Neste trabalho, exploramos como as células U2OS são afetadas por matrizes de nanopilares de polímero fabricados em superfícies de vidro planas. Nosso foco é descrever as mudanças na organização do citoesqueleto de actina e na localização, número e forma das aderências focais. A partir de nossas descobertas, identificamos que as células podem ser categorizadas em diferentes regimes com base em seu comportamento de propagação e adesão em nanopilares. Uma análise quantitativa sugere que as células semeadas em matrizes densas de nanopilares são suspensas no topo dos pilares com aderências focais formando-se mais perto da periferia da célula em comparação com superfícies planas ou matrizes esparsas de pilares. Essa mudança é análoga a respostas semelhantes para células semeadas em substratos macios.
Resultados
Neste trabalho, exploramos como as células U2OS são afetadas por matrizes de nanopilares de polímero fabricados em superfícies de vidro planas. Nosso foco é descrever as mudanças na organização do citoesqueleto de actina e na localização, número e forma das aderências focais. A partir de nossas descobertas, identificamos que as células podem ser categorizadas em diferentes regimes com base em seu comportamento de propagação e adesão em nanopilares. Uma análise quantitativa sugere que as células semeadas em matrizes densas de nanopilares são suspensas no topo dos pilares com aderências focais formando-se mais perto da periferia da célula em comparação com superfícies planas ou matrizes esparsas de pilares. Essa mudança é análoga a respostas semelhantes para células semeadas em substratos macios.
Conclusão
No geral, mostramos que a combinação de nanofabricação de alto rendimento, microscopia ótica avançada, ferramentas de biologia molecular para visualizar processos celulares e análise de dados pode ser usada para investigar como as células interagem com superfícies nanoestruturadas e no futuro ajudarão a criar substratos de cultura que induzem função celular.
Resumo gráfico
Histórico
In vivo, as células normalmente residem em um ambiente 3D complexo denominado matriz extracelular (ECM). O ECM não serve apenas como um suporte estrutural para as células, mas também é um transportador de sinais biomecânicos e bioquímicos e, portanto, regula uma série de processos, como morfogênese do tecido, homeostase e diferenciação. É composto por água, polissacarídeos e proteínas [1,2,3,4], e a composição varia entre os tipos de tecido.
Motivados pela necessidade de criar modelos de cultura de células que representem melhor as condições in vivo, os pesquisadores têm cada vez mais começado a estudar o comportamento celular também em matrizes 3D e em sistemas “semi-3D”. Uma série de diferenças nos fenótipos celulares entre substratos planos e sistemas com maior dimensão foram identificados [5, 6]. Por exemplo, características como viabilidade, proliferação, diferenciação e morfologia são conhecidas por diferirem entre células em superfícies planas e células embutidas em matrizes 3D [3, 7].
Substratos semelhantes a in vivo variam de substratos "semi-3D" / 2,5D, como superfícies planas decoradas com várias nanoestruturas, a sistemas "true-3D", como géis de colágeno ou matrizes matrigel [8,9,10,11]. Além disso, o posicionamento controlado de ligantes em superfícies pode dar novos insights sobre como as células interagem com vários padrões químicos [12,13,14]. Também fatores mecânicos, como rigidez da estrutura ou mesmo a química da superfície, mostraram influenciar a função celular [15,16,17]. Para tanto, um grande número de diferentes substratos para estudos celulares têm sido desenvolvidos [3,18,19,20,21,22].
Também foi sugerido que os sistemas de cultura 3D mais precisamente poderiam prever o efeito in vivo de uma droga e, portanto, esses sistemas poderiam encontrar aplicações na descoberta de drogas [16, 23, 24]. O controle preciso de padrões topográficos em nanoescala também pode ser usado para regular a morfologia celular. Por exemplo, rugas e sulcos podem ser usados para recriar o alinhamento estriado dos cardiomiócitos e, assim, representar melhor as condições fisiologicamente relevantes para modelar várias doenças [25, 26].
O citoesqueleto da célula que está conectado à ECM é facilitado por adesões focais (FAs), um complexo multiproteico que inclui integrinas de superfície celular e proteínas-esqueleto. Dependendo de um conjunto complexo de mecanismos regulatórios, os FAs se formam e se desmontam a uma taxa de renovação necessária para o movimento para a frente, por exemplo, na migração celular. Sabe-se que os FAs exercem força mecânica sobre a MEC e, inversamente, a força que exerce a MEC nas células é conhecida por influenciar a afinidade da integrina e a avidez na membrana [27].
Uma das proteínas conhecidas por ser parte integrante dos FAs é a vinculina. É uma das proteínas ligantes envolvidas na ancoragem da F-Actina ao complexo de integrina. A falta de vinculina altera a morfologia, adesão e motilidade celular [28], e prejudica a capacidade das células de transduzir força para o substrato [29,30,31]. A vinculina não está apenas envolvida na conexão mecânica do citoesqueleto de actina aos complexos de integrina, mas também tem a capacidade de reticular e agrupar os filamentos de actina [32,33,34], modificar os feixes de actina existentes [35], capar os filamentos de actina, nuclear novos locais de polimerização de actina [36] e recrutar modificadores de actina [37].
As células respondem às matrizes 3D mudando o número e o tipo de adesão célula-substrato e induzem mudanças na organização espacial do citoesqueleto. Estas modificações à sua vez influenciam a distribuição, o tamanho e a dinâmica das aderências formadas [4, 38,39,40,41]. Este rearranjo pode levar a mudanças na proliferação celular, morfologia e motilidade [42].
Para entender a influência de ambientes 3D complexos nas células, é necessário desenvolver novos sistemas de modelo onde os processos celulares possam ser estudados e comparados a controles planos. Como a resposta celular é conhecida por depender das características físicas, mecânicas e químicas do substrato de cultivo, é desejável fabricar substratos celulares com propriedades precisamente controladas [43,44,45]. Além disso, é altamente vantajoso se as células e o substrato podem ser facilmente estudados usando técnicas de análise já estabelecidas, como microscopia óptica.
Um tipo de substrato que recentemente ganhou atenção são as superfícies planas decoradas com nano-pilares ou nanofios [18, 21, 46,47,48,49,50,51,52,53]. Em comparação com, por exemplo, hidrogéis, essas superfícies estruturadas não imitam o verdadeiro ambiente 3D, mas têm topografia de superfície bem definida. Esses substratos são normalmente chamados de 2,5D. Tais sistemas já foram aplicados para facilitar a entrega de moléculas biologicamente relevantes nas células [54, 55], para monitorar a atividade enzimática [56], para testar a mecânica nuclear [57] e para estudar como o ajuste da curvatura da membrana influencia várias células relacionadas à membrana processos [58,59,60]. Ao fabricar nanoestruturas em substratos transparentes, é possível integrar essa abordagem à microscopia óptica.
O número de combinações possíveis de diferentes linhagens celulares, tipo de nanoestrutura e geometria é alto, e exemplos da literatura são abundantes. Li et al. descreveram o comportamento celular em superfícies decoradas com nanoestruturas de fosfeto de gálio posicionadas aleatoriamente e quantificaram a fração de células com grandes FAs [61]. A morfologia celular e FA foi investigada na superfície com várias densidades de área de nanofios. Os resultados indicaram que as células semeadas em superfícies de baixa densidade estavam em contato com o substrato e formaram grandes FAs em torno das bordas das células. Grandes FAs foram detectados em uma alta fração de células nessas matrizes. Para altas densidades de área de nanofios e, as células foram suspensas no topo das matrizes de nanofios e FAs pontuais sob as células foram observados. Uma fração inferior de células nessas matrizes mostrou grandes FAs em comparação com células na superfície com baixa densidade de área de nanofio.
Buch-Månson et al. estudaram as interações de superfície nanoestruturada da célula para matrizes de nanocolunas de silício posicionadas aleatoriamente em um substrato de Si [62]. No processo de fabricação usado, a densidade de área, mas não a distância pilar-pilar, foi controlada. A investigação de FAs mostrou que as células nas matrizes com a densidade de área intermediária tinham o maior número de FAs que também tinham a forma mais assimétrica. Foi sugerido que alguns desses FAs se formaram nas paredes laterais das nanocolunas. Isso não foi observado para superfícies com densidade de nanocoluna de área baixa e alta.
Em trabalho anterior, descrevemos protocolos detalhados para a fabricação de nanoestruturas de polímero SU-8 em superfícies de vidro planas [63], e exploramos o comportamento celular para duas linhagens celulares diferentes nessas superfícies [45, 48]. Neste trabalho, usamos litografia por feixe de elétrons (EBL) para fabricar superfícies decoradas com estruturas de polímero SU-8 alinhadas verticalmente para estudar mudanças no citoesqueleto de actina e organização de AF na linha de células epiteliais de osteossarcoma U2OS. Realizamos análises qualitativas e quantitativas das mudanças induzidas pela superfície com diferentes pistas topológicas.
Resultados
Usando protocolos previamente estabelecidos, fabricamos lamínulas de vidro decoradas com arranjos precisamente definidos de nanopilares SU-8 (NP) orientados verticalmente com separação variável e geometria definida [63]. Superfícies com densidades de área NP de 456, 205, 115 e 29 NPs / 100 μm 2 (correspondendo a passos de 500 nm, 750 nm, 1000 nm e 2000 nm) foram usados. Primeiro, examinamos as tendências gerais na morfologia celular, estrutura do citoesqueleto de actina e interações célula-substrato. Seguimos isso por comparação quantitativa da morfologia de células e FAs em vários substratos nanoestruturados e controles de vidro plano. Combinamos microscopia de alta resolução com fabricação de alto rendimento para fazer análises qualitativas de pelo menos \ ({\ aproximadamente 100} \) células para cada tipo de superfície, com imagens após 24 he 48 h. No total, analisamos> 400 imagens de alta resolução e 20 conjuntos de dados 3D.
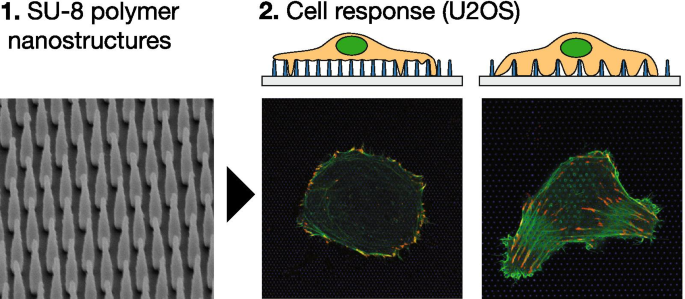
A Figura 7 mostra uma representação esquemática das matrizes NP (A, B) e imagens de microscopia eletrônica de substratos fabricados (C, D). Lâminas de vidro contendo estruturas nanofabricadas foram montadas com parafina sob fundo oco, cultura de 35 mm com as estruturas apontando para cima. As imagens de microscopia eletrônica de vista lateral e de cima para baixo mostradas na Fig. 1C, D mostram arranjos de nano-pilares com pitch e altura de 1000 nm. A Tabela 1 mostra os parâmetros geométricos das matrizes utilizadas neste trabalho, sua classificação, bem como a densidade numérica da área NP correspondente. Classificamos as matrizes NP em densas e esparsas, dependendo do comportamento de adesão celular observado (veja abaixo).
A Figura 1 mostra células U2OS representativas cultivadas em vidro (A) e superfície nanoestruturada (B – F) por 24 h. As células foram co-transfectadas com pCMV-LifeAct-GFP e pTAG-RFP-Vinculin, que permite a visualização de F-Actin e vinculina através da produção de LifeAct-TagGFP2 fluorescente (doravante:LifeActGFP) e proteínas de fusão TagRFP-vinculina, respectivamente. Rede F-actina e áreas ricas em vinculina em AFs são claramente detectadas. Na periferia da célula, o sinal LifeActGFP está próximo à membrana e, portanto, usamos esse sinal para visualizar a morfologia celular. O sinal dos NPs do SU-8 é mostrado em azul (consulte a seção “Experimental”).
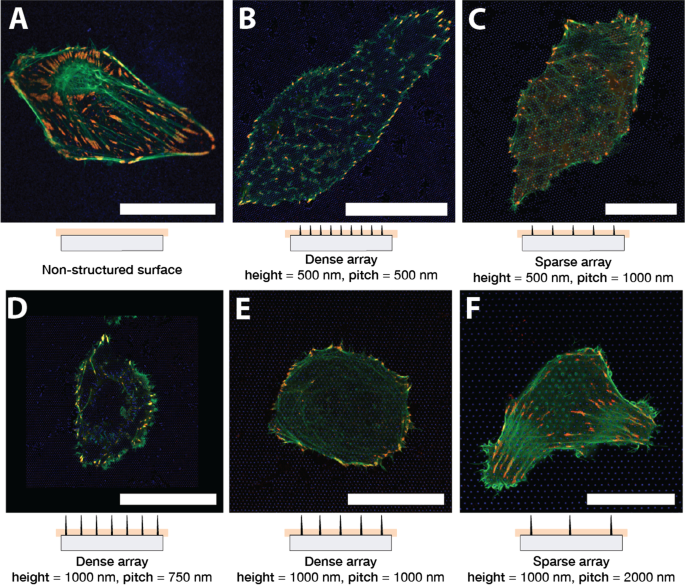
U2OS expressando LifeActGFP fluorescente (verde) e TagRFP-vinculina (vermelho) em diferentes tipos de superfície. A coloração amarela indica sinais sobrepostos dos canais LifeActGFP e TagRFP-vinculina. Abaixo de cada micrografia, uma vista lateral esquemática da matriz NP correspondente é mostrada, juntamente com a posição aproximada do plano de aquisição. Células com imagens em A superfície de vidro plana não estruturada e em arranjos de pilares com diferentes passos de arranjo e alturas de estrutura: B Passo de 500 nm e altura de 500 nm, C Passo de 1000 nm e altura de 500 nm, D Passo de 750 nm e altura de 1000 nm, E Passo de 1000 nm e altura de 1000 nm e F Passo de 2000 nm e altura de 1000 nm. Todas as imagens apresentadas são de células representativas. Barra de escalonamento 25 μm. Observe que C tem uma escala diferente das outras imagens
Os testes preliminares usando células transfectadas mostraram que as células semeadas em superfícies de vidro e estruturadas pareciam estar totalmente espalhadas após aproximadamente 6 h. Não foram observadas diferenças claras na propagação das células entre as diferentes superfícies, e a inspeção visual não revelou sinais de diminuição da viabilidade celular devido aos arranjos de pilares neste ponto ou em experimentos posteriores. Nos experimentos a seguir, as células foram transfectadas 6 h após a semeadura e, em seguida, fotografadas 24 h, 48 h após a transfecção, correspondendo à transfecção 30 h, 54 h após a semeadura. A seguir, esses dois momentos serão referidos pelo tempo de observação após a semeadura, ou seja, 24h, 48h.
As células foram semeadas em matrizes NP com altura 500 nm, 1000 nm e pitch 750 nm, 1000 nm, 2000 nm. Após a propagação inicial, as células foram observadas como redondas ou alongadas, semelhante à situação em superfícies planas. Esta morfologia geral foi considerada consistente em vários experimentos. As células semeadas em matrizes de NP esparsas geralmente tinham uma forma semelhante às células na superfície de vidro, consulte a Fig. 1F que descreve uma célula representativa em uma matriz de 2000 nm. Fibras de F-actina estavam presentes também na base das NPs e nas proximidades da superfície do vidro, indicando que as células conseguiram acessar a área próxima ao substrato. Como observado anteriormente [45, 62, 64], as células em matrizes densas normalmente pareciam estar suspensas no topo das NPs (Fig. 1B, D, E). As células em matrizes densas parecem ter F-actina menos proeminente perto da superfície do vidro, indicando que as fibras de actina não foram formadas entre os pilares nas proximidades do substrato. A relação entre a altura do NP e a separação determinou se as células aderiram ao substrato ou foram suspensas no topo da matriz NP. Isto é, por exemplo, ilustrado na Fig. 1C, onde NPs mais curtos levam à célula em contato com o substrato, enquanto NPs mais longos impedem o contato, Fig. 1E. Essas observações das fibras de actina foram ainda corroboradas por z-stacks realizados para algumas das superfícies, conforme apresentado na Fig. 2.
Para obter uma compreensão mais detalhada sobre como as células aderem à superfície estruturada e não estruturada, avaliamos a distribuição de FAs conforme visualizado pela presença da proteína de fusão TagRFP-vinculina. As células em superfícies planas normalmente formavam FAs alongados, distribuídos por baixo de todo o corpo celular, como mostrado na Fig. 1A.
Em matrizes esparsas, os U2OS foram capazes de entrar em contato com a superfície de vidro entre NPs e aderir de forma semelhante às células no vidro, mostrado na Fig. 1C, F. Para essas matrizes de NP, FAs formados no vidro entre NPs e o sinal de F-actina também foi detectado na imagem adquirida próximo à base dos NPs. Isso indicou que as células foram capazes de dobrar a membrana em torno das nanoestruturas. As células em matrizes mais densas, no entanto, como 750 nm, separação de 1000 nm e altura de 1000 nm, foram claramente impedidas de aderir ao substrato entre as nanoestruturas, como mostrado nas imagens da Fig. 1D, E que foi adquirida perto da base do NPs. No entanto, em torno da periferia, as células eram normalmente capazes de se prender ao substrato entre as nanoestruturas formadoras de FAs, muitas vezes direcionadas pela simetria da matriz de pilares subjacentes.
As células que se espalham em matrizes NP com comprimento mais curto e com um espaçamento entre pilares de 1000 nm formaram aderências tanto para a periferia quanto sob o corpo celular. A orientação da fibra F-actina foi direcionada pela simetria da matriz subjacente, como mostrado na Fig. 1C. No entanto, a localização e orientação da vinculina contendo FA não exibiu nenhum padrão claro, com a formação de FA entre NPs.
As células U2OS em pilares de 500 nm com distância entre pilares de 500 nm geralmente formaram menos e menores aderências em comparação com a superfície plana, como mostrado na Fig. 1B. Para células semeadas nesta matriz, as fibras de actina foram observadas principalmente na proximidade da superfície de vidro em posições onde terminaram em FAs. Novamente, isso é um sinal de que a rede de actina foi impedida de entrar em contato com a superfície e, portanto, as células foram consideradas suspensas no topo da matriz. No entanto, as células pareciam ter uma rede de F-actina mais intacta formando-se acima dos pilares. Partes da rede de actina que foi observada entre os pilares pareceram se alinhar com estruturas na matriz NP subjacente. Isso pode ser visto na Fig. 1B, uma vez que as fibras F-actina e FAs se formam predominantemente ao longo de uma das direções da rede, ou seja, paralelas às "linhas" abertas da matriz de pilar.
Em matrizes densas e esparsas, observamos estruturas de F-actina "semelhantes a anéis" que se formaram em torno de NP que se projetam para cima no corpo da célula. A estrutura do anel F-actina parecia ser mais proeminente em matrizes esparsas, como mostrado na Fig. 1E, F.
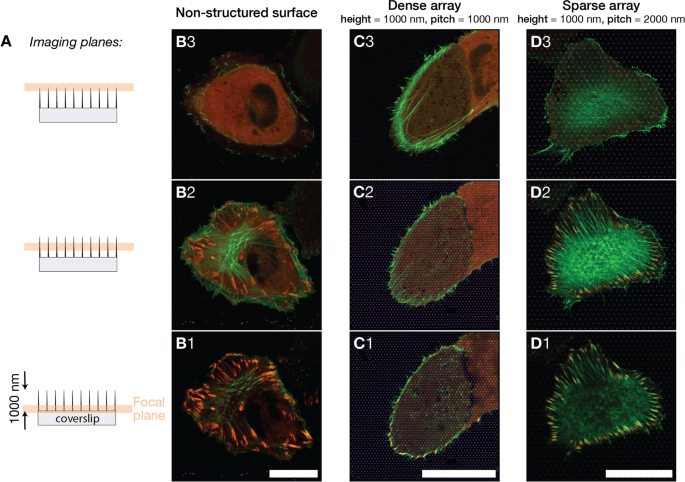
A Imagens de células U2OS em diferentes superfícies foram adquiridas em três planos focais indicados. As imagens foram obtidas próximo à base do pilar, aproximadamente na altura do meio pilar, e próximo ao ápice do pilar. Os planos de imagem foram localizados a 0,0 μm, 0,4 μm, 0,8 μm da superfície de vidro plana. B - D ) Imagens de fluorescência mescladas mostrando U2OS expressando LifeActGFP fluorescente (verde) e TagRFP-vinculina (vermelho) em diferentes tipos de superfície, a cor amarela é indicativa de sobreposição de LifeActGFP e TagRFP-vinculina. As superfícies da imagem eram B superfície de vidro não estruturada, C Matriz de pilares com inclinação de 1000 nm, D Matriz de pilares com inclinação de 2000 nm. As imagens apresentadas são representativas das células em cada tipo de superfície. A distância vertical entre os planos focais para cada tipo de superfície é de aproximadamente 400 nm. Barra de escalonamento 25 μm
Com base nos resultados apresentados acima, selecionamos três superfícies para uma descrição mais detalhada e quantitativa da morfologia celular e FAs. Nós estudamos células em arranjos densos (pitch 1000 nm, comprimento 1000 nm), arranjos esparsos (pitch 2000 nm, comprimento 1000 nm) e comparamos os resultados com células em superfícies de vidro plano usadas como controle.
Ao empregar o detector Airyscan junto com o pós-processamento de imagem dedicado, fomos capazes de realizar imagens com uma resolução xy de cerca de 140 nm e resolução z de cerca de 400 nm [65]. A Figura 2 mostra imagens de células nas três superfícies, com planos de imagem separados por aproximadamente 400 nm. A investigação de pacotes de F-actina em diferentes z . As células na superfície plana tinham uma rede de F-actina claramente visível no mesmo plano focal ou logo acima dos FAs, (ver Fig. 2-B2). Para células em matrizes densas, a rede F-actina foi encontrada em um plano focal superior dentro da célula em comparação com o plano FA, que estava em contato com o suporte de vidro (Fig. 2-C1 e C2 / C3). Para células em matrizes esparsas, a situação era semelhante à da célula em controles de vidro e rede de actina e FAs foram detectados na mesma altura (Fig. 2-D2). Esses dados apóiam a observação inicial de que as células em matrizes esparsas aderiram à superfície entre as estruturas, enquanto as células em matrizes densas foram principalmente capazes de aderir à superfície em torno da periferia celular.
Para analisar e quantificar as diferenças em FAs e morfologia celular para as três superfícies selecionadas, um script de análise de imagem baseado em Python foi usado (consulte “Experimental”). Para a análise quantitativa, mais de 300 imagens de alta resolução foram analisadas. Nessas imagens,> 400 células e> 7700 FAs foram identificados, a Tabela 2 lista o número de células detectadas e FAs para os três tipos de superfície incluídos na análise. Para todas as superfícies, as células foram fotografadas 24 h, 48 h após a transfecção. Na análise a seguir, parâmetros geométricos como área de superfície, circularidade e proporção de aspecto para células em 3 tipos de superfície e após 24 h, 48 h são comparados. Análises adicionais podem ser encontradas nas Informações Suplementares. A área de superfície, circularidade e razão de aspecto para células são mostradas na Fig. 3 e para o número de FAs, área de FA combinada por célula e fração de área de FA para célula na Fig. 4. Os parâmetros geométricos foram definidos conforme descrito no "Experimental" seção.
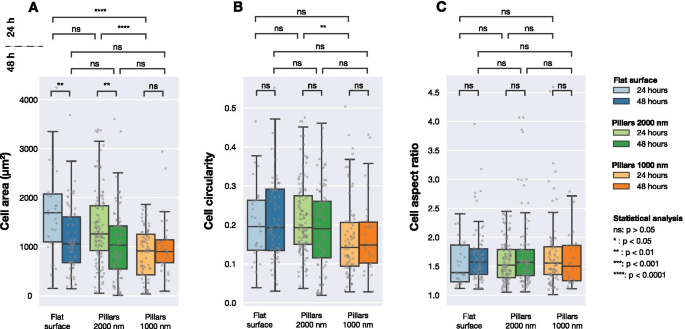
Área celular calculada, circularidade celular e proporção de aspecto celular para células U2OS que expressam proteínas de fusão LifeActGFP e TagRFP-vinculina fluorescentes em diferentes superfícies com imagens de 24 horas e 48 horas após a transfecção. A detecção da imagem foi realizada com base na intensidade do sinal do LifeActGFP. Cada ponto cinza corresponde a uma célula e os gráficos de caixa mostram valores medianos ( Q 2) bem como o primeiro ( Q 1) e terceiro quartil ( Q 3). As diferenças estatísticas entre as distribuições foram avaliadas usando o teste não paramétrico de Mann-Whitney
A Figura 3 resume os dados coletados para superfícies planas e estruturadas. Como mostrado na Fig. 3A, diferenças significativas na área celular foram observadas após 24 h de cultura de células. No entanto, após 48 h, não houve diferença significativa entre a área celular média nas superfícies estudadas. Ao considerar a circularidade celular (Fig. 3B), nenhuma diferença significativa foi detectada entre as diferentes superfícies, exceto entre as células semeadas em pilares densos e esparsos fotografados após 24 h. As células em todas as três superfícies tinham as mesmas relações de aspecto médias, conforme apresentado na Fig. 3C.
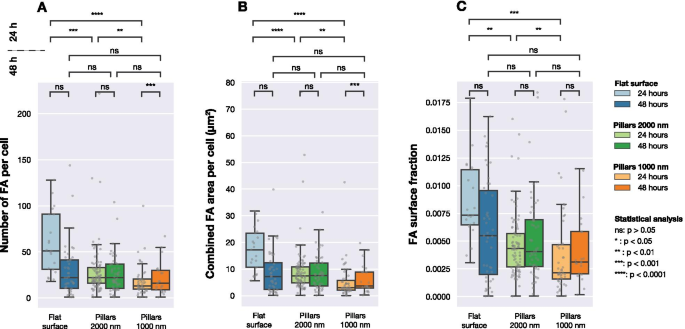
Número de FAs, área de FA combinada por célula e fração de área de FA para área de célula para células com imagens em três tipos de superfície diferentes (matrizes planas e pilares com intervalos de 1000 nm e 2000 nm). Cada ponto cinza corresponde a uma observação de uma célula. A significância estatística entre as distribuições foi determinada usando o teste não paramétrico de Mann-Whitney
A Figura 4 mostra a distribuição do número de FAs detectados por célula, a área de superfície total de FAs em cada célula e a proporção da área de FAs para a área celular. Após 24 h, o número de FAs formados por células nas três superfícies diferentes era significativamente diferente. Conforme mostrado na Fig. 4B, a área de superfície total de FA por célula era diferente para células semeadas em superfícies planas e estruturadas. O mesmo pode ser visto ao comparar a quantidade relativa de FAs (a área total de FAs detectados dividida pela área total de células) para as diferentes superfícies, como mostrado na Fig. 4C.
No entanto, após 48 h de cultura, não foram mais observadas diferenças significativas entre as populações de células. Ao considerar o número de FAs por célula, área combinada de FA por célula ou fração de superfície de FA, não foram encontradas diferenças entre as três superfícies.
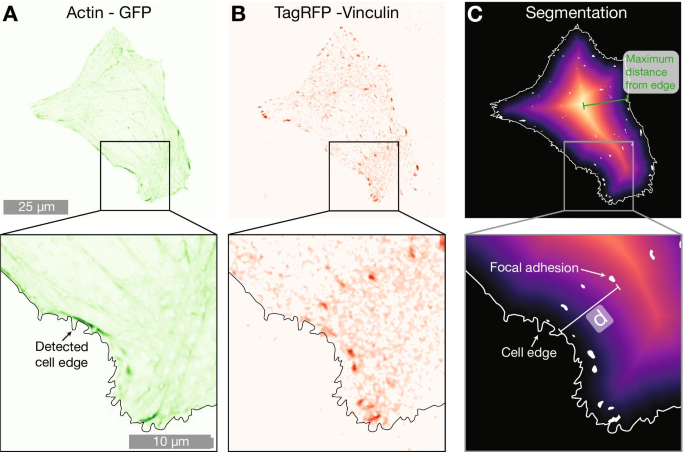
Exemplo de célula em matriz de pilar de 2.000 nm expressando A LifeActGFP (verde) e B TagRFP-vinculina (vermelho). Sobreposta em ambas as figuras está a borda da célula detectada, determinada usando o sinal da expressão de LifeActGFP (como mostrado em A ) C Mostra um mapa de distância da borda da célula detectada, bem como FAs detectados. A distância mais curta (denotada com d na figura) de cada mancha de vinculina segmentada (áreas brancas) para a periferia da célula (indicada com uma linha contínua em branco) foi calculada para todos os FAs nas imagens
Para entender se a presença de NPs influencia a localização dos FAs na célula, realizamos análises adicionais usando a localização dos FAs. Os dados microscópicos indicaram que os FAs nas células em matrizes NP densas estavam localizados mais perto da periferia da célula, como indicado pelas Figs. 1 e 2. Para quantificar essa tendência, calculamos a distância mais curta de cada FA até a borda da célula. Isso foi realizado conforme ilustrado na Fig. 5. F-actina foi usada para determinar a localização da periferia e, pela construção de mapas de distância, a distância entre cada centro de um FA detectado até a periferia da célula foi calculada. Para contabilizar as diferenças nos tamanhos das células, normalizamos as distâncias entre a borda da célula detectada e FA pela distância máxima da borda ao centro geométrico de cada célula (uma distância que é equivalente ao raio para células que têm uma forma circular). Os dados normalizados pela distância máxima são apresentados na Fig. 6 e na Tabela 3. Para células em superfícies planas, os FAs são distribuídos mais em direção ao centro da célula, enquanto em ambos os arranjos esparsos de 2000 nm e densos de 1000 nm, os FAs estão localizados mais perto da periferia da célula. Este efeito é mais notável para matrizes densas com pitch de 1000 nm. Os resultados de uma abordagem de normalização alternativa, em que os locais de FA foram normalizados pela área de superfície celular, estão incluídos nas Informações Suplementares. Esses dados mostram as mesmas tendências qualitativas dos dados normalizados pela distância máxima até a borda em cada célula apresentada na Fig. 6.
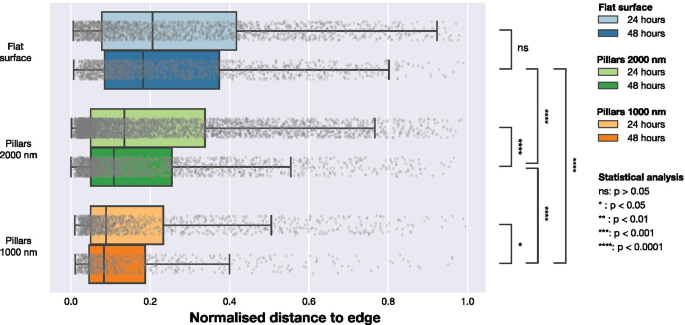
Distribuição das posições dos FAs em relação à borda da célula mais próxima normalizada pela distância máxima do centro geométrico da célula à borda. As distâncias foram obtidas calculando a distância de cada FA observada até a borda da célula definida pelo sinal LifeActGFP e plotada para os três tipos de superfície em 24 he 48 h. Os pontos cinza representam observações individuais de FAs e suas distribuições são resumidas nos gráficos de caixa. Para testar a probabilidade de que os FAs de diferentes superfícies e pontos de tempo fossem da mesma distribuição, os testes de Mann-Whitney foram realizados em todas as distribuições com níveis de significância denotados na figura
Discussão
A organização do citoesqueleto de actina e a formação de aderências são processos extensivamente estudados em superfícies planas. O presente estudo foi desenhado para investigar e quantificar as mudanças na organização do citoesqueleto de actina e aderências focais em arranjos nanopilares. Neste estudo, não investigamos como os arranjos de nanopilares afetam a migração celular. No entanto, esperamos que as células nas matrizes densas possam ter maior motilidade, como observado anteriormente nas mesmas matrizes de pilar para fibroblastos embrionários de camundongo [45].
Permitir que as células se espalhem e adiram à superfície por um período prolongado de tempo, torna possível observar a organização do citoesqueleto de actina e a presença de AGs totalmente maduros. 24 h após a semeadura, observamos diferenças significativas na área celular, circularidade e proporção de aspecto para as células semeadas nas superfícies. No entanto, nenhuma diferença significativa foi detectada após 48 h, indicando que as células após 24 h ainda não aderiram totalmente às superfícies, e que as nanoestruturas influenciam principalmente a organização de AF antes de estarem totalmente maduras. Nem após 24 h nem após 48 h qualquer formação de FA no topo ou nas laterais das NPs é observada.
Mudanças na organização do citoesqueleto de actina também estão relacionadas ao modo como a célula interage com o ambiente. Por exemplo, tanto as fibras de estresse quanto os AFs crescem quando sujeitos ao alongamento e parecem ser interdependentes funcionais [66]. Outros relataram que células mais arredondadas e localização de FA em torno das bordas são freqüentemente observadas para células semeadas em superfícies macias ou complacentes [67]. Em nossos resultados, observamos uma tendência semelhante. As células em matrizes densas tendem a mostrar fibras em torno da borda da célula, como mostrado nas Figs. 1E ou 2B. FAs parecem se formar perto da borda da célula para essas células. Especulamos que quando as células não têm uma superfície plana, como quando as células estão suspensas no topo dos pilares, a distribuição dos FAs se assemelha aos FAs em substratos macios, como os usados por Prager-Khoutorsky e colaboradores [67].
A partir de nossos resultados, observamos que as células suspensas no topo dos pilares parecem ter uma rede de actina desenvolvida acima dos pilares, mas sem a formação de FAs nos próprios pilares. Para células em matrizes esparsas, no entanto, as células parecem ser menos influenciadas pelos NPs e tanto a rede de actina quanto os FAs parecem mais "de superfície plana".
A interação entre os AFs e o citoesqueleto de actina é complexa e ainda não totalmente caracterizada. Os FAs que ligam o citoesqueleto de actina à MEC são conhecidos por atuar como pontos de tração e por promover a formação de fibras de estresse nas células. Por outro lado, as fibras de actina estão novamente influenciando a organização e maturação dos AFs. Numerosos estudos descrevem como as células tendem a ficar suspensas no topo de matrizes NP densas [61, 62, 68] e como as membranas celulares interagem com NPs únicos [69,70,71]. Essas observações são corroboradas por estudos teóricos [64] e o comportamento celular em pilares é bastante bem compreendido.
O mecanismo por trás da formação de FAs e fixação ao substrato ao redor da borda da célula em matrizes densas permanece obscuro. A este respeito, a comparação com células em um substrato macio é particularmente interessante. Para substratos macios, as fibras de actina são organizadas em forma de anel perto da borda da célula e os FAs se formam ao redor da periferia da célula [67]. On the nanopillar arrays similar type of architecture is observed, but the actin fibres are typically shorter. Similar qualitative trends in terms of actin organisation and FA formation were observed by Li et al. for cells seeded on random nanowire arrays made from gallium phosphide [61].
In our studies we also observed formation of F-actin rings around NPs. The formation of F-actin rings around NP has previously been described for fibroblasts on similar surfaces [45] and for U2OS cells on nanostructures with a range of structure sizes [58].
Contrasting our results to other studies highlight an important aspect of studies on cellular response to NP arrays:cellular response may vary considerably depending on cell type, NP material, NP geometry and as well as other parameters. For example, Buch-Månson et al. studied fibroblasts and investigations of FAs showed that cells suspended on arrays with intermediate NP density had the highest number of FAs. In our results we do not see a similar trend. However, these studies cannot be directly compared as Buch-Månson et al. studied another cell line using a system with different array geometry, surface porosity and NPs length [62].
There are also studies describing the effect FAs placement has on cells [41]. By modelling cells on planar substrates Stolarska et al. suggest that the cells can control intra-cellular stresses by three mechanisms:FA position, FA size and attachment strength. FAs around the periphery allows the cells to be more sensitive to changes in the micro-environment. This could also be an underlying mechanisms for cells on NPs. Yet, it is not obvious that the results for the planar substrate are directly transferable to NP decorated surfaces.
Cell-interactions with the surrounding environment, for flat substrate, NPs arrays or in vivo ECM, are regulated by a complex set of relations between actin organisation, membrane mechanics, cell dynamics and contact with FAs. To further explore these relations, applying flat surfaces structured with NPs could be one promising approach. Such surfaces may also aid in exploring discrepancies in the cellular response to environmental cues between different cell lines.
Conclusões
In order to create more physiologically relevant systems for cellular studies, a plethora of 3D and 2.5D approaches have been proposed. One approach is to use flat-surfaces decorated with vertically aligned nanostructures as a simple model system. High resolution live cell imaging of co-transfected U2OS cells expressing pCMV-LifeAct-GFP and pTAGRFP-Vinculin have been used to study the influence of nanopillar arrays on actin cytoskeleton focal adhesion organisation. Our present results indicate that the U2OS cells spreading on surfaces decorated with nanopillars can be categorised into three different regimes by how they respond to the nano-structures. These observed changes are quantified by analysing more than 400 high-resolution images, and indicate that tuning geometrical properties of the nanostructured surface can be used to direct cell behaviour.
More specifically, the U2OS cells were found to either contact the substrate, attach preferably around the cell edge, or be fully suspended on top of the vertical NP arrays. In the latter case, we hypothesise that the resulting reorganisation of FA and cytoskeleton is an effect analogous to what is seen for softer substrates.
Increased understanding of how cells behave on nano-structured surfaces, such as pillar arrays, could help us discover more details about complex cellular processes. For example, it is still poorly understood how changes in the actin cytoskeleton and its architecture influence cell signalling. By studying the cell response on nanostructured surfaces in a systematic way, the potential connection between actin cytoskeleton, cell adhesions and a plethora of biochemical signalling pathways could be further explored. We therefore envision that further development of the presented platform and analysis could have implications for advanced in vitro applications or for development of smarter in vivo biointerfaces.
Methods
Fabrication of Nanostructures and Sample Mounting
SU-8 nanostructures were fabricated as previously explained [63]. Briefly, 24 mm by 24 mm glass cover slips (#1.5, Menzel-Gläser, thickness 170 μm) were cleaned by immersion in acetone, isopropyl alcohol, rinsed in de-ionised water and dried. The cover slips were then oxygen plasma treated for 2 min (Diener Femto plasma cleaner, power 100 W, base pressure 0.3 torr), followed by dehydration for 10 min on a 150 °C hot plate. Samples were then placed in a desiccator containing an open vial of Hexamethyldisilazane (HMDS, Sigma Aldrich product no:440191). HMDS was applied by vapour deposition, the desiccator was pumped to low vacuum using a diaphragm pump for 5 min and the samples were kept in HMDS atmosphere for 60 min.
Substrates for EBL were prepared directly after HMDS treatment by spin coating SU-8 2001 (Microchem Corp.) to a desired thickness of 500 nm and 1000 nm. SU-8 was made fluorescent by adding either Oxazine 170 perchlorate, Rhodamine 800 or Coumarin 102 (all Sigma Aldrich) to a final concentration of 100 μg mL −1 resist. After spin coating samples were dehydrated on a hot plate at 95 °C. To mitigate charging during EBL exposure samples were then covered by a layer of conductive polymer AR-PC 5091 Electra 92 (AllResist GmbH) by spin coating at 2000 rpm for 60 s to thickness of 50 nm.
An Elionix ELS-G100 100 kV EBL-system was used to fabricate SU-8 nanopillars (NPs) with processing parameters as described in our previous work [63]. Table 1 summarise the arrays fabricated for this work. Pillar arrays were exposed using the Elionix dot-pattern generator where each pillar is exposed in a single exposure. Arrays were exposed over an area of 2000 μm by 4000 μm, with a current of 500 pA in write fields of 500 μm by 500 μm. NPs had a tip diameter of about 100 nm as a base diameter of 150 nm and 200 nm for structures of length 500 nm and 1000 nm respectively.
After EBL exposure, the samples were rinsed in DI-water to remove the conductive polymer, then post exposure baked for 2.3 min at 95 °C and developed twice in mr-Dev 600 (Micro Resist Technology GmbH) developer for 20 s, rinsed in isopropyl alcohol and dried. Samples were then treated with oxygen plasma (Diener Femto plasma cleaner, power 50 W, base pressure 0.3 torr) for 30 s to render SU-8 hydrophilic and to give it similar surface chemistry as glass by oxidising surface epoxy-groups to hydroxyl.
Fabricated structures were imaged using Scanning electron microscopy (SEM) and samples sputter coated with 5 nm Platinum/Palladium alloy deposited with a 208 HR B sputter coater (Cressington Scientific Instruments UK). SEM was performed with a FEI Apreo SEM, at 5 kV and 0.2 nA with sample 45° pre-titled stage and with additional tilting of 30°.

A Side view schematic representation of nano-structured surface mounted in petri dish. Glass slides are mounted using paraffin such that structures are pointing upwards. B Tilted schematic representation of nano-pillar array on flat surface, and two important parameters for the nano-pillar arrays (height and pitch). These figures are not drawn to scale. C , D Overview of the nanopillar arrays employed in this work. Top-down and tilted side-view scanning electron micrographs of fabricated nano-pillar array with pillars of height 1000 nm and pitch 1000 nm. Scalebars 2000 nm
When exposing the pillars, an indexing system was also exposed to make navigation during live-cell imaging more reliable. Arrays were optically inspected after fabrication to ensure free and standing pillars. The short Oxygen plasma treatment to render the SU-8 structures did not lead to any optically visible change to the structures. Lastly, the samples were mounted underneath 35 mm diameter dishes (Cellvis, Mountain View, CA, USA) with 14 mm holes and nano-structures pointing upwards, as indicated schematically in Fig. 7. As flat surfaces, areas outside the structured part of the same samples were used. Before usage, all dishes were disinfected with 70% ethanol twice and dried.
Cell Culture and Transfection
U2OS-cells (ATCC) were cultivated in Dulbecco’s modified Eagle’s Medium (DMEM Prod. 41965039, Fischer Scientific) with 10% fetal bovine serum (FBS) and kept at 5% CO2 and 37 °C. Before detachment, cells were washed with PBS and detached with Trypsin-ethylenediaminetetraacetic acid (trypsin-EDTA) and seeded on nanostrucutred or flat surfaces. For the diameter 14 mm glass wells 15,000 cells were seeded.
For the standard transfection experiments, cells were allowed 6 h for adhering to surfaces before transfection. U2OS cells were transiently transfected using Lipofectamine 2000 (Invitrogen, Fischer Scientific) by adapting the manufacturer protocol to our system. Briefly, 2 μL Lipofectamine 2000 was added to 50 μL Opti-MEM I Reduced Serum Media (Prod. 11058021, Gibco , Fischer Scientific) and incubated for 5 min at room temperature. Plasmid DNA coding for fluorescent LifeAct-TagGFP2 and TagRFP-vinculin fusion proteins were co-transfected by using 0.5 μg plasmid DNA (vinculin-pTagRFP and pCMVLifeAct plasmids) was diluted in 50 μL Opti-MEM I and incubated at room temperature for 5 min. For co-transfection of TagRFP-vinculin and pCMVLifeAct 0.5 μg of each plasmid was used.
The diluted DNA was added to the diluted Lipofectamine 2000 in a 1:1 ratio, and left to incubate for 20 min at room temperature. 40 μL of the combined transfection complex was then added to each well. After 18 h, 1.5 mL DMEM (Prod. 41965039) supplemented with 10% FBS and 1% 10000U/mL Penicillin-Streptomycin was added to each dish.
For reverse transfection experiments, the same amounts of reactants were used, but the transfection complex was added to a suspension of U2OS cells, and the suspension was then added to the wells.
Microscopy
Live cell imaging was performed usin g a Zeiss LSM 800 Airyscan with an inverted Axio Observer Z1 stand connect to a PeCon compact incubator. Imaging was performed in an humidified environment at 37 °C, with 5% CO2 fluxo. High resolution imaging was performed using a Zeiss Plan-Apochromat 63x/1.4NA DIC M27 oil objective with Cargille Immersion Oil Type 37 (n =1.51) suited for use at 37 °C. All images were taken using the system optimised pixel size both in-plane (typically 34 nm) and for stacks in the vertical axis (typically 180 nm).
To minimise imaging bias, imaging was performed in a standardised manner where each pillar array was raster scanned and cells expressing both LifeActGFP and Vinculin RFP were imaged. The high resolution images were then processed using a Zeiss algorithm for reconstruction of AiryScan images and exported as CZI-files for further manual and automatised image processing.
Image Analysis
For all cells, cell shape was based on the expression LifeActGFP fusion protein and expression of TagRFP-vinculin was used to identify FAs. Segmentation of images was performed using a script written in Python 3 [72] using CZIfile [73] (version 2017.09.12) for reading the microscopy images in Zeiss-format. The python packages Scipy [74] and Scikit-image [75] were used for multi-dimensional image processing and image segmentation respectively.
To reduce the influence from fluorescence cross-talk from pillars (due to Oxazine 170 perchlorate, Rhodamine 800 or Coumarin 102), the pillar/surface channel was used as a background and subtracted from the TagRFP-vinculin imaging channel. A median filter (size:10 pixels) was applied to remove noise from the TagRFP-vinculin channel, followed by classification of the image into regions based on their intensity value using a Multi-Otsu approach. Multi-Otsu thresholding with three classes was applied. The first class was typically the background, the second class constituted the cytosolic vinculin, whereas vinculin rich areas in FAs appeared brighter and could be classified into a third class. The quality of the image segmentation was briefly assessed by comparison to manual segmentation.
Area of cells and vinculin rich regions were described by counting pixel numbers and from this the actual area was found by correcting for the pixel size. Shape geometries were described by fitting each region with an ellipse with the same second-moment as the segmented region. In order to describe the cell area geometry, three measures were used:(1) Aspect ratio defined as the ratio of the ellipse major axis to the minor axis. (2) Circularity given as,
$$\begin{aligned} C =\frac{4\pi *\text{Area}}{\text{Perimeter}^2}, \end{aligned}$$ (1)
and roundness given as,
$$\begin{aligned} R =\frac{4*\text{Area}}{\pi *\text{MajorAxis}^2}. \end{aligned}$$ (2)
Segmented vinculin areas with a fitted ellipse that were too round (aspect ratio \(\le 1.5\)) or too elongated (aspect ratio \(\ge 8.5\)) were rejected. In addition, vinculin areas smaller than 0.05 μm 2 were filtered out. In order to find the distance between each vinculin area and the cell edge, the shortest euclidean distance between each centroid (the centre of the fitted ellipse for each vinculin area) and the cell edge was calculated.
Statistical Analysis
Statistical comparisons of distributions were performed by using the non-parametric two-tailed Mann-Whitney test neither assuming normal distribution nor equal standard deviation. P -values \(\ge {0.05}\) were considered to represent a non-significant (ns) difference between the two populations. Significant values were denoted with * for p in 0.01 to 0.05, ** for p in 0.001 to 0.01, *** for p in 0.0001 to 0.001 and lastly **** for p \(\le {0.0001}\).
Abreviações
- DMEM:
-
Dulbecco’s Modified Eagle’s Medium
- EBL:
-
Electron Beam Lithography
- ECM:
-
Extracellular Matrix
- EDTA:
-
Ethylenediaminetetraacetic Acid
- FA:
-
Focal Adhesion
- FBS:
-
Fetal Bovine Serum
- GFP:
-
Green Fluorescent Protein
- HMDS:
-
Hexamethyldisilazane
- NP:
-
Nanopillar
- RFP:
-
Red Fluorescent Protein
- SEM:
-
Scanning Electron Microscopy
Nanomateriais
- Exemplo de circuitos e listas de rede
- Baterias para fins especiais
- Célula solar
- Compostos de grafeno e polímero para aplicações de supercapacitor:uma revisão
- Estudo in vitro da influência das nanopartículas de Au nas linhas celulares HT29 e SPEV
- Síntese de nanocristais de ZnO e aplicação em células solares de polímero invertido
- Estudo da Força de Adesão e Transição de Vidro de Filmes de Poliestireno Fino por Microscopia de Força Atômica
- Promoção do crescimento celular SH-SY5Y por nanopartículas de ouro modificadas com 6-mercaptopurina e um peptídeo penetrador de neurônio
- Influência da rigidez elástica e adesão da superfície no salto de nanopartículas
- Avanços recentes em métodos sintéticos e aplicações de nanoestruturas de prata



