Biossensor ultrassensível para a detecção de DNA de Vibrio cholerae com nanoesferas de composto de ácido poliestireno-co-acrílico
Resumo
Um biossensor eletroquímico ultrassensível para a determinação de Vibrio cholerae patogênico ( V . cólera ) O DNA foi desenvolvido com base na matriz transportadora de DNA do composto de nanoesferas de látex de poliestireno-co-acrílico (PSA) (PSA-AuNPs). A voltametria de pulso diferencial (DPV) usando um marcador de oligonucleotídeo de antrraquninona eletroativa foi usada para medir a resposta do biossensor. O carregamento de nanopartículas de ouro (AuNPs) no eletrodo de partícula de látex de DNA amplificou significativamente a corrente faradaica de hibridização de DNA. Juntamente com o uso de uma sonda relatada, o biossensor demonstrou alta sensibilidade. O biossensor de DNA produziu uma faixa de resposta linear ampla e reproduzível para o DNA alvo de 1,0 × 10 −21 para 1,0 × 10 −8 M (desvio padrão relativo, RSD =4,5%, n =5) com um limite de detecção (LOD) de 1,0 × 10 −21 M ( R 2 =0,99). O biossensor obteve valores de recuperação satisfatórios entre 91 e 109% ( n =3) para a detecção de V . cólera DNA em amostras enriquecidas e pode ser reutilizado por seis ensaios de DNA consecutivos com um valor de RSD de repetibilidade de 5% ( n =5). A resposta do biossensor eletroquímico foi estável e sustentável em 95% de sua resposta original até 58 dias do período de armazenamento.
Histórico
Vibrio cholerae , um patógeno de origem alimentar, pode causar epidemias de cólera em humanos por meio da diarreia aquosa aguda. O surto de cólera ainda é um problema sério em algumas partes do mundo, por ex. Ásia e África, e leva a um baixo status socioeconômico [1,2,3,4]. Este patógeno entérico é uma das principais causas de morbidade e mortalidade, principalmente em países em desenvolvimento [5]. A epidemia e pandemia de cólera em várias regiões são causadas principalmente por V. cólera serogrupos O1 e O139 [1, 2, 6]. V. cólera O serogrupo O1 tem dois serotipos principais, ou seja, Inaba e Ogawa, que podem se alternar entre as epidemias de cólera. O terceiro sorotipo, Hikojima, também existe, mas é raro e instável. Os genes responsáveis pela biossíntese do antígeno O1 foram designados como rfb. A mutação que define os serotipos Inaba e Ogawa é uma mutação de deleção única no gene rfbT [7]. No entanto, também foram relatados surtos ocasionais de origem alimentar em humanos com diarreia grave como sendo causados por não-O1 / não-O139 V. cólera através da ingestão de frutos do mar mal cozidos [8] ou exposição a um ambiente aquático contaminado [9]. A primeira epidemia de V. cólera O139 ocorreu em 1992 em Bangladesh e na Índia e então se espalhou rapidamente para outros países do Sudeste Asiático [10]. Em todo o mundo, em 2005, um total de 131.943 casos de cólera e 2.272 mortes foram notificados à Organização Mundial da Saúde (OMS) [11].
A busca por um método eficaz de monitoramento ou diagnóstico de toxigênicos V. cólera a bactéria é fundamental para controlar a epidemia de cólera. Identificação tradicional de V. cólera é frequentemente alcançado através do isolamento e triagem da bactéria, onde envolve o pré-enriquecimento em água peptonada alcalina (APW) seguido pelo isolamento de V. cólera no meio de cultura tiossulfato citrato, sal biliar, sacarose, ágar (TCBS) e identificação por teste de aglutinação em lâmina com anti-soros específicos [12]. No entanto, esta técnica é muito demorada e trabalhosa, e o resultado obtido vários dias depois teria significado um atraso no diagnóstico clínico e no tratamento do paciente. Método molecular envolvendo amplificação por PCR para a detecção de V. cólera [13] reduziu o tempo de diagnóstico [14], mas o método de PCR requer profissionais qualificados e uma infraestrutura cara que é difícil de realizar em países com poucos recursos. Testes de diagnóstico rápido com base no princípio da imunocromatografia foram relatados para detecção discreta ou simultânea de V. cólera serogrupos O1 e O139. Algumas outras técnicas baseadas em imunoensaios usadas para a detecção de V. cólera são, tais como ensaio de imunoabsorção enzimática (ELISA), coaglutinação, imunofluorescência e microbalança de cristal de quartzo (QCM). No entanto, a maioria dessas técnicas requer instrumentação sofisticada, tempo de ensaio longo e pessoal altamente qualificado com conhecimento técnico detalhado [15,16,17,18,19,20].
Os métodos eletroquímicos têm atraído atenção considerável na detecção de ácido nucleico devido à sua alta sensibilidade, especificidade, simplicidade e protocolo econômico, bem como detecção rápida e compatível com a tecnologia de microfabricação [21, 22]. Além disso, o método eletroquímico que é acoplado a tecnologias de miniaturização pode ser usado para análise descentralizada in situ, por exemplo, o dispositivo biossensor de DNA baseado em chip microfluídico, que é muito útil para o ambiente prático [23]. Há uma grande variedade de eletrodos usados nas medições eletroquímicas, como eletrodo de carbono vítreo (GCE), eletrodo de pasta de carbono (CPE), eletrodo de ouro e eletrodo de platina. Recentemente, os estudos têm se concentrado no uso de eletrodos impressos em tela (SPEs) devido a algumas de suas propriedades exclusivas, como fornecer baixa corrente de fundo e ampla janela de potencial, custo-benefício visto que a tinta de carbono é barata e pode ser produzida em massa .
Existem alguns métodos eletroquímicos relatados para a detecção de V. cólera consistia em uma série de etapas complexas. Um eletroquímico V. cólera genossensor relatado por Liew et al. [24] usaram o método de adsorção eletroquímica para imobilizar a sonda de DNA no carbono SPE. As partículas de PSA de multicamadas modificadas com AuNPs liofilizadas com polieletrólitos formaram um bioconjugado com avidina para funcionar como o marcador repórter no ensaio de hibridização de DNA em sanduíche. No entanto, a adição de estabilizador de sorbitol foi necessária para preservar os bioconjugados PSA-AuNPs-avidina e alongar o período operacional do biossensor de DNA em até 30 dias. Eletroquímica enzimática V. cólera O biossensor de DNA foi recentemente desenvolvido por Yu et al. [25], em que a sonda de DNA marcada com fosfatase alcalina conjugada com antifluoresceína tiolada (anti-FCAP) foi ligada ao ouro SPE por meio da química do ouro-tiol. O DNA alvo foi marcado com uma fluoresceína universal para permitir o reconhecimento de hibridização de DNA alcançado através da conversão enzimática de α-naftil fosfato em α-naftol eletroativo. No entanto, este esquema de detecção exigiu um longo tempo de ensaio de aproximadamente 95 min para a hibridização do DNA, marcação dos híbridos de DNA com enzima funcional seguida pela incubação do eletrodo em substrato de fosfato de α-naftilo eletroinativo antes que uma medição amperométrica possa ser feita. Outro biossensor eletroquímico enzimático de DNA baseado em carbono SPE revestido com avidina conjugado com sonda de DNA biotinilado foi desenvolvido por Low e membros da equipe [26]. Uma sonda repórter marcada com digoxigenina (DIG) também foi usada nesta estratégia de dupla hibridação que flanqueava a sequência de cDNA. O anti-DIG ligado à peroxidase de rábano (anti-DIG-HRP) foi empregado como marcador eletroquímico, que poderia simultaneamente catalisar a oxidação de 3,3 ′, 5,5′-tetrametilbenzidina (TMB) com a redução de H 2 O 2 para produzir uma transferência de elétrons para a superfície do eletrodo para a transdução eletroquímica do evento de hibridização de DNA. Um projeto fácil de biossensor de DNA baseado em eletrodo de vidro revestido com ouro imobilizado com sonda de DNA tiolado foi descrito por Patel et al. [22] para detecção rápida de V. cólera , e azul de metileno foi usado como o indicador de hibridização de DNA. No entanto, a faixa de detecção linear do sistema está confinada a níveis de μM, o que limitou sua aplicação em amostras clínicas.
Nanopartículas de látex-ouro foram empregadas anteriormente como o marcador de hibridização de DNA via ligação de avidina / biotina à sonda de DNA na detecção de V. cólera [24], patógeno de peixes Aphanomyces invadans [27], E. coli [28], e hibridização inespecífica de DNA [29], em que as esferas de látex foram revestidas com uma multicamada de polieletrólito antes dos coloides carregados negativamente de nanopartículas de ouro serem eletrostaticamente ligados a elas. Kawde e Wang [29] anexaram as partículas de látex PSA a uma sonda repórter de DNA para ser usada como marcador de hibridização de DNA via carregamento de partículas de látex revestidas com estreptavidina com AuNPs revestidas com biotina. Kuan et al. [24, 27] e Liew et al. [24, 27] também relataram o mesmo método de ligação avidina-biotina usando conjugados de ouro-PSA-sonda de DNA. Pinijsuwan et al. [28] relataram o uso de método eletrostático para o carregamento de partículas de PSA ligadas à sonda repórter de DNA e as partículas de PSA revestidas com ouro de polieletrólito foram usadas como um marcador para hibridização que amplificou a resposta de corrente DPV.
Neste estudo, estamos relatando uma abordagem diferente de imobilização de DNA usando as nanopartículas de látex-ouro como substrato de imobilização de sonda de DNA para desenvolver um sistema de detecção altamente sensível para V. cólera DNA. A imobilização do DNA foi realizada com um procedimento muito simples e rápido usando cloridrato de 1-etil-3- (3-dimetilaminopropil) carbodiimida / N-hidroxisuccinimida (EDC / NHS) química como reagente de acoplamento para melhorar a eficiência de imobilização [30] no látex carboxilado superfície da partícula. A detecção de hibridização de DNA foi baseada no ensaio do tipo sanduíche, que envolveu a reação de hibridização entre a sonda de DNA imobilizada e a sequência alvo seguida por uma sonda de sinal / repórter. O sal de sódio monohidrato do ácido antraquinona-2-sulfônico (AQMS) foi empregado como um marcador eletroquímico para monitorar o evento de hibridização. As partículas de látex de tamanho submícron propostas melhoraram a capacidade de ligação da sonda de DNA, e a sensibilidade do biossensor de DNA foi aumentada com a incorporação das nanopartículas de ouro altamente condutoras (AuNPs). O biossensor de DNA demonstrou sensibilidade excepcional para a detecção de V. cólera cDNA e limite de detecção extremamente baixo em níveis zeptomolares em comparação com a tecnologia avidina-biotina relatada até agora [24, 26].
Métodos
Produtos Químicos e Reagentes
O estireno (St) e o ácido acrílico (AA) foram adquiridos à Fluka. HAuCl 4 · 3H 2 O, citrato trissódico desidratado, dodecilsulfato de sódio (SDS), cloridrato de 1-etil-3- (3-dimetilaminopropil) carbodiimida (EDC) e N-hidroxisuccinimida (NHS) foram obtidos da Sigma-Aldrich. Persulfato de amônio (APS), ácido bromídrico e bromo foram fornecidos por Riedel-De Haën, Ajax Finechem e Panreac, respectivamente. Todas as soluções químicas foram preparadas com água desionizada. Tanto o alvo de 30 bases quanto os oligonucleotídeos sintéticos com incompatibilidade foram adquiridos da Bio Service Unit (NSTDA). O DNA não complementar (ncDNA) e a sonda de sinal eram da Sigma e a sonda de captura modificada com 5'-amino era da Bioneer. A sonda de captura foi preparada em 0,05 M de tampão de fosfato de potássio (pH 7), enquanto as soluções de DNA alvo, alvo incompatível, sonda repórter e DNA não complementar foram preparadas em tampão de fosfato de sódio (0,05 M, pH 7). As sequências de oligonucleotídeos utilizadas no presente estudo são mostradas na Tabela 1.
Aparelho
A medição eletroanalítica foi realizada com um potenciostato / galvanostato (Autolab PGSTAT12, Metrohm) equipado com o software GPES (4.0.007). Os experimentos voltamétricos foram realizados com um sistema convencional de três eletrodos consistindo em um eletrodo de trabalho serigrafado em pasta de carbono (SPE) (Quasense, Bangkok, Tailândia), eletrodo de referência Ag / AgCl (3 M de KCl) e haste de platina ( 2 mm de diâmetro) contra-eletrodo. A técnica de voltametria de pulso diferencial (DPV) foi usada para todas as investigações eletroquímicas com potencial de passo de 0,02 V e taxa de varredura de 0,5 V / s de 0,0 a +1,0 V em 4,5 mL de tampão de medição (0,05 M de tampão de fosfato de potássio) em pH 7 e temperatura ambiente. Todos os potenciais medidos neste estudo foram encaminhados para eletrodo Ag / AgCl, e soluções homogêneas foram preparadas em banho de sonicação (Elma S30H). Um microscópio eletrônico de varredura (SEM, LEO 1450VP) foi usado para determinar o tamanho e a distribuição das esferas de látex.
Métodos
Síntese de nanopartículas de ouro coloidal
AuNPs coloidais foram preparados por redução com citrato de sódio seguindo o método de Turkevich [31]. Resumidamente, cerca de 10 mL de 5 mM de HAuCl 4 · 3H 2 O foi dissolvido em 180 mL de água desionizada e fervido sob condição de agitação contínua em um dispositivo combinado de placa quente com agitador magnético. Dez mililitros de 0,5% ( w / v ) de citrato trissódico foi então adicionado à solução em ebulição, e a cor da solução foi observada mudando gradualmente de vermelho pálido para vermelho rubi.
Preparação de Partículas de Látex
As partículas de látex foram preparadas por reação de copolimerização em emulsão sem sabão, conforme descrito por Polpanich et al. [23] com algumas modificações. Em resumo, cerca de 190 g de água desionizada foram purgados com gás nitrogênio em um frasco de três gargalos submerso em um banho de água por ~ 1 h sob agitação a 350 rpm. Vinte gramas de St e 0,5 g de AA foram então adicionados e a temperatura foi mantida a 70 ° C. Depois disso, cerca de 0,2 g de APS foi adicionado em 10 mL de água desionizada seguido por derramamento na formulação no frasco de três gargalos para iniciar a reação de polimerização radical, e o processo de polimerização continuou por 8 h. As esferas de látex de carboxila sintetizadas foram colhidas por centrifugação com água desionizada duas vezes a 13.000 rpm por 20 min [23, 27, 28] e ressuspensas em água desionizada à temperatura ambiente (25 ° C) até o uso. A morfologia e o tamanho médio das partículas de látex PSA foram determinados por microscopia eletrônica de varredura (MEV).
Modificação da superfície SPE e imobilização da sonda de DNA
Antes da modificação da superfície, o SPE de carbono foi enxaguado completamente com água desionizada e, em seguida, revestido por gota a gota com a suspensão de PSA a 3 mg / mL e deixado secar ao ar em condições ambientais seguido de vazamento com 5 mg / mL de AuNPs coloidais . As características eletroquímicas do carbono SPE antes e depois da modificação com partículas de látex e AuNPs foram examinadas com o método CV. O SPE de carbono modificado com látex-AuNPs (PSA-AuNPs-SPE) foi então enxaguado com água desionizada e imerso em 0,1 M de tampão de fosfato de potássio (pH 5) contendo reagentes de reticulação de carbodiimida, ou seja, 0,002 M de EDC e 0,005 M de NHS por 2 h [32] antes de imersão durante a noite em 0,05 M de tampão de fosfato de potássio (pH 7) contendo 5 μM de sonda de captura. Depois disso, o PSA-AuNPs-SPE modificado com DNA (DNA-PSA-AuNPs-SPE) foi completamente lavado com tampão de fosfato de potássio (0,05 M, pH 7) para remover a sonda fisicamente adsorvida. O eletrodo de DNA foi imerso em 0,05 M de tampão de fosfato de sódio a pH 7 contendo DNA alvo linear (1 μM) e rótulo de hibridização de DNA AQMS (5 mM) para hibridização parcial por 1 he mais tarde mergulhado em 0,05 M de tampão de fosfato de sódio (pH 7) condicionado com 1 μM de sonda repórter e 5 mM de AQMS por mais 1 h para o processo de hibridização de DNA completo. Finalmente, o eletrodo de DNA foi enxaguado com tampão de fosfato de potássio (0,05 M, pH 7) para a remoção de sequências de oligonucleotídeos não hibridizadas. A resposta eletroquímica de cada substância estendida anexada ao SPE foi investigada com o método DPV. A Figura 1 representa o procedimento passo a passo para o desenvolvimento de V. cólera Biossensor de DNA baseado em suporte sólido coloidal PSA-AuNPs.
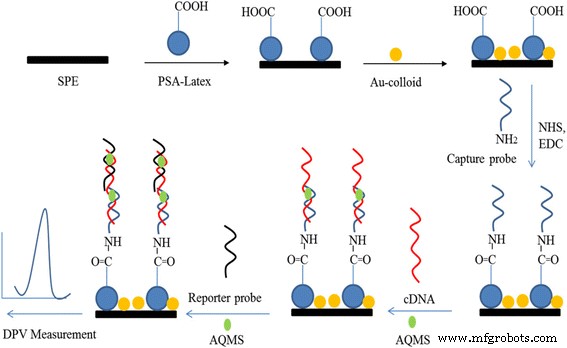
Representação esquemática do procedimento de fabricação passo a passo do biossensor de DNA
Otimização da hibridização de oligonucleotídeos sintéticos
O efeito de vários parâmetros, como sonda de DNA e concentrações de AQMS, pH, força iônica e capacidade tampão na resposta de hibridização da sonda de DNA imobilizada com sonda de sinal e DNA alvo, foi examinado antes da determinação da faixa linear dinâmica do DNA biossensor. Além disso, as durações de imobilização da sonda de captura e hibridização de DNA, bem como o tempo de vida e regeneração do biossensor também foram avaliados antes do desenvolvimento de V. cólera O biossensor de DNA estava pronto para a aplicação no experimento de spike-and-recovery. As cargas de sonda de captura e AQMS foram otimizadas variando suas concentrações de 1 a 6 μM e 0,1–5 mM, respectivamente, enquanto a concentração de DNA alvo e sonda de repórter foi mantida a 5 μM em 0,05 M de tampão de fosfato de sódio (pH 7,0). Os estudos do efeito do pH e da concentração do tampão foram conduzidos alterando o pH e a concentração do tampão de fosfato de sódio de pH 5,5 para 8,0 e 0,001 para 1.000 M, respectivamente. A presença de diferentes cátions na resposta de hibridização do DNA do biossensor eletroquímico de DNA foi realizada pela adição de Na + , K + , Ca 2+ e Fe 3+ íons a 1,0 M no tampão de hibridização de DNA contendo 1 mM de AQMS e 5 μM de cDNA e sonda de detecção de pH 7,0. O efeito da força iônica foi examinado pela variação da concentração de NaCl na faixa de 0,1–3,0 M em pH 7,0. A faixa dinâmica do biossensor de DNA foi então determinada em 1,0 × 10 −21 para 1,0 × 10 −8 M V. cólera cDNA usando uma concentração de sonda de sinal constante a 5 μM e pH 7,0. A duração da imobilização da sonda de DNA foi determinada mergulhando o eletrodo de DNA em 5 μM de solução de sonda de captura (pH 7,0) entre 1 e 13 h, e a resposta DPV foi tomada a cada 1-2 h. Enquanto isso, o tempo de hibridização do DNA foi determinado permitindo que a reação de hibridização do DNA ocorresse entre 15 e 180 min, e a resposta do biossensor de DNA foi registrada a cada 15-30 min. A vida útil do biossensor de DNA foi determinada medindo periodicamente a resposta do biossensor de DNA para a detecção de 5 μM V. cólera cDNA por 120 dias. A análise foi conduzida em cinco repetições usando um novo eletrodo de DNA para cada ensaio de hibridização em sanduíche. A regeneração do eletrodo de DNA foi feita usando 0,1 M de solução de NaOH por 4 min, e a re-hibridização do biossensor de DNA (60 min) foi realizada usando solução de re-hibridização contendo 5 μM de cDNA e sonda de detecção e 1 mM de AQMS a 2,0 M de força iônica em 0,05 M de tampão de fosfato de potássio (pH 7,0). O experimento de regeneração foi conduzido em seis repetições.
V. cólera Quantificação usando biossensor de DNA eletroquímico baseado em PSA-AuNPs
Vários V. cólera cepas bacterianas, nomeadamente J2126-I, J2126-II, J3324-I, J3324-II, J3330-I, J3330-II, CDHI 5294-II e UVC1324 incluindo Citrobacter freundii (CF-I) e Citrobacter freundii (CF-II) foram obtidos do Laboratório de Microbiologia, Faculdade de Ciências Aplicadas, AIMST University, Kedah. A extração de DNA genômico foi então conduzida sobre essas bactérias usando QIAGEN Genomic-tip 500 / G de acordo com o protocolo do fabricante. O DNA extraído foi então diluído 100 vezes usando tampão de fosfato de sódio (0,05 M, pH 7,0). Cerca de 300 mL do DNA extraído contendo 2,0 M de NaCl e 1 mM de AQMS foram sonicados por 15 min para liberar as quebras de DNA. Em seguida, a sonda de DNA imobilizada foi embebida por 1 h para permitir que o processo de hibridização de DNA ocorresse e foi lavada cuidadosamente com tampão de fosfato de potássio 0,05 M (pH 7,0) para remover o DNA não ligado. A avaliação da resposta do biossensor de DNA com base na corrente de pico do DPV foi medida e comparada com a resposta de corrente gerada pelo eletrodo de DNA sem reação com cDNA como sinal de controle. Cada experiência foi realizada em triplicata nas mesmas condições experimentais. Comum t teste foi usado para determinar a diferença significativa entre a resposta do biossensor de DNA e a resposta do controle. A recuperação de V. cólera J3324 e V. cólera DNAs UVC1324 em 1,0 × 10 −4 μg μL −1 , 1,0 × 10 −5 μg μL −1 , e 1,0 × 10 −6 μg μL −1 adicionado ao tampão de hibridação foi então realizado usando o proposto biossensor eletroquímico de DNA baseado em PSA-AuNPs.
Resultados e discussão
Morfologia de partículas de látex e comportamento eletroquímico de SPE modificada por nanopartículas de látex-ouro.
A Figura 2 mostra a micrografia SEM das esferas de látex carboxiladas com um tamanho médio de partícula de 186,1 ± 4,6 nm. A distribuição de tamanho uniforme das esferas de PSA permitiu a imobilização homogênea de moléculas de DNA na superfície do látex para aumentar a reprodutibilidade da resposta do biossensor de DNA. Um microscópio eletrônico de varredura (SEM, LEO 1450VP) foi usado para determinar o tamanho e a distribuição das esferas de látex.

Micrografia SEM das esferas de látex PSA sintetizadas com ampliações de 10.000
Os resultados eletrodinâmicos do SPE modificado estão tabulados na Tabela 2. A separação do potencial de pico (ΔEp) indica a cinética de transferência de elétrons do sistema. O SPE modificado com PSA (PSA-SPE) apresentou o maior valor de ΔEp devido ao lento processo de transferência de carga na camada de partícula do copolímero coloidal, o que fez com que o sistema se movesse para um estado quase reversível. No entanto, quando os AuNPs foram carregados no PSA-SPE, o decréscimo no ΔEp implica em uma melhora na taxa de transferência de elétrons na superfície do eletrodo.
A Figura 3 mostra os voltamogramas de pulso diferencial da resposta de oxidação AQMS no SPE modificado com látex e a resposta de hibridização sequencial de V. cólera Biossensor de DNA. Uma diferença significativa de corrente de pico de DPV foi observada entre os eletrodos que continham apenas microesferas modificadas com látex e sem DNA de captura imobilizado (experimentos (a) e (b)) e modificados com sondas de captura de DNA imobilizadas na presença de cDNA e sonda relatada (experimento (g )). Isso indica que as sondas de captura de DNA aminado foram imobilizadas com sucesso nas esferas de látex PSA carboxiladas revestidas via protocolo de acoplamento EDC / NHS [33]. O experimento (g) também mostra uma resposta de DPV muito maior em comparação com eletrodos de DNA na presença de ncDNA e sonda repórter relatada (experimento (d)), em DNA incompatível e sonda repórter relatada (experimento (e)), e cDNA alvo com nenhuma sonda relatada (experimento (f)). Isto é devido à hibridização completa do DNA alvo com sondas de captura e repórter através da reação de hibridização em sanduíche na superfície do biossensor de DNA, conforme demonstrado no experimento (g). Isso também mostra que o uso da sonda relatada pode aumentar o sinal de hibridização de DNA. No entanto, a corrente de DPV resultou da hibridização observada na presença de DNA alvo sem incorporação de uma sonda relatada (experimento (f)) é ainda maior do que os sinais de corrente de DPV observados para DNA não hibridizado (experimentos (c), (d) e (e)).
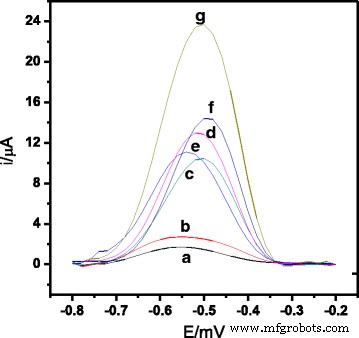
Voltamogramas de pulso diferencial sinal de AQMS dos eletrodos ( a ) PSA-SPE, ( b ) PSA-AuNPs-SPE, ( c ) captura sonda-PSA-AuNPs-SPE e quando na presença de ( d ) ncDNA e sonda repórter, ( e ) DNA incompatível e sonda repórter, ( f ) cDNA sozinho, e ( g ) cDNA e sonda repórter
Efeito do carregamento da sonda de DNA e concentração de AQMS
O efeito da concentração da sonda de DNA na resposta de hibridização de DNA foi observado através da resposta de oxidação eletroquímica AQMS. A Figura 4a mostra que a resposta do biossensor de DNA aumentou gradualmente com o aumento da quantidade de sonda de DNA imobilizada no PSA-AuNPs-SPE de 1 para 4 μM. Isso foi atribuído ao aumento da quantidade de AQMS eletroativo intercalado no DNA de fita dupla (dsDNA) para processar uma transferência de elétrons através da hélice de DNA imobilizada. Observou-se que a resposta DPV do biossensor de DNA tornou-se quase um platô entre 4 e 6 μM de sonda de DNA, o que indica que um carregamento ideal da sonda de DNA na superfície do eletrodo foi alcançado [34]. Portanto, a sonda de captura de 4 μM foi selecionada como o carregamento ideal da sonda de DNA nos experimentos subsequentes. A concentração do marcador AQMS também foi otimizada no eletrólito de medição entre 0,1 e 5,0 mM, e a concentração de AQMS a 1 mM foi considerada suficiente para a reação de intercalação de DNA ideal (Fig. 4b).
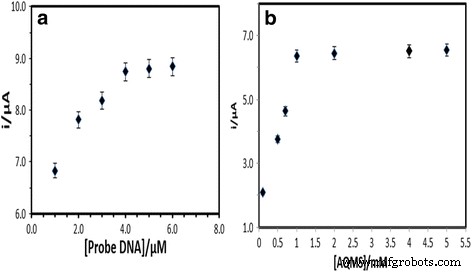
O efeito da sonda de captura ( a ) e concentrações de AQMS ( b ) na resposta do biossensor de DNA realizada com 5 μM de cDNA e sonda de sinal em tampão de fosfato de sódio 0,05 M (pH 7,0)
Efeito do pH, força iônica e capacidade tampão
A taxa de reação de hibridização do DNA depende muito do pH da solução. Como pode ser visto na Fig. 5a, sob um ambiente mais ácido, a protonação da estrutura fosfodieter do DNA reduziu a solubilidade da molécula de DNA, o que acabou diminuindo a taxa de reação de hibridização do DNA. Enquanto no meio básico, ele quebrou as fracas ligações de hidrogênio que mantinham os pares de bases do DNA juntos. A reação ótima de hibridização de DNA foi mais favorável em condição neutra, pelo que promoveu mais sondas de captura para hibridizar com DNA alvo e subsequentemente permitiu a intercalação de sondas redox AQMS para tornar o reconhecimento de hibridização de DNA em negócios. Assim, 0,05 M de tampão de fosfato de sódio a pH 7,0 foi usado como o meio de hibridação de DNA para estudos subsequentes de biossensores de DNA. Íons carregados positivamente, como Ca 2+ , Na + , K + e Fe 3+ Os íons podem interagir com a cadeia de fosfodiéster carregada negativamente do DNA. Esta reação iônica irá neutralizar a carga da molécula de DNA, diminuindo assim as repulsões estéricas entre as moléculas de DNA para facilitar a reação de hibridização de DNA. A Figura 5b representa o efeito de alguns cátions na resposta de hibridização do DNA. A resposta de hibridização de DNA foi observada para aumentar na presença de íons carregados positivamente na ordem de Na + > K + > Fe 3+ > Ca 2+ . Ambos Ca 2+ e Fe 3+ verificou-se que íons reduzem consideravelmente a resposta de hibridização de DNA devido às interações iônicas de Ca 2+ e Fe 3+ íons com íons fosfato da solução tampão, o que levou à formação de compostos de fosfato insolúveis. Isso reduziu o conteúdo iônico do meio, aumentando assim a repulsão eletrostática entre as moléculas de DNA. A maior corrente de hibridização de DNA foi obtida na presença de Na + íon devido ao seu tamanho menor e afinidade mais forte para a estrutura de DNA açúcar-fosfato em comparação com K + íon para superar o obstáculo estérico e a repulsão eletrostática entre grupos fosfato carregados negativamente de DNAs.
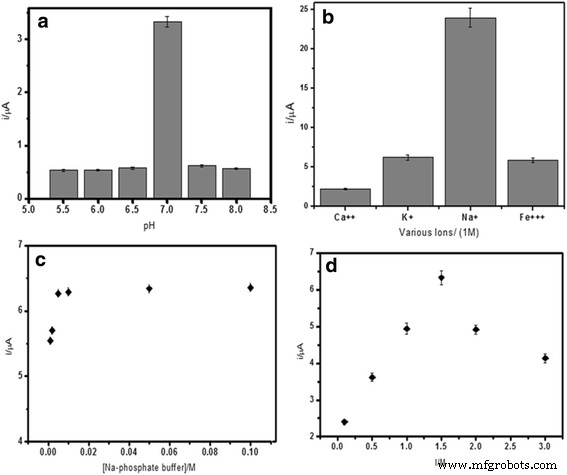
O efeito do pH ( a ) vários cátions ( b ), concentração de tampão ( c ) e força iônica ( d ) na resposta de hibridização de DNA do eletroquímico V. cólera Biossensor de DNA. A hibridização foi realizada com 5 μM de cDNA e sonda repórter seguida por intercalação com 1 mM de AQMS
Além disso, a força iônica da solução também afetaria a resposta do biossensor de DNA. A Figura 5c, d mostra a capacidade ótima do tampão e a força iônica foram alcançadas usando 0,05 M de tampão de fosfato de sódio com o pH fixado em pH 7,0 e 2,0 M de NaCl, respectivamente. Nessa condição, favoreceu ao máximo a reação de hibridização do DNA; portanto, foi produzida uma alta resposta de DPV. Com a capacidade tampão ideal e a força iônica da solução, a repulsão eletrostática entre as moléculas de DNA diminuiu, melhorando assim a reação de hibridização do DNA. Em contraste, quando um conteúdo iônico muito baixo ou muito alto foi usado, o impedimento estérico e a repulsão eletrostática tornaram-se dominantes e restringiram a hibridização das moléculas de DNA.
Estabelecimento de V. cólera Curva de calibração do biossensor de DNA
A partir do resultado mostrado na Fig. 6a, a resposta do biossensor de DNA aumentou proporcionalmente com o aumento da concentração de cDNA de 1,0 × 10 −21 para 1,0 × 10 −8 M ( R 2 =0,99) com um limite de detecção de 1,0 × 10 −21 M. O limite de detecção foi calculado com base em três vezes o desvio padrão da resposta do biossensor na curva de resposta aproximando o limite de detecção dividido pelo declive de calibração linear. A ampla faixa de detecção linear do biossensor de DNA foi devido às partículas de látex PSA altamente monodispersas e esféricas em uma faixa de tamanho submicrônico usadas como a matriz transportadora para imobilização de DNA. A camada rica em ácido acrílico na superfície das partículas de látex ofereceu um grande local de ligação para fixação de sondas de captura de DNA para criar uma superfície coberta máxima pela camada receptiva de DNA. Além disso, a incorporação de AuNPs no SPE modificado por PSA amplificou ainda mais o sinal analítico da resposta de hibridização de DNA, e isso rendeu alta sensibilidade do biossensor de DNA (Fig. 6b).
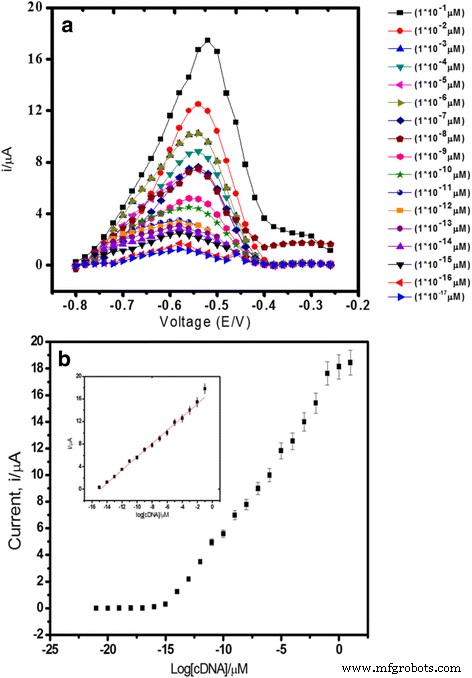
Differential pulse voltammograms (a ) and DNA biosensor linear range (b ) obtained using various cDNA concentrations from 1.0 × 10 −15 to 1.0 × 10 −1 μM V. cholerae target DNA and 5 μM signal probe
DNA Probe Immobilisation and Hybridization Times
It took about 8 h for the capture probe to be immobilised on the PSA copolymer particles surface, as illustrated by the DNA biosensor response in Fig. 7a, which showed a current increment from 1.0 to 8.0 h of capture probe immobilisation time, after which no obvious change in the DPV current was observed. Longer immobilisation time resulted in a higher amount of DNA probes immobilised onto the latex. After 8.0 h of exposure to the DNA probes, the hydrophilic functional latex with reactive carboxyl groups at the surface was presumably fully attached with the DNA probes. DNA hybridisation time, on the other hand, is the rate limiting step, which determines the response time of the DNA biosensor. Based on the DNA biosensor response trend in Fig. 7b, the response time of the V. cholerae DNA biosensor developed in this study was estimated to be about 60 min for the dual hybridisation processes to complete.
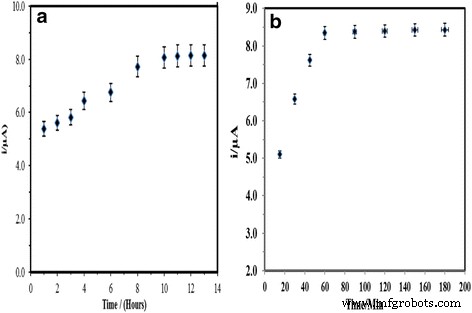
DNA probe immobilisation duration on the immobilised PSA latex colloidal particles (a ) and DNA hybridization duration of the DNA biosensor (b ) in 0.05 M potassium phosphate buffer at pH 7.0 containing 5 μM target DNA and reporter probe and 1 mM AQMS at 2.0 M ionic strength
Long-Term Stability and Regeneration of V.cholerae DNA Biosensor
Figure 8 shows the shelf life of the V. cholerae DNA biosensor. The DNA biosensor showed the highest response to the detection of 5 μM of V. cholerae cDNA for the first month of the experimental period. The electrochemical DNA biosensor was able to retain 95% of its initial DPV current after 58 days of storage period. The DNA hybridisation response was then gradually decreased to about 75% of its original response on the 75th day and exhibited 40% of its initial performance on the 100th operational day. The bioactivity of the immobilised capture probe was finally declined to 30% after 3 months of storage period. The reproducibility of each calibration point, which was repeated on five replicate DNA electrodes, gave satisfactory relative standard deviation (RSD) between 2.4 and 4.5% (n =5).
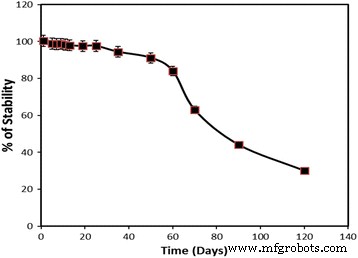
The life span profile of the fabricated V. cholerae DNA sensing electrode. The electrode was stored in 0.05 M potassium phosphate buffer (pH 7.0) at 4 °C after every DPV measurement was taken
Regeneration of biosensor indicates whether the biosensor is reusable for a series of consecutive analyses. The regeneration method used in this study was conducted based on previously reported protocol in other studies [11, 21] with slight modifications. In this study, 0.1 M of NaOH solution was used as the regeneration solution to break the hydrogen bonds between base pairs of hybridised dsDNA. With the result from Fig. 9, it is notable that the DNA biosensor response declined significantly after incubation in 0.1 M of NaOH and the percentage of the DNA biosensor response reduced from 35.1 to 5.2% relative to the DNA biosensor initial response after incubation in 0.1 M of NaOH solution from 30 to 240 s. The DNA biosensor response decreased with the increasing incubation time signifies the hydrogen bonds between hybridised dsDNA were broken up by the alkaline regeneration solution. However, rehybridisation of the DNA biosensor was able to attain almost 100% of its initial response for a consecutive six DNA analyses with a reversibility RSD of 5%.
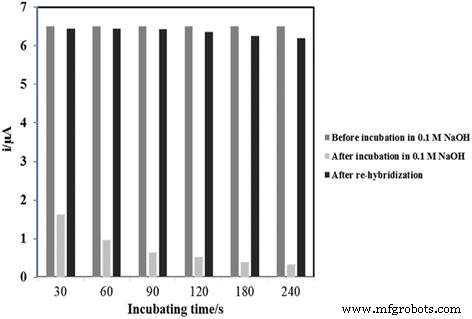
Repeatability of V. cholerae DNA biosensor using 0.1 M NaOH regeneration solution and rehybridization solution containing 5 μM cDNA and detection probe and 1 mM AQMS at 2.0 M ionic strength in 0.05 M potassium phosphate buffer (pH 7.0)
Determination of V. cholerae Bacteria with the Developed DNA Biosensor
The optimised DNA biosensor has been applied to quantify the V. cholerae DNA extracted from various V. cholerae bacterial strains. Table 3 presents the results acquired from DNA tests carried out with the hybridisation medium spiked with different strains of V. cholerae DNAs and other bacterial species at a concentration within the calibration range of the DNA biosensor. The DNA biosensor showed superior selectivity towards V. cholerae J3324–I, V. cholerae J3324–II, and V. cholerae UVC1324 with high DPV current response and low current signals were obtained for the evaluation of both Citrobacter freundii (CF-I) and Citrobacter freundii (CF-II).
Recovery of V. cholerae J3324 and V. cholerae UVC1324 DNAs at three different concentrations spiked into the hybridisation buffer demonstrated 91.4 ± 2.2% to 108.9 ± 4.8% (n =3) of recoveries percentage (Table 4). This result suggests that the proposed PSA-AuNPs-based electrochemical DNA biosensor could be adopted for highly reliable and accurate detection of V. cholerae DNA in environmental and clinical samples.
Performance Comparison with Other Reported V. cholerae DNA Biosensors
Based on the data summarised in Table 5, the proposed electrochemical DNA biosensor based on PSA-AuNPs immobilisation material shows an exceptional broad linear quantification range compared to other planar two-dimensional electrodes as the DNA supporters. This clearly demonstrates the advantage of the micro-sized latex particles where the polymeric PSA is capable to intensify the probe binding capacity with a simple loading method via the classical EDC/NHS coupling compared to avidin-biotin technology [24, 26] and ultra-low detection limit in zeptomolar range with reasonable assay time.
Conclusions
This study reports the development of an electrochemical DNA biosensor for the detection of one of the most devastating high-risk V. cholerae patógenos. The PSA-AuNPs-modified DNA biosensor can be used for direct detection of DNA of interest from the extracted DNA without the need of amplification reaction via conventional PCR method, which is commonly used in those previously reported V. cholerae DNA biosensors. In addition, no further dilution of the extracted DNA is needed as the high-capacity AuNPs-doped latex microspheres-based DNA biosensor is highly sensitive for the quantitation of DNA at extremely low level in sub zeptomolar range. Therefore, the electrochemical DNA biosensor is greatly suitable as a surveillance and diagnostic tool to control the epidemic of the fatal intestinal infection.
Nanomateriais
- O potencial para integrar dados visuais com a IoT
- Demonstração de um biossensor flexível à base de grafeno para detecção sensível e rápida de células cancerosas do ovário
- A detecção fotoeletroquímica aprimorada de ácido úrico em nanopartículas de Au Eletrodo de carbono vítreo modificado
- Um biossensor eletroquímico de DNA altamente sensível do nanocompósito de ouro acrílico para a determinação do gênero do peixe aruanã
- Nanoesferas de carbono monodispersas com estrutura porosa hierárquica como material de eletrodo para supercapacitor
- Estabilidade aprimorada de nanopartículas magnéticas de ouro com poli (ácido 4-estirenossulfônico-ácido co-maleico):propriedades ópticas sob medida para detecção de proteínas
- Síntese controlada de BaYF5:Er3 +, Yb3 + com morfologia diferente para o aprimoramento da luminescência de conversão ascendente
- Um Aptasensor Fluorescente à Base de Óxido de Grafeno para a Detecção Ligada de CCRF-CEM
- Composto nanofibroso TPU eletricamente condutivo com alta elasticidade para sensor de deformação flexível
- Objetos impossíveis são parceiros da BASF para impressão 3D composta



