Biossensor de transistor de efeito de campo de grafeno magnético para detecção de DNA de cadeia simples
Resumo
Aqui, um biossensor de transistor de efeito de campo magnético de grafeno foi preparado através da transferência de um filme de grafeno de deposição de vapor químico em um substrato de vidro para produzir um filme de detecção e canal condutor. Ao fixar o éster succinimidílico do ácido 1-pirenobutanóico no filme de grafeno como uma âncora, um aptâmero de sonda foi imobilizado no filme de grafeno a fim de capturar DNA de fita simples complementar magneticamente marcado. Nossos experimentos mostraram que, dentro de um campo magnético periódico, a impedância do biossensor exibia uma oscilação periódica, cuja amplitude estava correlacionada à concentração de DNA complementar. Com base neste princípio, o transistor de efeito de campo de grafeno magnético foi utilizado para detectar DNA de fita simples com limitação de detecção de 1 pM. Os resultados foram racionalizados usando um modelo em que a força magnética faz com que a fita de DNA dobre, resultando assim em nanopérolas magnéticas / modulação de DNA da camada condutiva dupla de transistores de grafeno. Além disso, uma vez que um campo magnético periódico pode ser introduzido para produzir mudanças de impedância periódicas de MGFETs, a integração de amostragem pode ser usada para melhorar a razão sinal-ruído de forma eficiente, aumentando o número de períodos do campo magnético externo. Portanto, um novo biossensor para detecção de DNA com alta sensibilidade foi apresentado neste trabalho. Com base no princípio de detecção, este sistema também pode ser uma ferramenta potencial para detectar outras biomoléculas, células, etc.
Introdução
A detecção de DNA é de grande importância para o estudo da biologia molecular e o diagnóstico de doenças genéticas [1,2,3]. Até o momento, vários biossensores para detecção de DNA foram desenvolvidos, incluindo biossensores fluorescentes [4, 5], biossensores eletroquímicos [6,7,8,9] e biossensores de transistor de efeito de campo (FET) [10,11,12,13 ], tendo este último atraído atenção generalizada devido à sua elevada sensibilidade e especificidade. Kaisti et al. [12] desenvolveram um biossensor FET para detectar DNA de fita simples não marcado usando sondas de ácido nucleico de peptídeo. Kim et al. [13] fabricou um sensor de carga de DNA do tipo FET baseado na tecnologia de semicondutor de óxido de metal complementar padrão.
Devido à sua alta área de superfície específica, alta condutividade elétrica e excelente mobilidade de elétrons, o grafeno foi anunciado como um material ideal para a fabricação de biossensores FET [14,15,16]. Cai et al. [15] desenvolveram um biossensor de grafeno FET (GFET) para detecção ultrassensível de DNA via hibridização de DNA de ácido nucleico de peptídeo. Nosso grupo também propôs um biossensor GFET multicanal para determinar a cinética de ligação e a afinidade de hibridização de DNA e incompatibilidade de base única [16].
Em um GFET convencional, um campo elétrico de eletrodo de porta externa gera uma camada condutiva dupla na interface entre o filme de grafeno e o eletrólito da solução [17,18,19]. Baseado em um modelo cativo de GFETs [16], o eletrodo da porta carrega e descarrega a camada condutiva dupla através do eletrólito, modulando assim a condutividade do GFET. Portanto, a condutividade de um GFET está relacionada à intensidade do campo elétrico externo e à concentração de íons no eletrólito.
Durante a pesquisa, constatou-se que a pesquisa sobre a sensibilidade dos GFETs atingiu o patamar de fM. Por exemplo, Ping et al. [20] e Zheng et al. [21] relataram biossensores GFET convencionais com limite de detecção em nível de fM. No entanto, a literatura acima alcança sensibilidade extremamente alta por detecção de analisador de semicondutor, o que é caro e inconveniente para aplicações práticas. Além disso, os eletrodos Ag / AgCl são comumente usados como eletrodos de porta externa, que são inadequados para a construção de biossensores integrados devido ao seu tamanho e reutilização.
Aqui, um biossensor magnético GFET (MGFET), no qual um campo magnético em vez de um campo elétrico é utilizado para modular a condutividade GFET, foi desenvolvido. O canal condutor foi obtido por meio de um filme de grafeno de deposição química de vapor (CVD) transferido para um substrato de vidro com dois eletrodos de óxido de índio e estanho (ITO). O filme de grafeno foi funcionalizado com ácido 1-pirenobutanóico succinimidil éster (PBASE) para permitir a ligação de uma sonda de aptâmero para capturar e hibridizar com DNA de fita simples (cDNA) magneticamente marcado complementar. Aplicando um campo magnético periódico na parte de trás dos MGFETs, uma impedância elétrica MGFET periódica foi alcançada. Além disso, a flutuação da impedância elétrica dos MGFETs em um campo magnético periódico estava relacionada à concentração de cDNA. Um dispositivo de detecção feito em laboratório correspondente foi construído para detectar a impedância MGFET em tempo real. Uma vez que o campo magnético não está em contato direto com os MGFETs, os MGFETs aqui preparados são mais fáceis de integrar e aplicar do que os biossensores GFET convencionais. A preparação dos MGFETs, a construção do sistema de detecção feito em laboratório e o princípio de detecção foram todos descritos em detalhes neste artigo.
Métodos
Materiais e instrumentos
Um substrato de vidro com eletrodos ITO foi adquirido de Hua Nan Xiang Cheng Ltd. (China). A sonda aptâmero, cDNA e DNA incompatível foram adquiridos de Sangon Biotech Inc. (Shanghai, China). A sequência do aptâmero da sonda era (5′-NH 2 -TGG ACC CCC TCA TAA CGC CTC CTT TTC-FAM-3 ′), a sequência do DNA complementar era (5′-NH 2 -GAA AAG GAG GCG TTA TGA GGG GGT CCA-3 ′), a sequência do DNA completamente incompatível era (5′-NH 2 -TCC CCT TCT TAT GGC CTG TTT TTC AAC-3 ′), e a sequência do DNA incompatível de base única era (5′-NH 2 -GAA AAG GAG TCG TTA TGA GGG GGT CCA-3 ′). PBASE e dimetilsulfóxido (DMSO) foram obtidos da Sigma-Aldrich (Shanghai, China). Nanopérolas magnéticas (MBs) modificadas com grupos carboxila (10 mg / mL) foram obtidas de Xianfeng Nano Material Technology Co., Ltd. (Nanjing, China). Cloridrato de 1-etil-3- (3-dimetilaminopropil) carbodiimida, N-hidroxissuccinimida, dodecilbenzenossulfonato de sódio (SDS) e solução salina tamponada com fosfato de dodecil sulfato de sódio (PBS, P5368-10PAK; pH 7,4) foram adquiridos na Sigma-Aldrich (Shanghai , China).
Um sistema microscópico Raman (SPEX-1403, SPEX) foi utilizado para caracterizar a qualidade do grafeno e também para verificar a funcionalização dos MGFETs. Um fotômetro de fluorescência (LS55, PerkinElmer) foi usado para caracterizar o acoplamento de nanopartículas magnéticas ao cDNA. Um sistema de aquisição de dados feito em laboratório foi usado para registrar a impedância dos MGFETs em tempo real.
Acoplando cDNA a MBs
Depois de uniformemente disperso por ultrassom por 20 min, uma suspensão de 20 μL de MBs modificados com grupos carboxila foi misturada com 200 μL de cloridrato de 1-etil-3- (3-dimetilaminopropil) carbodiimida (2 mg / mL) e 200 μL de N- hidroxisuccinimida (2 mg / mL) por 15 min para obter MBs ativados [22, 23]. Em seguida, 20 μL de solução de cDNA foram adicionados à solução MBs e incubados por 2 h em temperatura ambiente com agitação suave contínua. Um campo magnético foi então introduzido para enriquecer as amostras de cDNA através de MBs. Os conjugados de nanopérolas magnéticas / DNA (MB / cDNA) foram lavados três vezes com PBS e dispersos em PBS para uso futuro.
Fabricação de MGFETs
A preparação de MGFETs é descrita em detalhes abaixo. Primeiramente, um filme de grafeno CVD foi transferido para uma placa de vidro como canal condutor entre os dois eletrodos ITO (Fig. 1a), conforme descrito anteriormente [18, 19]. Em segundo lugar, PBASE (10 mM) dissolvido em DMSO foi injetado nos MGFETs por 12 h em temperatura ambiente e deixou-se reagir completamente com grafeno através de empilhamento π – π (Fig. 1b). Os MGFETs foram então lavados sucessivamente com DMSO e PBS para remover qualquer PBASE que não reagiu. Em terceiro lugar, 2 μM da sonda de aptâmero foram introduzidos nos MGFETs e incubados com PBASE por 4 h à temperatura ambiente, permitindo que a sonda de aptâmero reaja suficientemente com PBASE (Fig. 1c). Os MGFETs foram então lavados respectivamente com SDS a 0,2% três vezes para remover qualquer aptâmero de sonda não ligado.
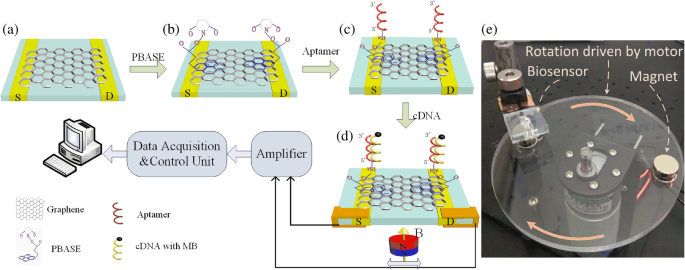
Princípio de funcionalização e detecção dos MGFETs. a Filme de grafeno desenvolvido por deposição química de vapor. b Funcionalização do grafeno por PBASE. c Imobilização do aptâmero da sonda via PBASE. d Hibridação do aptâmero de sonda com cDNA. e Fotografia do dispositivo de detecção
Resultados e discussão
Caracterização de MGFETs
Filme de grafeno produzido pelo método CVD foi transferido em um substrato de vidro como um canal condutor entre dois eletrodos ITO (Fig. 1a). O filme de grafeno transferido foi caracterizado com espectro Raman (Fig. 2). O aparecimento dos três picos característicos do grafeno demonstrou a transferência bem-sucedida do filme de grafeno para o substrato de vidro [24, 25]. A relação de intensidade entre a banda 2D e a banda G (I 2D / I G ) indicou que o grafeno transferido era um filme multicamadas [26]. Além disso, a relação de intensidade entre a banda D e a banda G (I D / I G ) era pequeno, indicando uma densidade de defeito muito baixa.
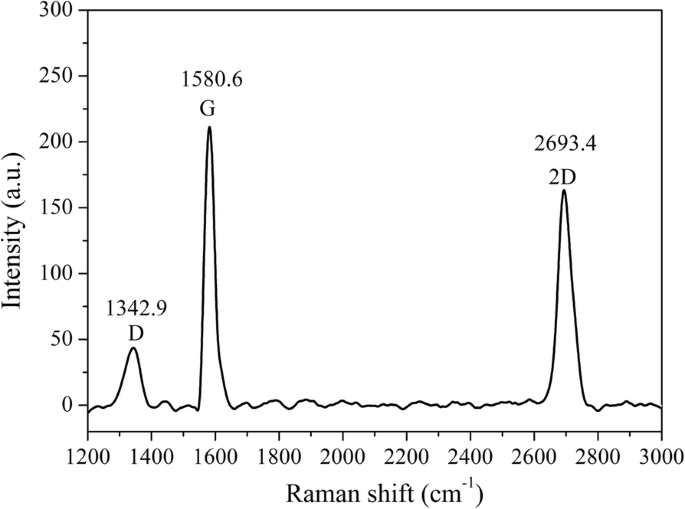
Espectro Raman
Devido à falta de grupos funcionais, as cadeias de aptâmeros eram difíceis de modificar no filme de grafeno CVD. Portanto, com base em seu grupo pirenil aromático, PBASE foi modificado nos filmes de grafeno via empilhamento π – π como um ligante. Na outra extremidade da PBASE, a porção succinimida da PBASE poderia ser acoplada ao 5′-NH 2 Aptâmero de sonda marcada com base na reação de reticulação de N-hidroxissuccinimida (NHS) (Fig. 1c). A fim de avaliar a ligação do aptâmero da sonda no filme de grafeno, a extremidade 3 'do aptâmero da sonda foi marcada usando o fluoróforo FAM (sequência:5'-NH 2 -TGG ACC CCC TCA TAA CGC CTC CTT TTC-FAM-3 ′). Imediatamente após a introdução do aptâmero, a intensidade da fluorescência foi obviamente aumentada, indicando sua modificação bem-sucedida na superfície de grafeno (Fig. 3). O aumento da concentração do aptâmero da sonda levou a um aumento na intensidade de fluorescência, atingindo um valor constante e, portanto, indicando a saturação do aptâmero da sonda nos MGFETs, em aproximadamente 2 μM. Portanto, os experimentos subsequentes foram realizados em uma concentração de aptâmero de sonda de 2 μM.
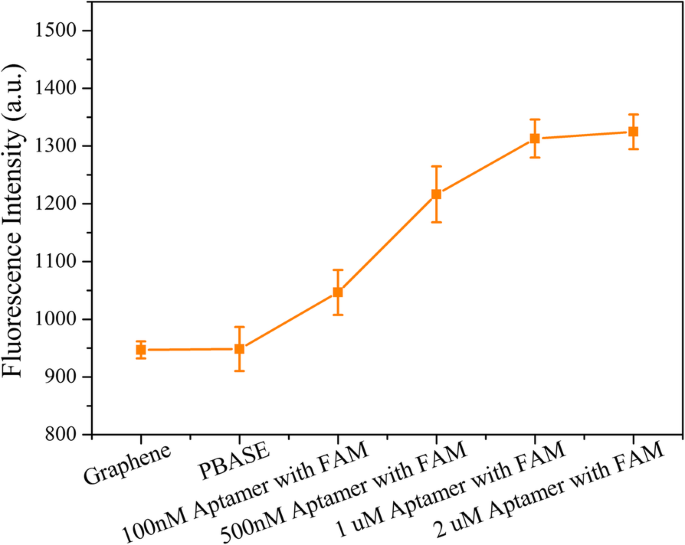
Caracterização da modificação de MGFETs pelo aptâmero de sonda. A barra de erro representa o desvio padrão de 5 análises independentes
Caracterização de MB / cDNA
A morfologia dos conjugados MBs e MB / cDNA foi caracterizada por microscopia eletrônica de transmissão (TEM) (Fig. 4a, b). A distribuição do tamanho de partícula de MBs mostrou um tamanho médio de partícula de aproximadamente 7 nm (Fig. 4c). A fim de garantir sensibilidade e precisão no biossensorio para cDNA, MBs devem ser excessivos para cDNA a fim de capturar cDNA completamente. Os MBs a uma concentração de 4 mg / mL foram ativados para garantir a ligação às amostras de cDNA aqui utilizadas. Através da marcação de cDNA por FAM, a intensidade de fluorescência foi explorada para caracterizar a eficiência de acoplamento e otimizar a concentração de cDNA (Fig. 4d). Na verdade, a intensidade de fluorescência do sobrenadante diminuiu obviamente após a introdução de MBs nas soluções de cDNA, indicando que o cDNA foi capturado e enriquecido pelos MBs. O sucesso da captura de cDNA por MBs foi confirmado pela observação de que, a uma concentração de cDNA de 10 nM, a intensidade de fluorescência do sobrenadante era equivalente à do PBS, indicando que todo o cDNA foi capturado e enriquecido por MBs (Fig. 4d )
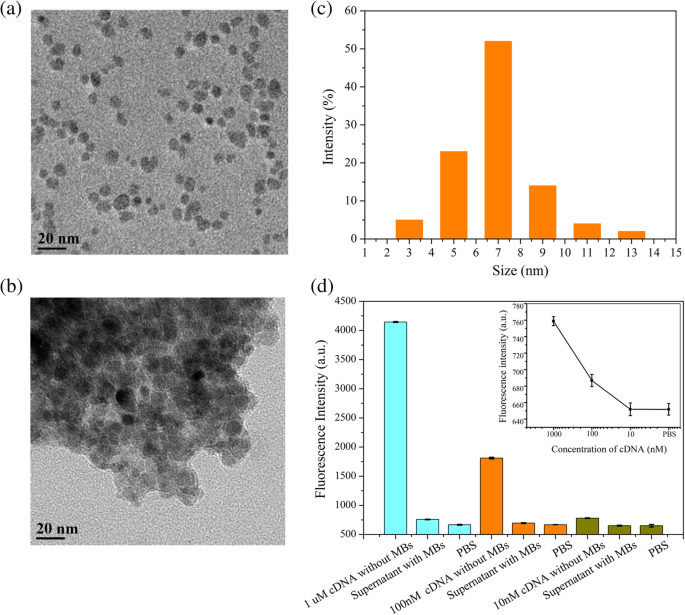
Caracterização do acoplamento MB / cDNA. a TEM de MBs. b TEM de conjugados MB / cDNA. c Distribuição de tamanho de partícula de MBs. d Caracterização do acoplamento MB / cDNA (FAM). A barra de erro representa o desvio padrão de 5 análises independentes
Análise da intensidade do campo magnético
Conjugados MB / cDNA foram adicionados aos MGFETs por 10 min para permitir a hibridização completa de cDNA com o aptâmero de sonda. Uma vez que o aptâmero de sonda não pode acoplar com MBs sem os grupos amino modificados, o excesso de MBs pode ser removido através da lavagem dos MGFETs três vezes com PBS. Portanto, apenas os conjugados MB / cDNA foram deixados nos MGFETs (Fig. 1d). Um ímã permanente foi montado em um motor rotativo para aplicar um campo magnético periódico aos MGFETs (Fig. 1e). Um dispositivo de detecção feito em laboratório foi usado para registrar a flutuação da impedância dos MGFETs.
Uma vez que a impedância dos MGFETs foi modulada por um campo magnético como a porta traseira, a correlação entre a intensidade do campo magnético e a impedância dos MGFETs foi investigada para otimizar os parâmetros de intensidade do campo magnético (Fig. 5). Acredita-se geralmente que a dupla camada condutiva formada entre o grafeno e o eletrólito é modulada pelo campo elétrico externo, modulando assim a condutividade dos GFETs [19, 27, 28]. Nos MGFETs, por meio da força magnética entre os MBs e o campo magnético, a distância entre os conjugados MB / cDNA e o filme de grafeno era controlada mecanicamente, modulando assim a dupla camada condutiva dos MGFETs [29, 30]. A impedância dos biossensores MGFET variou com o aumento da intensidade do campo magnético em três estágios, o que pode ser explicado tomando a cadeia de MB / cDNA como uma haste elástica fina [31]. A primeira etapa ocorreu em uma intensidade de campo magnético inferior a 100 mT neste trabalho. Com base no modelo de bastão fino elástico de cadeias de DNA, porque a força do campo magnético é menor que a força de suporte radial da fita de DNA, a força do campo magnético é difícil de causar a curvatura da fita de DNA; portanto, os MGFETs não são sensíveis ao campo magnético. No segundo estágio, com a força do campo magnético de 100 a 200 mT, a força do campo magnético é suficiente para superar a força de suporte radial da haste fina elástica de DNA, resultando em uma curvatura rápida do MB / cDNA e, em seguida, uma resposta sensível de os MGFETs para o campo magnético. Por fim, no terceiro estágio com intensidade de campo magnético acima de 220 mT, a flexão da haste elástica de DNA atinge seu limite; portanto, os MGFETs não responderão à mudança do campo magnético, resultando em uma impedância estável dos MGFETs, conforme mostrado na Fig. 5b.
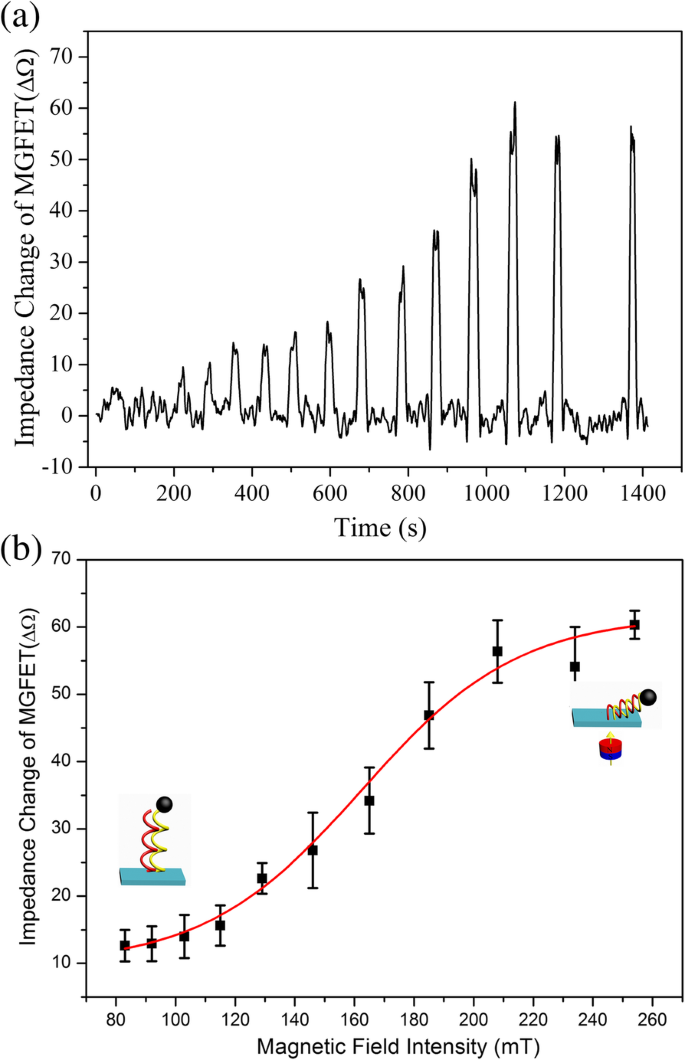
Influência da intensidade do campo magnético na impedância dos MGFETs. a Impedância de MGFETs sob uma intensidade de campo magnético variável no domínio do tempo. b Relação entre impedância de MGFETs e intensidade do campo magnético. A barra de erro representa o desvio padrão de 5 análises independentes
Detecção de cDNA
As alterações na impedância MGFET com concentrações variáveis de conjugado MB / cDNA foram medidas sob uma força de campo magnético fixa de 240 mT para determinar a viabilidade e a sensibilidade para a detecção de cDNA.
A impedância MGFET em cada concentração de cDNA foi registrada em tempo real (Fig. 6a). Quando um ímã permanente foi carregado na parte traseira dos MGFETs, a impedância aumentou rapidamente. Por outro lado, quando um campo magnético periódico foi aplicado, uma mudança periódica na impedância foi observada. Com base nesta periodicidade de impedância, um algoritmo de integração de amostra (SIA) foi usado para aumentar a relação sinal-ruído dos MGFETs. Dado o período sem aplicação de campo magnético foi T 0 e o período com a aplicação do campo magnético foi T M (Fig. 6a), o SIA pode ser descrito com as seguintes etapas:(1) durante T 0 , todos os pontos de dados, produzidos pelo ruído, foram normalizados para zero, (2) os pontos de dados obtidos durante cada T M período foram amostrados e calculada a média em ordem. Após o processamento de SIA ao longo de quatro ciclos, a mudança periódica de impedância em MGFETs foi obtida como mostrado na Fig. 6b. Em teoria, a relação sinal-ruído dos MGFETs poderia ser melhorada efetivamente usando tempos de amostragem suficientemente longos.
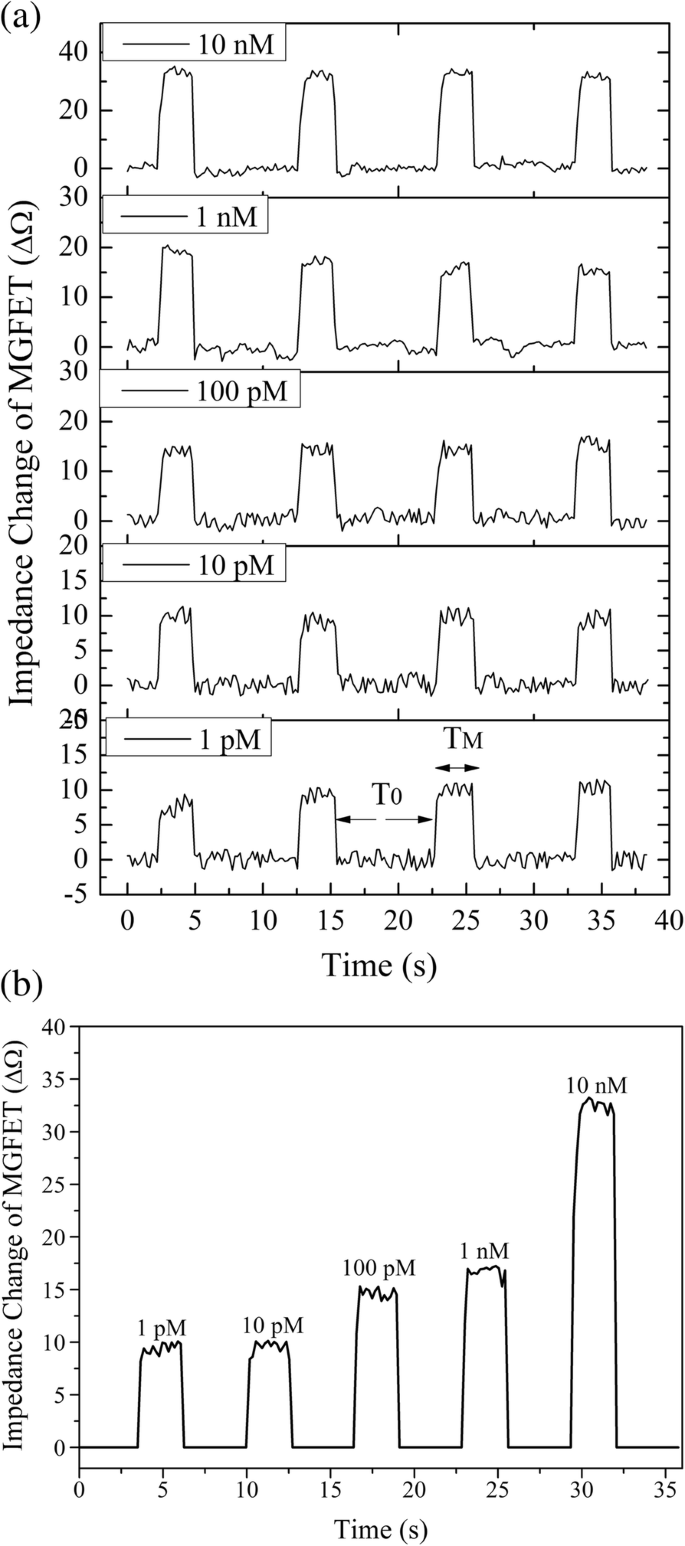
a Domínio de tempo das flutuações de impedância com diferentes concentrações de cDNA. b Alterações de impedância de MGFETs de acordo com a concentração de cDNA
As alterações de impedância em MGFETs tiveram uma correlação positiva com a concentração de cDNA (Fig. 6b). A correlação entre a alteração da impedância dos MGFETs e a concentração de cDNA foi avaliada (Fig. 7). A alta sensibilidade dos biossensores MGFET neste trabalho é baseada principalmente nos dois aspectos a seguir:em primeiro lugar, o movimento mecânico dos conjugados MB / cDNA poderia aumentar o efeito de modulação na camada dupla condutora em comparação com o caso do DNA sozinho e, em segundo lugar, uma vez que um campo magnético periódico pode ser aplicado para produzir mudanças periódicas de impedância de MGFETs, com base no princípio de integração de amostragem, apenas a impedância de MGFET com o campo magnético foi amostrada e integrada para reduzir o ruído. Portanto, a relação sinal-ruído do sistema pode ser bastante otimizada aumentando o número de períodos do campo magnético externo.
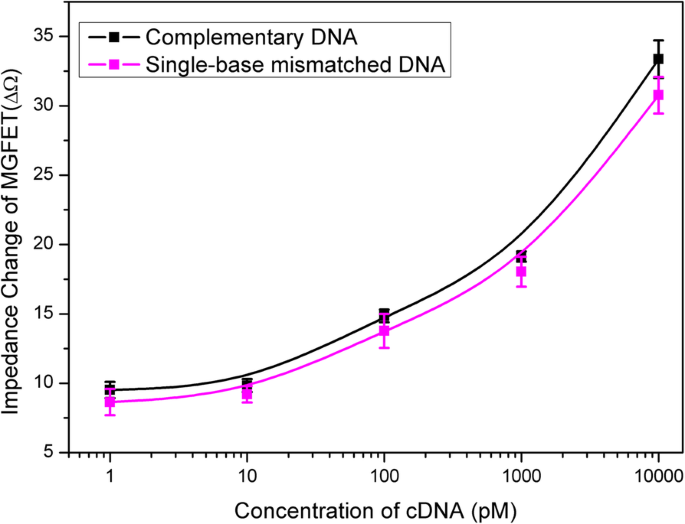
Relação entre impedância de MGFETs e concentração de DNA alvo. A barra de erro representa o desvio padrão de 5 análises independentes
Seletividade dos MGFETs
A especificidade dos MGFETs foi avaliada pela detecção de duas sequências de DNA alvo diferentes, incluindo cadeias de DNA completamente incompatíveis e cadeias de DNA incompatíveis de base única. Semelhante ao procedimento descrito acima, um DNA completamente incompatível (sequência:5′-NH 2 -TCC CCT TCT TAT GGC CTG TTT TTC AAC-3 ′) e DNA de base única incompatível (sequência:5′-NH 2 -GAA AAG GAG TCG TTA TGA GGG GGT CCA-3 ′) foram acoplados a MBs, respectivamente. O MB / DNA incompatível dissolvido em solução de PBS foi adicionado aos biossensores MGFET por 10 min para reagir com o aptâmero suficientemente. Os MGFETs foram lavados com PBS por três vezes para remover o DNA incompatível. Para cadeias de DNA completamente incompatíveis, devido ao conjugado de MB / DNA não poder hibridizar com o aptâmero, quase todos os conjugados de MB / DNA foram removidos. Portanto, a adição de MB / DNA completamente incompatíveis quase não tem efeito sobre a condutividade do grafeno como mostrado na Fig. 8, o que indica uma alta seletividade do biossensor. Além disso, também investigamos a seletividade dos biossensores através de cadeias de DNA incompatíveis de base única, como mostrado na Fig. 7. Pode-se constatar que a alteração da impedância de MGFET com cadeias incompatíveis de base única foi ligeiramente menor do que as fitas complementares e maior do que a fita alvo não complementar em cada concentração. Portanto, a fita incompatível de base única pode ser detectada neste trabalho. Embora o aptâmero e as cadeias de DNA complementares sejam todos produtos comerciais que determinam principalmente a seletividade dos biossensores, os MGFETs e seu sistema de detecção também têm contribuído para a alta sensibilidade para detecção de DNA.
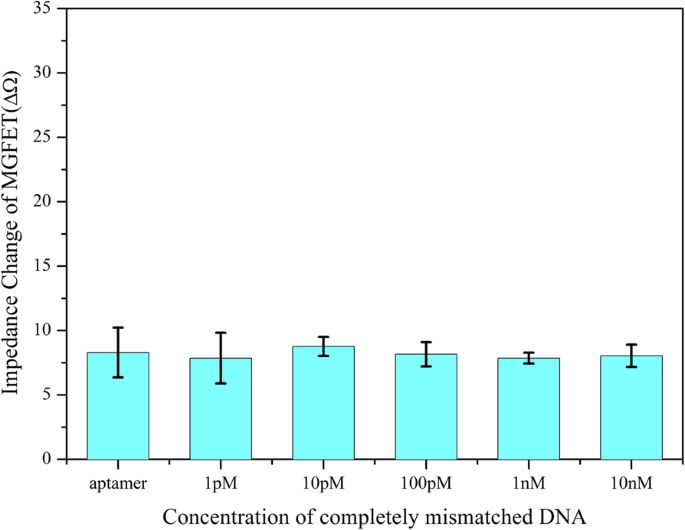
Relação entre impedância de MGFETs e concentração de DNA completamente incompatível. A barra de erro representa o desvio padrão de 5 análises independentes
Conclusões
Aqui, um biossensor MGFET baseado em grafeno e nanopartículas magnéticas foi apresentado para detectar cDNA. Nos MGFETs, nanopartículas magnéticas foram modificadas no final da sequência de cDNA. Através da força magnética entre os MBs e o campo magnético, a distância entre os conjugados MB / cDNA e o filme de grafeno foi controlada mecanicamente, modulando assim a dupla camada condutiva dos MGFETs. Além disso, também podemos concluir que, para uma fita de DNA particular, a impedância dos MGFETs refletirá o estresse da fita de DNA, que por sua vez reflete a curvatura da fita de DNA (inserção, Fig. 5b). Assim, os presentes MGFETs têm potencial para serem utilizados no estudo de parâmetros mecânicos de cadeias de DNA. Portanto, os MGFETs podem não apenas funcionar como um biossensor para a detecção de cDNA, mas também podem detectar potencialmente os parâmetros mecânicos das cadeias de DNA.
Disponibilidade de dados e materiais
Todos os dados gerados ou analisados durante este estudo estão incluídos no artigo.
Abreviações
- cDNA:
-
DNA complementar de fita simples magneticamente marcado
- CVD:
-
Deposição de vapor químico
- DMSO:
-
Dimetilsulfóxido
- FET:
-
Transistor de efeito de campo
- GFET:
-
Transistor de efeito de campo de grafeno
- MBs:
-
Nanobeads magnéticas
- MGFET:
-
Transistor de efeito de campo magnético de grafeno
- NHS:
-
N-Hidroxissuccinimida
- PBASE:
-
Éster succinimidílico de ácido 1-pirenobutanóico
- PBS:
-
Solução salina tamponada com fosfato de dodecil sulfato de sódio
- SDS:
-
Sulfonato de Dodecilbenzeno de Sódio
- SIA:
-
Algoritmo de integração de amostra
- TEM:
-
Microscopia eletrônica de transmissão
- ITO:
-
Óxido de índio estanho
Nanomateriais
- Blog:Detecção de genes através de microarray
- Nanodiamonds para sensores magnéticos
- Demonstração de um biossensor flexível à base de grafeno para detecção sensível e rápida de células cancerosas do ovário
- Compostos de grafeno e polímero para aplicações de supercapacitor:uma revisão
- Nanodots de carbono como nanosensores de modo duplo para detecção seletiva de peróxido de hidrogênio
- Um biossensor eletroquímico de DNA altamente sensível do nanocompósito de ouro acrílico para a determinação do gênero do peixe aruanã
- Cristalização dependente da temperatura de nanoflocos MoS2 em nanofolhas de grafeno para eletrocatálise
- Biossensor ultrassensível para a detecção de DNA de Vibrio cholerae com nanoesferas de composto de ácido poliestireno-co-acrílico
- Método sensível para detectar defeitos no transistor
- Sensor de estado sólido para detecção e caracterização de campos elétricos



