O Estabelecimento e os Estudos de Aplicação em Indicador de pH de Lisossomo Preciso Baseado em Nanopartículas Auto-Decomponíveis
Resumo
O pH ácido dos lisossomos está intimamente relacionado à autofagia; assim, bem conhecidos dos lisossomos precisos, as mudanças de pH darão mais informações sobre o processo e o status da autofagia. Até agora, no entanto, apenas mudanças de pH em uma faixa relativamente ampla puderam ser indicadas, a detecção exata do pH dos lisossomos nunca chegou. Em nosso estudo, estabelecemos um indicador de pH endo / lisossoma com base no SiO auto-decomposto 2 sistema de nanopartículas com parâmetros de síntese específicos. O azul de metileno concentrado central (MB) nas nanopartículas estruturais ocas apresentou liberação sensível em função dos valores de pH 4,0-4,8, que é exatamente a faixa de pH dos lisossomos. A correlação linear dos valores de densidade óptica (DO) e os valores de pH foi construída, a qual tem sido usada para a detecção do pH dos lisossomas em 6 linhas celulares diferentes. Além disso, por este sistema, conseguimos detectar com precisão as mudanças médias de pH dos lisossomos antes e após a endocitose NP de silício mesoporoso preto (BPSi), esclarecendo o mecanismo de terminação da autofagia após a endocitose de BPSi. Assim, o indicador de pH luminal baseado em nanopartículas auto-decomponíveis pode fornecer uma nova metodologia e estratégia para conhecer melhor o pH do lisossoma e, então, indicar mais detalhes sobre o processo de autofagia ou outra sinalização importante sobre metabolismos.
Introdução
Os lisossomos servem como destino final para macromoléculas, onde essas macromoléculas são degradadas por enzimas hidrolíticas ativadas por baixo pH [1]. O pH ácido dos lisossomas mantido pela H + −ATPase vacuolar (v-ATPase) [2] que bombeia prótons do citoplasma para o lúmen lisossomal era para manter a atividade de ~ 60 tipos de enzimas hidrolíticas [3]. Além disso, relatórios recentes da literatura revelaram que o pH ácido dos lisossomos está intimamente relacionado à autofagia [4], de modo que as mudanças precisas do pH do lisossoma bem conhecidas fornecerão mais informações sobre o processo e o status da autofagia. Com base em nossos estudos e revisão da literatura, a endocitose de nanopartículas carregadas com amina positiva provavelmente aumentará a mudança de pH em endo / lisossomas, como nanopartículas decoradas com PEG de amina primária e secundária ou alguma decoração hidrofílica na superfície da partícula [5, 6].
O aumento de pH induzido pela endocitose de nanopartículas de amina aumentará dramaticamente na localização nuclear do fator de transcrição EB (TFEB) [7], resulta não apenas na suprarregulação transcricional da via, mas também causa disfunção lisossomal, resultando em bloqueio do fluxo autofágico [ 7,8,9]. Como o TFEB regula a autofagia e, consequentemente, sua superexpressão leva a um aumento significativo na produção de autofagossomo em células em cultura.
Assim, para prever o processo de autofagia e os detalhes da autofagia, o pH preciso do lisossoma e sua medição de alteração são muito importantes. Até agora, a partir do valor de pH do endo / lisossoma indicando revisões da literatura [10] e os produtos comerciais para detectar os valores de pH do endo / lisossoma, apenas mudanças de pH em uma faixa relativamente ampla poderiam ser indicadas, e a detecção exata do pH do lisossoma nunca chegou . Assim, para conhecer os detalhes de percepção da autofagia, o estabelecimento de um método preciso de detecção de alteração do pH luminal é uma abordagem importante.
Com base em nossas experiências anteriores em SiO auto-decomposto 2 nanopartículas, neste estudo, estabelecemos um indicador de pH preciso que poderia realizar a detecção de alterações de pH luminais. SiO 2 nanopartículas têm boas vantagens em tamanho ajustável e biocompatibilidade [11]. Ao definir parâmetros de síntese específicos, o SiO auto-decomposto estabelecido 2 O indicador de pH pode liberar com sensibilidade a carga útil de azul de metileno (MB) em pH 4,0–4,8, que é exatamente a faixa de pH dos lisossomos. Além disso, a liberação de MB apresentou correlação linear com a variação do valor de pH (esquema 1). Em seguida, testamos a viabilidade do indicador de pH em níveis celulares, introduzindo 6 linhas celulares diferentes, conseguindo determinar as mudanças médias de pH dos lisossomas antes e depois da endocitose de NPs de silício mesoporoso preto (BPSi), esclarecendo o mecanismo de terminação da autofagia após BPSi endocitose. Assim, o indicador de pH luminal baseado em nanopartículas auto-decomponíveis pode fornecer uma nova metodologia e estratégia para conhecer melhor o pH do lisossoma e, em seguida, indicar mais detalhes sobre o processo de autofagia ou outras sinalizações importantes sobre o metabolismo.
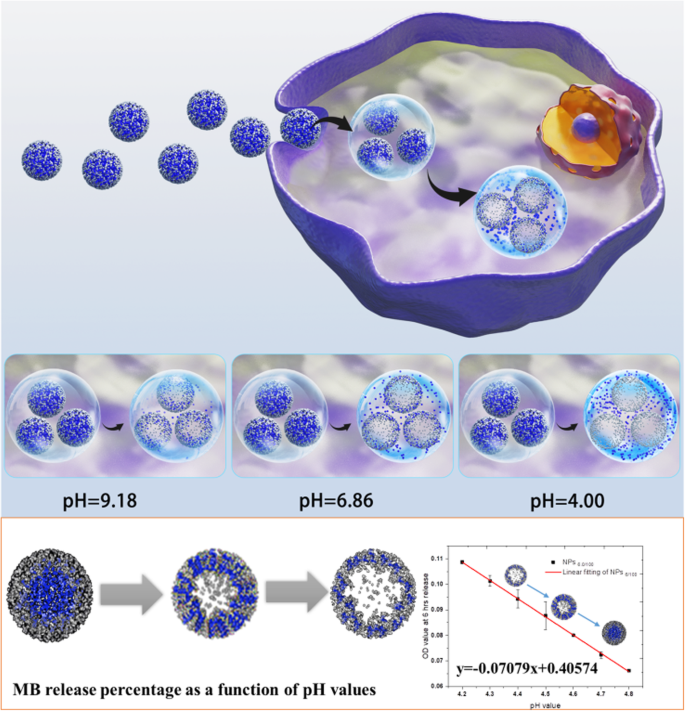
Ilustração esquemática de MB @ SiO 2 medição do pH do lisossoma em células vivas
Materiais e métodos
Seção de material
Siliceto de sódio (NaSi) e bolachas de Si (diâmetro 20 cm, p + (100), 0,01–0,02 Ω cm) foram fornecidos por SiGNa Chemistry Inc. e Ocmetic Inc., respectivamente. Brometo de amônio (NH 4 Br, 99%), brometo de sódio (NaBr, 99%), tolueno (anidro, 99,8%), ácido clorídrico (HCl, 37%), MB e ortossilicato de tetraetila (TEOS) foram adquiridos na Sigma-Aldrich. 0,5 kDa metoxi-PEG-silano e 2 kDa metoxi-PEG-silano foram adquiridos a Fluorochem Ltd. e Laysan Bio Inc. separadamente. O meio RPMI 1640 foi fornecido pela Life Technologies. O soro fetal bovino (FBS) foi comprado da tecnologia biológica TianHang. Bicarbonato de sódio, sulfato de estreptomicina, penicilina G, HEPES, solução de lisozima, CellLight Early-endosomes-GFP, LysoTracker ™ Red DND-99, Pierce® BCA Protein Assay Kit, quimiluminescência aprimorada, conjugado de transferrina vermelha de pHrodo ™, solução de imagem de células vivas, e O reagente Trizol foi comprado da Thermo Fisher Scientific. Etanol e água-amônia foram fornecidos pelo Sinopharm. O kit de síntese de primeira cadeia de cDNA BioRT Master HiSensi foi comprado da Hangzhou Bioer Technology Co., Ltd. O lisado de RIPA foi comprado da Heart Biological Technology Co., Ltd. P62, TFEB e o anticorpo β-actina foram comprados do Proteintech Group , Inc. O anticorpo LC 3B foi fornecido pela Abcam. 2- (4-Piridil) -5 - ((4- (2-dimetilaminoetil-laminocarbamoil) metoxi) fenil) oxazol (PDMPO) foi fornecido pela Yeasen Biotech Co., Ltd.
Objetivo, projeto e cenário do estudo
O objetivo deste estudo é (1) explorar a influência das nanopartículas de BPSi na autofagia em células HepG2, (2) descobrir o mecanismo subjacente das alterações do valor de pH do lisossoma que afetam a autofagia, (3) estabelecer um indicador de pH do lisossoma preciso que possa medir o pH dos lisossomos com precisão e, finalmente, (4) indicam a influência da flutuação do pH na autofagia. A fim de realizar o objetivo do estudo acima, empregamos o experimento de sequenciamento do transcriptoma para explorar as mudanças no gene do transcriptoma em células HepG2 após a alimentação de nanopartículas de BPSi e verificamos por RT-qPCR e experimentos Western. Corantes fluorescentes, como PDMPO, foram usados para medir a mudança do pH lisossomal em células HepG2 após a alimentação com BPSi. Para medir o pH dos lisossomos com precisão, desenvolvemos MB @ SiO 2 nanopartículas com 10 parâmetros e testou as caracterizações desses 10 tipos de nanopartículas por meio de experimentos como DLS e HR-TEM. A eficiência de carregamento do MB e o estudo da cinética de liberação de sistemas de nanopartículas auto-decomponíveis da série 10 foram testados em diferentes soluções de pH e células HepG2. Para verificar a localização intracelular das nanopartículas após entrar na célula, fizemos o experimento com a célula TEM e a microscopia confocal de células vivas. Finalmente, medimos as mudanças de pH lisossomal em 6 tipos de células após a alimentação com BPSi para verificar a universalidade de MB @ SiO 2 nanopartículas para medir as alterações do pH lisossomal.
Síntese de nanopartículas de BPSi
As nanopartículas de BPSi foram preparadas por nosso método anterior [12] e fornecidas por nosso colaborador (Wujun Xu, Departamento de Física Aplicada, University of Eastern Finland). A preparação de BPSi, NaSi, sal de amônio e NaBr (NaSi:NH 4 Br:NaBr de 1:4:4, w / w / w) foi moído em um porta-luvas com atmosfera de Ar. Eles foram autorizados a reagir em um forno tubular sob um N 2 atmosfera a 240 ° C durante 5 h (Eq. 1). Depois de serem resfriadas à temperatura ambiente, as micropartículas obtidas foram purificadas por enxágue com soluções de HCl 0,5 M e HF 1,0 M separadamente. As micropartículas foram moídas em etanol a 1000 rpm por 15 min, e as nanopartículas de BPSi com o diâmetro desejado foram coletadas por meio do ajuste da velocidade de centrifugação.
$$ \ mathrm {NaSi} + {\ mathrm {NH}} _ 4 \ mathrm {Br} \ to \ mathrm {NaBr} + {\ mathrm {NH}} _ 3+ \ mathrm {Si} / \ mathrm {H} + { \ mathrm {H}} _ 2 $$ (1)
Por meio do experimento de espalhamento dinâmico de luz, a distribuição do diâmetro e a carga superficial da nanopartícula foram estudadas. Todos os NPs foram dispersos no meio após a esterilização por ultra-som leve (5 s para torná-los uniformemente dispersos na solução, limpador ultra-sônico SB-5200DT, Ningbo Scientz Biotechnology Co., Ltd.) logo antes de sua introdução nas células.
Estabelecimento do sistema de nanopartículas auto-decomponíveis da série 10
As 10 nanopartículas auto-decomponíveis da série foram sintetizadas pelas metodologias que relatamos antes [13,14,15,16], com parâmetros modificados. Em um procedimento típico, uma certa quantidade de MB foi primeiramente adicionada a uma mistura de etanol (75 mL) com solução de amônia-água (25%, 3,4 mL), depois que certa quantidade de TEOS foi adicionada. A série auto-decomponível MB @ SiO 2 Os NPs foram obtidos após agitação por 24 he lavados 3 vezes antes de serem secos. As quantidades de MB e TEOS adicionadas nos protocolos foram conforme descrito na Tabela 1. O significado de 1.0 / 100 nas NPs 1.0 / 100 representou o inventário de MB e TEOS quando sintetizamos as nanopartículas, com 1,0 mg de MB e 100 μL de TEOS. E os significados de 1,5 / 100 nos NPs 1,5 / 100 e outros são consistentes com 1.0 / 100.
Cultura de células
A fim de testar a eficiência e universalidade do indicador de pH baseado em nanopartículas auto-decomponíveis, tentamos testá-lo em linhas de células cancerosas derivadas de tumor específicas. Assim, selecionamos as linhagens celulares de câncer de fígado, pulmão, câncer de cólon e melanocitoma como objetos de pesquisa. As linhas celulares de células de câncer de cólon humano HCT116, HCT8 e HCT15; células de câncer de fígado humano HepG-2; células de câncer de pulmão humano A549; e células de melanoma de camundongo B16 foram mantidas em meio RPMI 1640 (Life Technologies) suplementado com 10% de FBS inativado por calor, 2,0 g / L de bicarbonato de sódio, 0,1 g / L de sulfato de estreptomicina, 0,06 g / L de penicilina G e 5,958 g / L HEPES. As células foram mantidas em uma incubadora de cultura de células padrão a 37 ° C em uma atmosfera umidificada com 5% de CO 2 .
Caracterizações de sistemas de nanopartículas auto-decomponíveis de 10 séries
A morfologia de todas as nanopartículas da série foi caracterizada por HR-TEM com modo STEM, e o mapeamento de Si foi estudado por mapeamento de elemento EDS. A análise da distribuição do tamanho das nanopartículas foi realizada pelo software ImageJ calculando os diâmetros das nanopartículas em imagens STEM selecionadas aleatoriamente. O potencial zeta das nanopartículas e o índice de polidispersidade (PDI) foram medidos pelo estudo de espalhamento dinâmico de luz (DLS), em buffers série com valores de pH específicos. Os dados foram analisados pelo SPSS15.0 e os resultados estatísticos foram apresentados como média ± D.P.
O estudo de eficiência de carregamento e cinética de liberação de MB de sistemas de nanopartículas auto-decomponíveis da série 10
A fim de estudar a eficiência de carregamento de MB e a cinética de liberação, a curva padrão de MB em concentrações em série foi estabelecida pela primeira vez. A absorção do MB foi realizada por espectro UV-Vis com absorbância em 660 nm, que é o λ máx. do monômero MB. A eficiência de carregamento de MB foi calculada pela equação abaixo, eficiência de carregamento de MB (%) =a quantidade de MB encapsulado / (quantidade total de entrada de MB).
A liberação de MB de nanopartículas da série 10 foi estudada em água pura e tampões de pH com diferentes valores de pH (pH 4,0, pH 6,86 e pH 9,18) e solução de lisozima (Thermo Scientific ™ # 90082). Além disso, a cinética de liberação de MB após uma duração específica em diferentes tampões de pH também foi investigada. Os valores de DO em 660 nm e as porcentagens de liberação de MB em função do tempo foram então estudados.
Em mais detalhes, os estudos de liberação de MB foram realizados pelos protocolos abaixo; dissolveu as nanopartículas da série 10 em 15 mL de tampão padrão de pH 4,0, 6,86 e 9,18 com solução de lisossoma, respectivamente; e realizou a liberação de MB em um misturador Hula a 37 ° C. Durante os 15 dias seguintes, 1 mL de cada amostra foi coletado e centrifugado a 12.000 rpm por 10 min. O sobrenadante e o precipitado foram medidos para seus espectros de absorção em 200-800 nm.
Além disso, a liberação de MB em tampões de pH precisos com solução de lisozima na faixa de pH de 4,1 a 5,5 também foi investigada com os mesmos protocolos acima. As durações de tempo específicas (6 h, 12 h e 24 h) foram colocadas como pontos de tempo de observação. As absorções em 660 nm foram registradas em cada amostra. A relação linear de absorção de cada solução de pH e a soma dos quadrados dos resíduos foram contados em cada ponto de tempo, respectivamente.
Para detectar os perfis de liberação de MB nas células, as células HepG-2 foram cultivadas em um 2 de 75 cm frasco de cultura e alimentado com NPs (300 μg / mL) quando as células proliferaram em 70% do frasco de cultura. A cada 30 minutos depois, as células foram coletadas. As células foram repetidamente congeladas e descongeladas para liberar completamente o MB nas células. Os lisados celulares foram centrifugados a 12000 r / min durante 10 min. O sobrenadante foi obtido e medido sua absorbância a 660 nm para calcular a quantidade total de MB liberado. Neste estudo, as células HepG2 foram escolhidas como objetos de pesquisa, devido à sua rápida proliferação celular que poderia minimizar a variância entre os 10 grupos testados.
Colocalização celular das nanopartículas da série 10 e o desempenho de liberação em 6 linhas celulares diferentes
A célula TEM foi empregada para estudar a co-localização da nanopartícula nos endo / lisossomas seguindo os protocolos padrão da célula TEM. As células foram semeadas na intensidade de 1 × 10 6 células / frasco e incubadas por 24h, permitindo a fixação das células. Dez séries de nanopartículas em meio com a mesma concentração (100 μg / mL) foram incubadas com as células por mais 12 e 24 h, respectivamente. As células foram então lavadas por PBS por 3 vezes para remover o excesso de nanopartículas e, em seguida, fixadas em solução de glutaraldeído a 2,5% por mais de 1 dia. As células fixadas foram então lavadas e coradas por tetróxido de ósmio, 1% em água desionizada por 1 h, seguido por lavagem com PBS por 3 vezes e água DI por 2 vezes. O protocolo TEM de células clássicas [17, 18] foi realizado a seguir, e seções com uma espessura de 90 nm foram coletadas para observação TEM. A liberação de MB em função dos valores de pH foi estudada em 6 linhas de células com ambos NPs 6/100 e NPs 7.5 / 80 . Além disso, os valores de OD da liberação do MB e as porcentagens de liberação do MB foram registrados para análise de dados.
Investigação de captação intracelular de MB @ SiO 2 Nanopartículas
Microscopia confocal de células vivas foi usada para avaliar a absorção celular e o destino intracelular do MB @ SiO 2 nanopartículas. Os endossomos iniciais das células HepG-2 foram corados (CellLight Early-endosomes-GFP, BacMam 2.0 ThermoFisher Scientific C10586, com excitação / emissão ~ 488/510 nm) por 16 h. E então as células foram incubadas com NPs 6/100 (Excitação / emissão MB:640 / 650-700 nm) na concentração de nanopartículas de 100 μg / mL em intervalos de tempo específicos (2 h, 2,5 h, 3 h, 5 h e 6 h). Antes de as imagens serem obtidas, o lysotracker foi corado com LysoTracker ™ Red DND-99 (Thermo Fisher Scientific L7528, excitação / emissão:577/590 nm) por 40 min. Depois disso, remova a solução de coloração e lave as células 2-3 vezes em PBS. As imagens foram tiradas usando um microscópio confocal Nikon A1R.
Sequenciamento do transcriptoma para avaliar a mudança da expressão gênica após alimentação com BPSi
A extração de RNA total do grupo de controle e do grupo tratado com BPSi foi realizada usando o reagente Trizol seguindo os procedimentos operacionais padrão. A qualidade da amostra de RNA total inicial para o experimento de sequenciamento foi detectada usando um espectrofotômetro NanoDrop ND-2000. O RNA total que passou no controle de qualidade foi usado em experimentos de sequenciamento subsequentes. Uma comparação da expressão do gene foi realizada por sequenciamento de próxima geração. Todos os programas de sequenciamento foram realizados pela BGI-Shenzhen Corporation (Shenzhen, China) usando a plataforma BGISEQ-500. Os dados brutos obtidos por sequenciamento são realizados no controle de qualidade para determinar se os dados de sequenciamento são adequados para análise subsequente. Se aprovado, execute uma análise quantitativa de genes com base nos níveis de expressão gênica e execute uma análise de enriquecimento significativo de funções de ontologia gênica (GO) nos genes diferencialmente expressos entre as amostras selecionadas.
Ensaio de reação em cadeia da polimerase quantitativa da transcrição reversa (RT-qPCR) para confirmar a ativação da rede de genes TFEB-CLEAR
O RNA total foi extraído das células HepG-2 cultivadas dos grupos controle e tratados com BPSi usando o reagente Trizol e transcrito reversamente para cDNA usando o kit de síntese de primeira cadeia de cDNA BioRT Master HiSensi (Hangzhou Bioer Technology Co., Ltd .) com primers aleatórios. O cDNA foi usado para amplificar a rede de genes TFEB-CLEAR por PCR quantitativo com o sistema de PCR em tempo real Applied Biosystems ™ 7500 (Applied Biosystems, Life Technologies, Carlsbad, CA) com actina como controle de referência. Os primers usados para RT-PCR quantitativo foram listados na Tabela S4.
Ensaio de Western Blot para confirmar que a autofagia está ativada após a alimentação com BPSi
Proteínas celulares do grupo de controle e grupos tratados com BPSi de concentração diferente foram extraídos por lisado de RIPA (Heart Biological Technology Co., Ltd.). O inibidor de protease foi adicionado ao lisado de RIPA e pré-resfriado em gelo. Lavou as células 3 vezes por PBS pré-resfriado. Despejou o líquido completamente e colocou o prato no gelo por 2 min. Quatrocentos microlitros de lisado de RIPA foram adicionados à superfície de todo o prato, pipetados várias vezes com uma pipeta e incubados em gelo por 30 min, durante os quais o prato foi agitado várias vezes para lisar completamente as células. O fluido celular lisado foi transferido para tubos Eppendorf de 1 ml e centrifugado a 13.000 rpm durante 10 min, 4 ° C. O sobrenadante obtido foi fervido em água por 10 min e colocado a - 20 ° C para uso posterior. A concentração de proteína foi quantificada usando o Pierce® BCA Protein Assay Kit (Thermo Scientific).
Extratos celulares contendo 25 μg de proteína total foram diretamente submetidos a SDS-PAGE e transferidos. As membranas foram bloqueadas com 5% de leite desnatado e sondadas com anticorpos primários que reconhecem P62 (Proteintech # 18420–1-AP), TFEB (Proteintech # 13372–1-AP), LC 3B (Abcam # ab192890), e β-actina (Proteintech # 20536–1-AP). Os anticorpos secundários foram escolhidos de acordo com a espécie de origem dos anticorpos primários e detectados por quimioluminescência intensificada (Pierce) ou usando o Bio-Rad ChemiDoc XRS + Gel Imaging System (Bio-Rad, EUA). A intensidade da banda normalizada de P62, TFEB e LC 3B em relação à β-actina foi quantificada por densitometria usando o software ImageJ nos grupos BPSi, e os dados são a média ± D.P. de três experimentos independentes.
Medição do pH da célula por PDMPO e conjugado de transferrina vermelha pHrodo ™ por microscopia confocal
Estudo PDMPO
1 × 10 5 As células HepG-2 foram cultivadas em placas confocais estéreis durante a noite e as nanopartículas de BPSi foram alimentadas com uma concentração de 100 μg / mL. No dia seguinte, antes da coloração de imunofluorescência, as lâminas foram lavadas três vezes com solução salina tamponada com fosfato 0,01 M (PBS), pH 7,4, em seguida, adicionado 1 μM de corante PDMPO (Ex / Em =329/440). Após lavagem com PBS por três vezes, as células foram incubadas com meio de cultura RPMI-1640 fresco e observadas em um microscópio de fluorescência (Nikon A1R, Japão) com uma câmera CCD e tirar fotos em 5 min, e a proporção da intensidade de fluorescência azul e verde nos lisossomos foi então calculado de acordo com o procedimento de Chen et al. [19].
Estudo de conjugado de transferrina vermelha pHrodo ™
As células HepG-2 foram semeadas em placas confocais da mesma forma para fixação das células por 24 h, depois mantidas as placas em gelo por 10 min. Células lavadas com solução fria de imagem de células vivas contendo glicose 20 mM e BSA a 1%. Adicionado conjugado de transferrina vermelha pHrodo ™ (Ex / Em =560/585 nm) a 25 μg / mL em solução de imagem de célula viva e incubar a 37 ° C por 20 min, em seguida, as células lavadas em solução de imagem de célula viva. A observação também foi realizada por microscopia confocal. A análise quantitativa da intensidade das imagens microscópicas foi realizada pelo software ImageJ.
Detecção de pH lisossomal celular
As células A549, HepG-2, HCT8, HCT15, HCT116 e B16 foram cultivadas em um 2 de 75 cm frasco de cultura e alimentado com NPs quando as células proliferaram em 70% do frasco de cultura, e 6 h depois, as células foram coletadas. As células foram repetidamente congeladas e descongeladas para liberar completamente o MB nas células. Os lisados celulares foram centrifugados a 12000 r / min durante 10 min. O sobrenadante foi obtido e medido sua absorbância a 660 nm para calcular a quantidade total de MB liberado. A absorbância dos NPs 6.0 / 100 em um pH padrão foi comparado para obter o valor de pH de cada célula.
Análise estatística
A análise estatística foi feita com o software SPSS15.0 usando uma análise de variância (ANOVA) de dois fatores para grupos independentes e usando o método Tukey HSD para o teste de comparações múltiplas. A significância estatística foi baseada em um valor de P <0,05.
Resultados e discussão
Em primeiro lugar, detectamos a expressão diferencial do gene quando as células alimentadas com nanopartículas de sílica porosa preta funcional de PEG dupla (BPSi NPs) (oferecido por nosso laboratório colaborador [12]). A mudança do potencial zeta de -18,5 para + 2,8 mV também indicou que a PEGuilação dupla de superfície foi bem-sucedida (Fig. S1a). O diâmetro médio das nanopartículas de BPSi foi de 156 nm (Fig. S1b). Com base no mapa de calor de agrupamento de expressão diferencial de genes (Fig. S2), selecionamos expressões de genes diferenciais de mais de 2 vezes para uma investigação mais aprofundada. Go e KEGG foram introduzidos para analisar os genes diferenciais. A partir dos mapas de bolhas de enriquecimento Go (Fig. 1a), os genes metabólicos e associados aos lisossomas, incluindo montagem de fagolisossomos, fagocitose e processo metabólico xenobiótico, foram selecionados para análise posterior. Notavelmente, as expressões gênicas associadas a TFEB-CLEAR [17] aumentaram significativamente. Os resultados de RT-PCR (Fig. 1b) também verificaram os resultados de sequenciamento de genes, os genes em TFEB - a expressão lisossomal coordenada e a via de regulação (CLEAR) aumentaram significativamente, como CTSD, CTSF, TFEB, MFN1, LAMP2 e TPP1 . Esses genes foram marcados na via do lisossoma, como mostrado na Fig. S3. Sua expressão é maior do que a do grupo de controle e tem significância estatística ( P Controle BPSi VS <0,05). E o TFEB regula positivamente a expressão de genes lisossomais, controla a população de lisossomas e promove a degradação celular de substratos lisossomais.
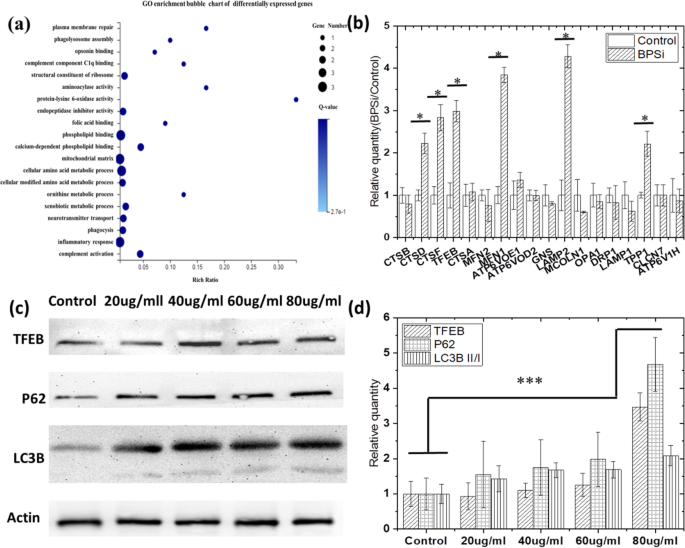
A autofagia é ativada nas células HepG-2 após a alimentação com BPSi. a Mapa de bolhas de enriquecimento GO dos genes diferencialmente expressos encontrados por sequenciamento do transcriptoma. b Verificação de alterações gênicas na rede gênica TFEB-CLEAR após BPSi tratado por experimentos RT-qPCT. c Expressão da proteína P62, TFEB, LC3B II / I após tratamento com BPSi. d Intensidade de cinza de banda normalizada da proteína P62, TFEB, LC3B II / I em grupos tratados com BPSi de acordo com o grupo de controle. Os dados foram apresentados como média ± D.P.
Além disso, o TFEB regula a autofagia, e sua superexpressão leva a um aumento significativo na produção de autofagossomo em células em cultura, devido ao fato de que a principal função do gene TFEB é induzir a biossíntese de lisossoma e promover a ocorrência de autofagia [20]. A análise de Western blot também foi empregada para testificar se a autofagia aconteceu quando as células alimentadas com NPs de BPSi. O objetivo do teste Western foi confirmar ainda mais que a expressão de TFEB foi aumentada e a ocorrência de autofagia celular após a alimentação com BPSi. As proteínas LC3B e P62 são marcadores de autofagia. Quando ocorre autofagia, a expressão da proteína associada a microtúbulos 1A / 1B-cadeia leve 3B (LC3B) II / I aumenta. O p62 é um receptor de vesículas que serão degradadas pela autofagia e também é um receptor de agregados de proteína ubiquitinados a serem depurados, e sua expressão diminui quando ocorre autofagia. Portanto, medimos a expressão dessas proteínas no experimento ocidental.
A partir dos resultados de Western blot mostrados na Fig. 1c, d, TFEB ( P 80 μg / ml de controle VS =0,000008), LC3B II / I ( P 80 μg / ml de controle VS =0,000297), e p62 ( P 80 μg / ml de controle VS =0,000016) proteínas todas significativamente reguladas positivamente. Como a regulação positiva das proteínas TFEB e LC3B II / I indica a ativação da autofagia [18], suspeitamos que a endocitose do BPSi promove a ocorrência de autofagia. No entanto, a proteína p62 deve ser regulada para baixo durante o processo de autofagia, devido à natureza da proteína transportadora que trouxe os endossomos aos lisossomas e finalmente degradados. Em nosso estudo, a regulação positiva significativa de p62 indicou o término da degradação durante o processo de fusão de endo-lisossoma [21], que provavelmente foi causado pelo aumento do pH em vesículas de endo-lisossoma. Assim, a endocitose do BPSi pode, em primeiro lugar, induzir a ocorrência de autofagia, e então inibir o processo de autofagia pelo aumento dos valores de pH do endo / lisossoma, devido à sua alcalinidade amida.
A fim de testemunhar as características de aumento de pH em endo / lisossomas por endocitose BPSi, duas sondas fluorescentes de pH comerciais foram empregadas em nosso estudo, pHrodo ™ Red Transferrin Conjugate (Thermo Fisher # P35376) e RatioWorks ™ PDMPO.
O pHrodo ™ Red como um indicador de pH intracelular comercial geralmente apresenta fluorescência fraca em pH neutro, mas aumenta a fluorescência conforme o pH cai. Era suposto quantificar o pH citosólico celular na faixa de 9–4 com um pKa de ~ 6,5 com excitação / emissão de 560/585 nm. Pudemos obter uma conclusão de análise qualitativa a partir de 6 determinações de linha celular de que a endocitose de NPs de BPSi tem a capacidade de aumentar os valores de pH em endo / lisossomas, devido ao enfraquecimento dos sinais fluorescentes vermelhos (Fig. S4 e S5). No entanto, após repetir os experimentos várias vezes seguindo os protocolos de operação do produto, dificilmente analisamos quantitativamente o valor exato de pH que diminuiu entre as diferentes linhagens de células antes ou depois da alimentação com NPs de BPSi devido a não haver correlação entre a intensidade e os valores de pH estabelecidos.
O PDMPO foi então empregado como a melhor solução para indicar as mudanças no valor do pH após a endocitose do BPSi, o que introduz técnicas de imagem de razão na medição quantitativa do pH. PDMPO [2- (4-piridil) -5 - ((4- (2-dimetilaminoetil-laminocarbamoil) metoxi) fenil) oxazol] é caracterizado como sonda de pH de excitação dupla acidotrópica e emissão dupla. Ele emite fluorescência verde intensa em pH mais baixo e dá fluorescência azul intensa em pH mais alto. Esta fluorescência dependente do pH única torna o PDMPO uma sonda de pH ideal para organelas ácidas com pKa =4,47. O PDMPO marca seletivamente organelas ácidas (como lisossomos) de células vivas e os dois picos de emissão distintos podem ser usados para monitorar as flutuações de pH de células vivas em medições de proporção. No entanto, ainda falhamos na medição dos valores de pH em 6 linhas de células antes e depois da alimentação com BPSi. Como os resultados mostrados na Fig. S6, nenhuma diferença significativa foi observada em todas as 6 linhas de células antes e depois da alimentação com BPSi. Embora uma correlação tenha sido estabelecida entre a relação azul / verde e os valores de pH (Fig. S7), a correlação não linear de pH 4-5 faz com que o método PDMPO falhe na análise quantitativa de endo / lisossomos antes e depois das alimentações com BPSi.
A partir dos dados de dois indicadores comerciais de pH acima, primeiro demonstramos nossa suspeita de que nanopartículas decoradas com PEG com amida na cadeia de PEG poderiam fazer o pH do endo / lisossoma aumentar devido à natureza alcalina da amida. No entanto, sem a análise quantitativa de mudanças precisas de pH (intervalo de pH 0,1), ainda não podemos estabelecer as correlações entre o estado de autofagia e os valores de pH de endo / lisossoma, portanto falhou na previsão de autofagia.
Com base em nosso estudo anterior sobre nanopartículas auto-decomponíveis [13, 14] [15, 22], mantivemos a mesma concentração de hidróxido de amônio em 75% de etanol, mas ajustamos as concentrações de MB e TEOS. Duas séries de quantidade de TEOS foram definidas como 100 μL e 80 μL, a fim de obter diferentes espessuras de casca e tamanhos de poros. Dez séries de quantidade de MB foram definidas para obter diferentes tamanhos da estrutura oca central e eficácia de carregamento de MB.
As quantidades de MB e TEOS adicionadas nos protocolos foram conforme descrito na Tabela 1 abaixo.
Conforme mostrado na Fig. 2a, b, o tamanho das nanopartículas aumentou com o aumento da quantidade de MB, em ambas as concentrações de TEOS (100 μL e 80 μL). Na mesma concentração de MB, o tamanho de partícula aumentou com o aumento da quantidade de TEOS. Além disso, com o aumento da quantidade de TEOS, a espessura da casca aumentou, o que foi comprovado pelo mapeamento dos elementos (mostrado na Fig. 2c). The polydispersity index (PDI) and surface charge of the nanoparticles are shown in Fig. S8 and Table S1. The morphology studies predicted that with the increase of MB amount, the loading efficiency will grow up, leading to the faster release profile, while with the increase of TEOS amount, the release will slow down. And we need to find out the appropriate MB and TEOS concentration, with which we could obtain the optimized nanoparticle systems, that we may be able to make the MB release profile linear correlated with the pH changes.

Morphology characterization of 10 different self-decomposable nanoparticles with specific MB or TEOS amount. a STEM figures. b Nanoparticle size distribution analysis. c Si mapping of 10 self-decomposable nanoparticles. Scale bars in all figures are 100 nm. The size distribution analysis was performed by randomly chosen 100 nanoparticles from STEM figures and measured by ImageJ software. Data was presented as mean ± S.D.
The MB loading efficiency was determined by UV-Vis spectrum. The standard curve (Fig. S9) of MB was firstly drawn using series concentrations of MB solution (from 6.25 to 46.88 μg/mL), with the equation as y = 67.63x + 0.10919, R 2 = 0.9987. As calculated with the equation above, we obtain MB loading efficiency of 10 self-decomposable nanoparticles with specific parameters, detailed data shown in Fig. S10.
Before the study of the MB release profiles in different pH solutions, the release profiles in pure water have been studied. As shown in Fig. S11 and Fig. S12, all the nanoparticles with TEOS amount of 80 μL presented increased MB release along with the duration increase, which was reflected by the UV–Vis absorption. Moreover, with the MB encapsulated amount increase, the growth trend of MB release becomes more significant. Also, the release velocity grows faster. However, as the TEOS amount increase to 100 μL, the particle surface became more densed and the release becomes slower when the MB amount below 3.0 mg; almost no increase trend could be observed in the MB release in water during 14 days of release. As long as the MB amount increases to above 4.0 mg, an obvious increase trend of MB release could be observed. One thing to be noticed is that the nanoparticle parameter of both NPs7.5/80 and NPs6.0/100 presented solid growth as the time prolongs, almost showed a linear increase trend during the first 7 days, and then reached the platform.
Then, we focused on the MB release behavior in different pH buffers to figure out whether self-decomposable nanoparticles with specific parameters could have the linear pH-dependent MB release.
Firstly, we carried out the MB release experiments at pH 4.0 buffer solution. From Fig. S13, we could easily reach the conclusion that with the same TEOS amount of 100 μL, the MB release velocity presented a similar trend was observed in the 5 nanoparticle systems of TEOS at 80 μL (Fig. S14), the center positive correlation with the MB encapsulated amount.
Concentrated MB diffuses into the surrounding solution via diffusion due to concentration difference. The bigger concentration gradient makes the faster MB release. Compared with the MB release in pure water, we found that the acidic environment speeded up the release of MB (Fig. S13 and S14 compared with Fig. S11 and S12), indicating that the MB release is not only driven by diffusion; however, in acidic solutions, electrostatic repulsion is also an important driven force due to the positive charge nature of MB. We then calculated the release percentage of each nanoparticle parameter according to the MB loading efficiency, MB standard curve, and the dilution ratio at measurements. The release percentage reflected the release speed of MB in pH 4.0 acidic solution, and the results (Fig. 3) showed that only the release percentage of NPs7.5/80 presented linear release in pH 4.0 solution. Other nanoparticle systems with specific MB and TEOS parameters showed similar release trends, and the release percentage did not have a linear growth. One exception is NPs6/100 , and the MB release reached the platform in only 72 h; thus, it was hard to tell whether the MB release could grow linear before that duration at this stage.
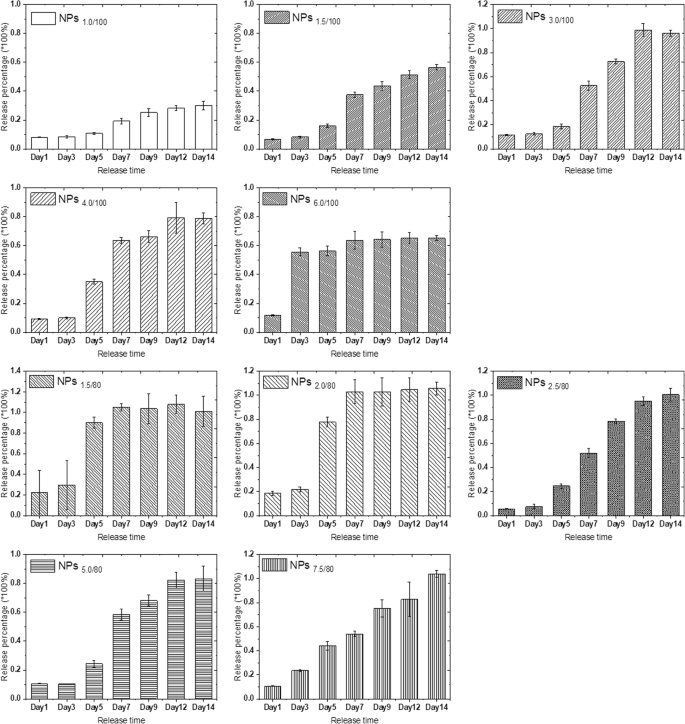
MB release percentage of 10 series self-decomposable nanoparticles after a specific duration in pH 4.0 buffer. All experiments were triple repeated, and the data were shown as mean ± S.D.
Meanwhile, we tested the MB release profiles in near-neutral and alkali buffers (pH 6.86 and pH 9.18). The results in both Fig. S15, S16, Fig. S17, and S18 demonstrated that the MB release slowed down with the solution pH increase to 6.86; moreover, with the central MB concentration increased, the MB release percentage decreased. At pH 9.18, all nanoparticles with 10 specific parameters presented a very slow MB release (Fig. S19 and S20); no matter in UV–Vis absorption or the release percentage, the trend was similar with the one in pH 6.86 buffer, but with even lower release percentage. So, it was clear that the self-decomposable nanoparticles only presented MB release linear growth in acidic solutions. We thought back to the endo/lysosomes pH, from 4 to 5, which is exactly the pH range of MB linear growth as a function of time in a specific MB/TEOS parameter. Thus, we get more confident that the self-decomposable nanoparticle system may be an accurate measuring tool for quantitative determining the endo/lysosome average pH, then provide evidence on the exact pH value of autophagy status.
The precondition of using the specific self-decomposable nanoparticles as an endo/lysosome pH indicator is that the nanoparticles stay stable in the endo/lysosome during the whole measurement process. Secondly, the MB release in endo/lysosome should occur smoothly when the measurement carried out.
The colocalization of the nanoparticle in the endo/lysosomes by cell TEM study and the MB release in 6 different cell lines were studied. From the cell TEM results, all of the 10 series nanoparticles stayed in the endo/lysosomes without escaping, after 24-h incubation with the HepG-2 cells (Fig. 4). Since the diameter of HepG2 cells used in Fig. 4 is about 10–20 μm and the diameter of MB@SiO2 nanoparticles is between 75 and 200 nm, it will be very difficult to clarify the nanoparticle morphologies using the images with low magnification (as shown in Fig. S21). We also investigated the intracellular location of the nanoparticles in the other 5 cell lines, and 4 nanoparticles were randomly selected to demonstrate the nanoparticles were trapped in the endo/lysosomes (Fig. S22). The nanoparticles with all parameters showed a central hollow structure in all other 5 cell lines after 24-h incubation, indicating the MB release. Moreover, under more precise observation, we noticed the MB release may be different due to different hollow sizes, pointing to the fact that (1) the endo/lysosome pH in different cells is different and (2) the MB release from the nanoparticles is very sensitive to the endo/lysosomes pH, especially for the NPs6/100 and NPs7.5/80 . From Fig. S23, we can find that nanoparticles have already realized the endocytosis and stayed in the vesicles 2 h after nanoparticle feeding, then nanoparticles gradually accumulated in lysosomes.
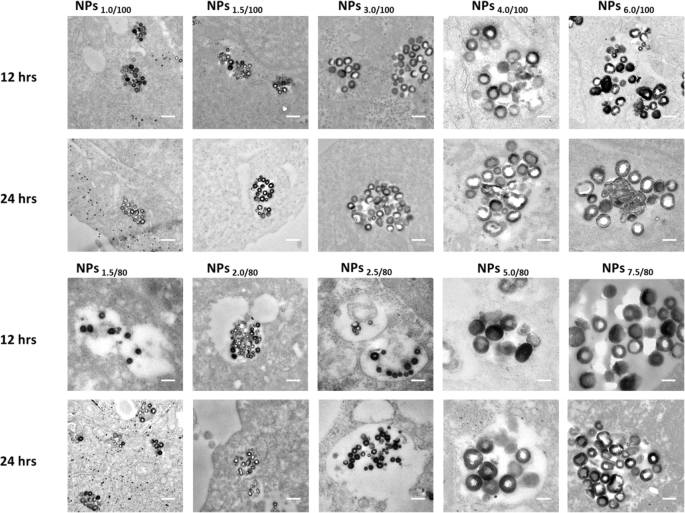
The colocalization of the nanoparticle in the endo/lysosomes by cell TEM study after 12 and 24 h incubation with the 10 series nanoparticles. The scale bar is 200 nm in the TEM images
We then evaluated the correlation between the pH values and the OD values in pH 4.0–4.8. From the results in Fig. 5 and Fig. S24, for NPs6/100 and NPs7.5/80 nanoparticle systems, the MB release presented a linear decrease as a function of pH in the pH range from 4.0 to 4.8.
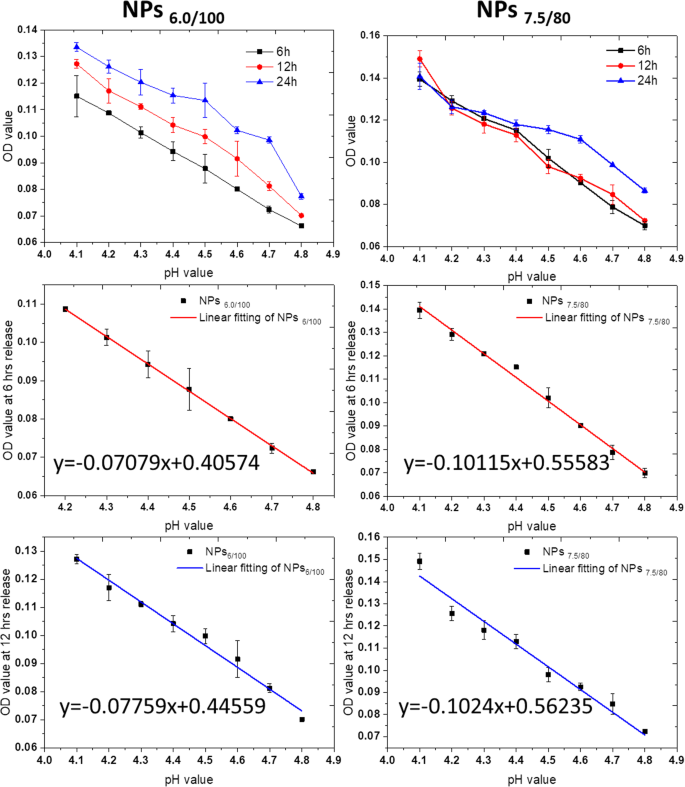
MB release as a function of pH values in NPs6/100 and NPs7.5/80 after specific incubation duration, 6 h, 12 h, and 24 h. The linear correlation equations were also calculated for 6 h and 12 h for MB release from both NPs6/100 and NPs 7.5/80 as a function of pH values. All experiments were carried out triplicated, and the data were shown as mean ± S.D.
We then converted the OD value to the MB release percentage according to the MB loading efficiency and feeding amount. As shown in Fig. S25, in the first 6 h, the MB release percentage in NPs6/100 and NPs7.5/80 nanoparticle systems also presented as a function of pH values. We then calculated the residual sum of squares and Pearson’s related coefficient at 6 h and 12 h release duration, respectively, as the residual sum of squares present a negative correlation with closeness of linear fitting, while the closer the absolute value of Pearson’s related coefficient to 1, the more linear it is. As shown in Table S2 and S3, the highest degree of linearity is the fitting of NPs6.0/100 nanoparticle systems, followed by the one of NPs7.5/80 at 6 h release.
Till then, we were so excited by the results that the method for precisely monitoring the pH values has been established, especially with the accuracy less than or equal to 0.1 pH value interval. That means, we have great possibilities to figure out the correlation between endo/lysosome pH values and the autophagy status, which is of great significance for better studying the autophagy mechanism and predicting the autophagy process. As we can see in Fig. S26, the MB release in HepG-2 cells have already reached the plateau after incubation for 4 h. Thus, we chose 6 h as the observation time point.
We then carefully investigated the MB release of NPs6.0/100 in 6 cell lines in the nanoparticle cell interaction duration of 6 h, including liver cancer HepG-2 cell line, colon cancer HCT8, HCT 15, and HCT 116 cell lines, lung cancer A549 cell line, and myomelanocytic cancer B16 cell line.
As shown in Table 2, we clearly differentiate the endo/lysosomes in 6 cancer cell lines, with the accuracy at 0.01 pH values, which is impossible to be done with the commercial intraocular pH indicator kits.
Moreover, we re-evaluated pH values in endo/lysosomes of the HepG2 cells before and after cultured with BPSi nanoparticles. We reached the conclusion that BPSi uptake significantly increases the endo/lysosome pH values, from 4.70 ± 0.09 to 5.59 ± 0.05, perfectly illustrating the reason for BPsi uptaken induced the autophagy initially then terminated the autophagy flux. The intracellular uptake of BPSi makes the quantities of the endo/lysosomes increased, which was consistent with the results of gene sequencing, that autophagy-related genes (TFEB-CLEAR) were activated. Meanwhile, the autophagy termination by the increased pH values in endo/lysosomes also coincide with the results of p62 proteins upregulation in Western blot study.
Discussion
Nanoparticles can generally cause autophagy in cells [23], and studies have shown that the autophagic response to nanoparticles presenting a neutral or anionic surface involves enhanced clearance of autophagic cargo. Cell exposure to nanoparticles presenting a cationic surface, on the other hand, results in transcriptional upregulation of the TFEB pathway, but also causes lysosomal dysfunction, ultimately resulting in blockage of autophagic flux [7]. And our results are in consistent with these previous conclusions. In our study, we found that the expression of autophagy-related genes and proteins in HepG2 cells has been increased after feeding of BPSi nanoparticles through transcriptome sequencing, RT-qPCR, and Western experiments. However, the expression level of autophagy-related P62 protein does not decrease as the autophagy is activated. We suspect that the PEG-amine on the surface of BPSi nanoparticles raises the pH value of the lysosome, resulting in inhibition of P62 degradation. Existing lysosomal pH indicators cannot verify our guess. To accurately measure the lysosomal pH of living cells, we established a new method for endo/lysosomes pH qualitative determination based on self-decomposable nanoparticle systems. Ten nanoparticle systems with specific MB/TEOS parameters were employed for obtaining optimized pH sensitively responsive measurement method. The radial MB concentration gradient from inner out served as a major driving force for MB release. The drug release proceeded with simultaneously carrier decomposition, which was driven by a diffusion-controlled mechanism. Moreover, as the pH value decreases, the hydrogen ion concentration increases, and the enhanced electrostatic interaction promotes inner MB to release faster than in neutral solution [24]. The optimized central hollow nanoparticle system could release the central concentrated MB as a linear function of precise pH values in the range of pH 4.0–4.8, which is exactly the pH of lysosomes. Finally, by this qualitative pH indicator based on self-decomposable nanoparticles, we have succeeded in the detection of the average pH values of lysosomes in 6 cell lines. Moreover, by this system, we can qualitatively differentiate the pH changes of lysosomes before and after BPSi nanoparticle endocytosis by HepG-2 cells, clarifying the mechanism of the autophagy occurrence and then termination after BPSi endocytosis. The self-decomposable nanoparticle systems pave a brand new way for studying the luminal pH values, providing new tools to know better of the cell signaling and metabolism, and then providing new ways and methods for the treatment of cancer [25, 26].
Conclusão
In this study, we found that BPSi can promote cell autophagy through transcriptome sequencing, but the amino groups on the surface of the nanoparticles can increase the pH of the lysosome and inhibit the degradation of autophagic flow. Thus, the lysosome pH significantly influences the autophagy stages. And precisely acquiring the information of lysosome pH will promote the perceiving of autophagy. However, the existing fluorescent lysosomal pH indicators could only determine a wide range of lysosomal pH; thus, we established a precise lysosomal pH indicator based on the self-dissociation system. By adjusting the synthesis parameters of MB@SiO2 , the release of MB loaded on the nanoparticles was linearly and negatively correlated with pH. And the nanoparticles mainly stay in the lysosome after entering the cell. By measuring the amount of MB released in the cells, the pH value of the lysosome can be calculated exactly according to the linear function. The established precise pH indicator provided a brand new tool and methodology to precisely study the lysosome pH values and further acquire more information on autophagy.
Disponibilidade de dados e materiais
Todos os dados gerados ou analisados durante este estudo estão incluídos neste artigo publicado e seus arquivos de informações complementares.
Abreviações
- BPSi:
-
Black mesoporous silicon
- FBS:
-
Soro fetal bovino
- GO:
-
Gene ontology
- HCl:
-
Ácido clorídrico
- KEGG:
-
Kyoto Encyclopedia of Genes and Genomes
- MB:
-
Azul de metileno
- NaBr:
-
Sodium bromide
- NaSi:
-
Sodium silicide
- NH4Br:
-
Ammonium bromide
- NPs:
-
Nanopartículas
- OD:
-
Densidade ótica
- PBS:
-
Salina tamponada com fosfato
- PDMPO:
-
2-(4-Pyridyl)-5-((4-(2-dimethylaminoethy- laminocarbamoyl) methoxy) phenyl) oxazole
- RT-qPCR:
-
Reação em cadeia da polimerase quantitativa com transcrição reversa
- TEOS:
-
Tetraethyl orthosilicate
Nanomateriais
- Monitoramento de aplicativo SaaS:Vendo a floresta e as árvores
- O desenvolvimento e aplicação de tecido de fibra de vidro
- Preparação de nanopartículas de mPEG-ICA carregadas com ICA e sua aplicação no tratamento de dano celular H9c2 induzido por LPS
- Absorvedor perfeito de banda ultra-estreita e sua aplicação como sensor plasmônico na região visível
- Toxicidade potencial para fígado, cérebro e embrião de nanopartículas de dióxido de titânio em camundongos
- Propriedades de formação e luminescência de Al2O3:Nanocompósitos de SiOC na base de nanopartículas de alumina modificados por feniltrimetoxisilano
- A preparação da nanoestrutura de casca de gema de Au @ TiO2 e suas aplicações para degradação e detecção de azul de metileno
- Adepto verde na síntese e estabilização de nanopartículas de cobre:atividades catalíticas, antibacterianas, citotoxicidade e antioxidantes
- Estudos de TEM e STEM nas morfologias transversais de filmes anti-reflexo de SiO2 de banda larga dupla / tripla
- Película de capacitor:as propriedades, construção e aplicação



