MicroRNA exossômico derivado de macrófago M1-326 suprime a progressão celular do carcinoma hepatocelular por meio da via de sinalização NF-κB de mediação
Resumo
O acúmulo de evidências mostrou que o microRNA (miR) derivado de exossomos derivados de macrófagos M1 pode regular a progressão do carcinoma hepatocelular (HCC). No entanto, o efeito de miR-326 derivado de exossomos derivados de macrófagos M1 em HCC não foi relatado. Portanto, o objetivo do presente estudo foi explorar o mecanismo do miR-326 exossômico de macrófagos M1 na regulação da progressão celular do CHC. RT-qPCR detectou a expressão de miR-326 em linhas de células HCC. A expressão de miR-326 em HCC foi alterada por transfecção, e o efeito de miR-326 na expressão de CD206 e NF-κB, proliferação celular, formação de colônia, migração, apoptose e invasão foi detectado. Posteriormente, os exossomos foram isolados de macrófagos M1. RT-qPCR identificou a expressão de miR-326 em exossomos derivados de macrófagos M1. A expressão de miR-326 em exossomos derivados de macrófagos M1 foi alterada por transfecção. Os exossomos derivados de macrófagos M1 foram co-cultivados com células HCC para descobrir seus efeitos no progresso biológico das células HCC. Finalmente, experimentos in vivo foram realizados para verificar os resultados in vitro. MiR-326 foi diminuído em células HCC e enriquecido em exossomos derivados de macrófagos M1. A regulação positiva do miR-326 inibiria a proliferação de células HCC, formação de colônias, migração, invasão e expressão de CD206 e NF-κB e promoveu apoptose, e inibiu o crescimento de tumores HCC in vivo , enquanto a regulação negativa do miR-326 mostrou efeitos opostos. Exossomos derivados de macrófagos M1 inibiram a proliferação de células HCC, formação de colônias, migração, invasão e expressão de CD206 e NF-κB e apoptose aumentada, enquanto a superexpressão de miR-326 aumentou o efeito de exossomos derivados de macrófagos M1 em células HCC. É revelado que miR-326 exossômico derivado de macrófagos M1 suprime a proliferação, migração e invasão, bem como avança a apoptose de HCC através da regulação negativa da expressão de NF-κB.
Introdução
O carcinoma hepatocelular (CHC) é o quinto câncer mais comum em todo o mundo e o câncer primário de fígado mais comum [1]. Manifestado pelos dados do Registro Nacional de Câncer da China, a taxa de mortalidade por câncer primário de fígado está listada em terceiro lugar, enquanto a incidência é a quarta entre as doenças malignas comuns [2]. Os principais fatores de risco para CHC são infecção crônica com vírus da hepatite C e vírus da hepatite B, alimentos contaminados com aflatoxina, obesidade, tabagismo, ingestão excessiva de álcool e diabetes tipo 2 [3]. A quimioembolização transarterial é um tratamento estabelecido para o CHC em estágio intermediário, o que melhora a sobrevida da maioria dos pacientes com CHC em estágios intermediários ou avançados [4]. Atualmente, o diagnóstico de CHC depende principalmente de biomarcadores séricos e técnicas de imagem [5]. A taxa de sobrevida em 5 anos relacionada ao CHC é de apenas 60%, e a incidência aumenta ano a ano nos últimos anos [6]. Diante disso, pesquisar um alvo terapêutico preciso é uma prioridade no tratamento do CHC.
Os macrófagos são células efetoras e principais reguladores do sistema imunológico, exercem funções enormes na remodelação e reparo de tecidos e realizam funções metabólicas orquestradas em quase todos os tecidos in vivo [7]. É revelado que os macrófagos M1 exercem efeitos promotores de tumor e aumentam a motilidade das células HCC [8]. O exossomo é uma vesícula discóide com diâmetro de 40-150 nm [9]. De acordo com Xu et al., MicroRNAs exossômicos (miRNAs) têm funções na proliferação, invasividade, metástase e resistência a drogas do HCC via modulação da expressão gênica nas células-alvo [10]. Um estudo demonstrou que exossomos contendo miR-326 podem ser um alvo clínico potencial na esclerose múltipla [11]. MiRNAs podem atuar como oncogenes ou inibidores de tumor através da modulação de um grande número de expressão de genes codificados por proteínas por meio da degradação do mRNA e bloqueio da tradução em uma sequência específica [12]. Um estudo discutiu que miR-326 suprime o crescimento de células HCC através da perturbação da progressão do ciclo celular, bem como aumentando a apoptose, além de suprimir a invasão celular através da diminuição do fenótipo de transição epitelial-mesenquimal [13]. Outro estudo relatou que o miR-326 pode ser um alvo terapêutico potencial para o tratamento de pacientes com CHC [14]. Portanto, o presente estudo discutiu o mecanismo do miR-326 exossômico regulando a invasão e migração de células HCC.
Materiais e métodos
Declaração de Ética
Todos os experimentos com animais estavam de acordo com o Guia para o Cuidado e Uso de Animais de Laboratório pelos Comitês Internacionais. O protocolo foi aprovado pelo Comitê de Uso de Cuidados com Animais Institucionais do Terceiro Hospital da Universidade de Jilin.
Indução e identificação de macrófagos
A linha de células monocíticas humanas THP-1 (Kunming Institute of Zoology, CAS, Kunming, China) foi cultivada em meio RPMI 1640 (Gibco, CA, EUA; Thermo Fisher Scientific, MA, EUA) contendo 10% de soro fetal bovino inativado por calor ( FBS). As células THP-1 foram reagidas com 150 ng / mL de forbol 12-miristato 13-acetato (PMA; P8139, Sigma-Aldrich, SF, CA, EUA) e incubadas em meio RPMI por 24 h para obter macrófagos M0. Em seguida, a morfologia celular antes e após a indução foi observada pela coloração de Wright. As células THP-1 e os macrófagos induzidos foram ressuspensos em 5 μL de PBS, colocados na lâmina de vidro, corados pela solução corante de Wright e misturados com a solução tampão a 1:2. Tingidas por 10 min e enxaguadas com água corrente, as células foram observadas ao microscópio. Além disso, os marcadores de macrófagos M0 (CD68 e CD206) foram medidos por reação em cadeia da polimerase quantitativa de transcrição reversa (RT-qPCR). Em seguida, os macrófagos foram induzidos em macrófagos M1 através da incubação com 20 ng / mL de Interferon (IFN) -γ (# 285-IF; R&D Systems, MN, EUA) e 10 pg / mL de LPS (# 8630; Sigma-Aldrich) por 18 h. Marcadores de macrófagos M1 (IDO1 e IL-12 p35) foram examinados por RT-qPCR [15].
Extração de exossomos
Os exossomos foram extraídos por kit de separação de exossomos (ExoEasy Maxi Kit, Qiagen, Hilden, Alemanha). O sobrenadante do macrófago foi acumulado em um tubo de centrífuga de 15 mL sob condições assépticas e filtrado por um filme de filtragem de 0,8 μM. O sobrenadante celular em cada grupo foi anexado com tampão XBP (1:1) e, em seguida, centrifugado com coluna de centrifugação de afinidade de membrana exoEasy a 500 g . As células foram suplementadas com 10 mL de tampão XWP e centrifugadas com 3000-5000 g . A coluna de centrifugação de afinidade de membrana exoEasy foi incubada com 400 μL de tampão de eluição XE e centrifugada a 500 g . O tampão de eluição foi transferido para coluna de centrifugação de afinidade de membrana exoEasy e centrifugado a 500 g . O tampão de eluição foi armazenado por 24 h a 4 ° C e, em seguida, usado para identificação. O resto foi armazenado a -80 ° C.
Observação de TEM e análise de rastreamento de nanopartículas (NTA)
Os exossomos obtidos acima foram colocados na malha de cobre da membrana suportada por carbono e adicionados com 2% de ácido fosfotúngstico. A amostra foi observada em um TEM, e a imagem ideal foi coletada e analisada.
As impurezas e partículas em PBS foram removidas por um filtro microporoso de 0,22 μM. De acordo com a densidade de partículas dos exossomos, o PBS filtrado foi diluído para a concentração apropriada e detectado usando um detector de nanopartículas Nanosight NS300 (Malvern, Westborough, MA, EUA).
Após a identificação, exossomos derivados de macrófagos transfectados com inibidor miR-326 e controle negativo do inibidor miR-326 (NC), miR-326 mimetizador e miR-326 mimetizador NC foram extraídos por kit de separação de exossomos (Invitrogen).
RT-qPCR
O RNA total foi extraído pelo Trizol Reagent (Thermo Fisher), e a PCR em tempo real foi realizada usando o SYBR-Green PCR Master Mix (Roche) e o ABI 7500 Real-Time PCR System (Life Technologies, Grand Island, NY, EUA ) As sequências de primer são mostradas na Tabela 1. A análise quantitativa foi realizada usando o método de 2 - △△ Ct .
Análise de Western Blot
A proteína total das células e exossomos foi extraída. A concentração de proteína foi determinada pelo kit de ácido bicinconínico (BCA) (Boster Biological Technology Co. Ltd., Wuhan, Hubei, China). A proteína foi anexada com tampão de amostra e fervida a 95 ° C, e cada poço foi carregado com 30 µg. A proteína foi separada com eletroforese em gel de poliacrilamida 10% (Boster Biological Technology) e eletroblot em membrana de fluoreto de polivinilideno, seguida de selagem em albumina de soro bovino a 5% (BSA). A membrana foi incubada com anticorpo primário contra CD63 (1:1000, Developmental Studies Hybridoma Bank, University of Iowa, Ames, IA, USA), CD181 (1:1000, R&D Systems), GAPDH (1:2000, Jackson ImmunoResearch Laboratories, PA, EUA) e com o anticorpo secundário (1:500, Jackson ImmunoResearch Laboratories) marcado por peroxidase de rábano. As imagens foram obtidas pelo sistema de imagem de varredura por fluorescência de infravermelho de duas cores Odyssey, e os valores de cinza das bandas foram medidos pelo software de análise de imagem Quantity One.
Cultura de células e triagem
A linha celular normal do fígado HL-7702 e a linha celular humana HCC BEL-7404, HepG2, SMMC-7721 e QGY-7703 foram selecionadas e cultivadas em Gibco RPMI Media 1640 com 10% de soro fetal bovino (FBS), penicilina (100 U / mL ) e estreptomicina (100 mg / mL). A expressão de MiR-326 foi detectada por RT-qPCR e as linhas celulares adequadas foram selecionadas.
Marcação de exossomos e captação de exossomos
Os exossomos foram ressuspensos com 250 μL de Diluente C e suavemente triturados evitando causar danos à membrana do exossomo. O corante PKH67 (1 μL, Sigma-Aldrich) foi adicionado a 250 μL de Diluente C para atingir 500 μL e incubado. A solução foi anexada com 500 μL de BSA a 1% e incubada a 37 ° C por 1 min. Os exossomos foram obtidos por centrifugação a 120.000 g 4 ° C por 2 h. Os exossomos marcados com PKH67 foram obtidos por centrifugação a 120.000 g 4 ° C por 2 h. Os exossomos foram ressuspensos em 6 mL de meio RPMI-1640 evitando a luz. Em seguida, os exossomos marcados foram co-cultivados com células HCC por 12 h. Depois disso, o meio de cultura foi removido e lavado com PBS por 3 vezes, 5 min / tempo, e os exossomos marcados com fluorescência que não foram absorvidos internamente pelas células HCC foram completamente lavados. Os exossomos foram fixados com paraformaldeído 4% e tingidos com 4'-6-diamidino-2-fenilindol. Após a selagem, a distribuição da fluorescência foi observada por um microscópio confocal a laser.
Agrupamento e tratamento de células
As células HepG2 e as células SMMC-7721 foram semeadas na placa de 12 poços em 0,5-1 × 10 6 células / poço. Com confluência de 50-60%, as células foram transfectadas com Lipofectamine 2000 (Invitrogen, Carlsbad, CA). As células HepG2 foram distribuídas no grupo miR-326-mimetizador (transfectado com miR-326 mimetizador) e grupo NC-mimetizador (transfectado com miR-326 mimetizador NC). As células SMMC-7721 foram atribuídas ao grupo inibidor miR-326 (transfectado com inibidor miR-326) e grupo inibidor NC (transfectado com inibidor miR-326 NC). miR-326-mimetizador, miR-326-inibidor e seus NCs foram misturados com Lipofectamine 2000 para transfecção. As células HepG2 e as células SMMC-7721 sem qualquer tratamento foram definidas como o grupo em branco. miR-326-mimic, miR-326-inibidor e seus NC foram concebidos e compostos por Guangzhou RibBio Co., Ltd. (Guangzhou, China) (Tabela 1).
Co-cultura de exossomos derivados de macrófagos M1 com células HCC
A concentração de proteína da suspensão de exossomos derivados de macrófagos M1 foi detectada pelo método BCA, e o volume da suspensão de exossomos correspondentes com 50 μg de proteína foi calculado. As células HepG2 e as células SMMC-7721 foram semeadas em placas de 12 poços em 1 × 10 5 células / mL por poço. As células HepG2 foram distribuídas em 4 grupos:grupo de controle (células HepG2 não co-cultivadas com exossomos), grupo de exossomos (Exo) (células HepG2 co-cultivadas com exossomos derivados de macrófagos M1), grupo Exo-miR-326-mimético (HepG2 células co-cultivadas com exossomos derivados de macrófagos M1 que transfectaram com miR-326 mimetizador), grupo Exo-NC-mimético (células HepG2 co-cultivadas com exossomos derivados de macrófagos M1 que transfectaram com miR-326 mimetizador NC). As células SMMC-7721 também foram atribuídas em 4 grupos:grupo em branco (células SMMC-7721 não co-cultivadas com exossomos), grupo Exo (células SMMC-7721 co-cultivadas com exossomos derivados de macrófagos M1), Exo-miR-326- grupo inibidor (células SMMC-7721 co-cultivadas com exossomos derivados de macrófagos M1 que transfectaram com inibidor miR-326), grupo inibidor Exo-NC (células SMMC-7721 co-cultivadas com exossomos derivados de macrófagos M1 que transfectaram com miR- 326 inibidor NC).
Ensaio de 3- (4, 5-Dimetiltiazol-2-il) -2,5-difeniltetrazólio (MTT)
As células foram destacadas com tripsina e semeadas em placa de 96 poços com densidade celular de 4 × 10 4 células por poço. O meio de cultura foi abandonado após a cultura de 12, 24, 36, 48, 60 h, respectivamente. Incubadas com 500 μL de solução de 0,5 g / L de MTT, as células foram anexadas com 200 μL de solução de dimetilsulfóxido, trituradas e incubadas. Os valores de densidade óptica (DO, 490 nm) foram medidos por um leitor de microplaca.
Ensaio de formação de colônia
Cultivadas por 24 he destacadas com tripsina, as células foram semeadas em um pequeno prato de 35 mm com 300 células por prato. A solução foi substituída a cada 3 d. Após 10 dias de cultura, as células foram fixadas com 40 g / L −1 paraformaldeído e tingido com 1 g / L −1 solução de violeta cristal e seca. O número da colônia (mais de 50 células) foi calculado sob um microscópio.
Ensaio Transwell
Células (1 × 10 5 ) foram suspensos com 200 μL de meio de cultura em branco. Os experimentos foram conduzidos em conformidade com as instruções da câmara Transwell (Corning Glass Works, Corning, N.Y., EUA) (matrigel era necessário para o experimento de invasão, mas não para o experimento de migração). RPIM 1640 (10% FBS, 600 μL) foi adicionado à câmara inferior. As câmaras superior e inferior foram separadas por uma membrana Transwell pré-revestida por matrigel (BD Biosciences, Franklin Lakes, NJ, EUA). Cultivada por 24 horas, a câmara foi fixada com álcool 95%. Após a coloração com solução de cristal violeta, as células foram observadas em cinco campos visuais sob o microscópio.
Citometria de fluxo
Ciclo celular:as células foram separadas pela tripsina. Células (1 × 10 6 ) foram suspensos com 0,5 mL de PBS e triturados em uma suspensão única. Misturado com 4,5 mL de etanol a 70% pré-resfriado em gelo, as células foram centrifugadas a 3000 g , enxaguado com 5 mL de PBS e centrifugado novamente a 3000 g . Em seguida, as células foram suspensas com 1 mL de solução de coloração PI / Triton X-100 (20 μg PI / Triton X-100 0,1%) contendo 0,2 mg de RnaseA. O ciclo celular foi detectado por citometria de fluxo.
Apoptose celular:células tripsinizadas (1 × 10 6 ) foram suspensos com 1 mL de PBS, triturados e centrifugados a 3000 g . As células foram enxaguadas sucessivamente com tampão de incubação (10 mmol / L Hepes / NaOH, pH 7,4, 140 mmol / L NaCl, 5 mmol / L CaCl 2 ) e centrifugado a 3000 g . Em seguida, as células foram incubadas com 100 μL de solução de marcação (FITC-Anexina V e PI foram adicionados ao tampão de incubação para atingir 1 μg / mL), centrifugadas a 3000 g , lavado uma vez com tampão de incubação e incubado com solução fluorescente (SA-FLOUS). A apoptose celular foi detectada por citometria de fluxo. O comprimento de onda da citometria de fluxo foi de 488 nm, e a fluorescência FITC foi detectada por filtro de banda em 515 nm, enquanto o PI com comprimento de onda maior que 560 nm. Os resultados foram analisados automaticamente por computador.
Xenoenxerto de tumor em camundongos nus
Quarenta camundongos (College of Animal Science, Jilin University, Jilin, China) com idades entre 4-6 w foram distribuídos aleatoriamente em 8 grupos, com 5 camundongos em cada grupo. Os camundongos foram alimentados em um laboratório de animais de grau livre de patógenos específicos por 1 semana, e a ração, a almofada e a garrafa d'água foram substituídas a tempo. O estado de saúde dos ratos deve ser observado todos os dias. Uma semana depois, as células HCC foram preparadas em suspensão de células e injetadas por via subcutânea no pescoço e nas costas em 0,1 mL de suspensão de células (1 × 10 6 ) O crescimento do tumor foi observado após 3-5 d. Os camundongos nus foram pesados a cada 4 dias e o volume do tumor foi medido. Os camundongos nus foram sacrificados 20 dias após a injeção.
Análise estatística
Todos os dados foram interpretados pelo software SPSS 17.0 (SPSS Statistics, Chicago, IL, USA). Os dados de medição foram indicados como média ± desvio padrão. As comparações entre dois grupos foram formuladas pelo t -teste, enquanto as comparações entre vários grupos foram avaliadas por análise de variância unilateral (ANOVA). P valor <0,05 foi indicativo de diferença estatisticamente significativa.
Resultados
Identificação de macrófagos e exossomos M1
A coloração de Wright foi utilizada para observar a morfologia das células THP-1 induzidas por PMA. Foi retratado que o volume de células THP-1 antes da indução era pequeno e a proporção de carioplasma era maior; a morfologia das células após a indução era irregular, o volume tornou-se maior e a proporção de carioplasma diminuiu; o citoplasma era mais rico e apresentava-se em azul claro, com partículas abundantes e alguns vacúolos; o núcleo era vermelho arroxeado e frequentemente inclinado para um lado, mostrando que as células tinham as características morfológicas típicas dos macrófagos (fig. 1a).
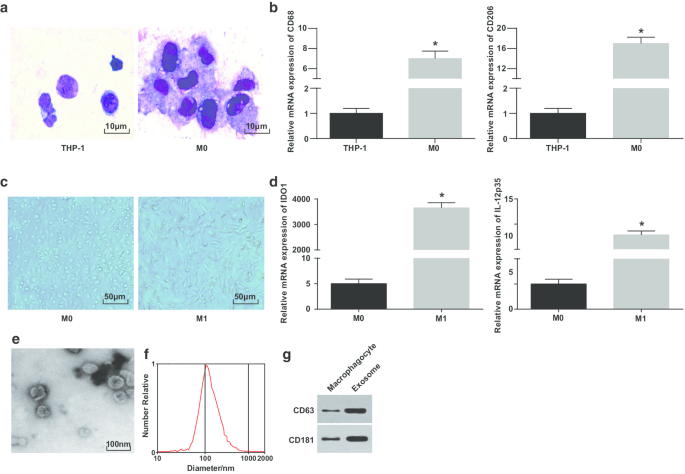
Identificação de macrófagos e exossomos M1. a Coloração de Wright para observar a morfologia das células THP-1 antes e após o tratamento com PMA. b Expressão de CD68 e CD206 em células THP-1 e células THP-1 tratadas com PMA detectadas por RT-qPCR; c morfologia de macrófagos e macrófagos M1. d Expressão de IDO1 e IL-12 p35 em macrófagos M0 e macrófagos M0 tratados com LPS e INF-γ detectados por RT-qPCR. e TEM para observação de exossomos. f Detecção da distribuição de tamanho de partícula de exossomos por NTA. g Bandas de proteína de CD63 e CD181. No painel b, * P <0,05 versus células THP-1; No painel d, * P <0,05 contra macrófagos M0. Os dados de medição foram representados como média ± desvio padrão ( N =3), as comparações entre dois grupos foram realizadas por teste t
Para verificar ainda mais o sucesso da indução de macrófagos, a expressão de CD68 e CD206 antes e depois da indução foi testada por RT-qPCR. Foi manifestado que a expressão de CD68 e CD206 estava elevada desde a indução de PMA, indicando que as células THP-1 induzidas com sucesso por PMA em macrófagos M0 (Fig. 1b). Em seguida, macrófagos M0 foram polarizados em macrófagos M1 por indução de LPS e INF-γ. A morfologia e o marcador de superfície de macrófago do tipo M1 IDO1 e a expressão de IL-12 p35 de macrófagos foram observados e testados. Os macrófagos M0 apresentaram morfologias aderentes diversas e irregulares, apresentando formato redondo, elíptico ou fusiforme. A morfologia dos macrófagos estimulados por IFN-γ demonstrada com mais pseudópodes e protrusões e fusiformes (Fig. 1c). RT-qPCR descreveu (Fig. 1d) que após o tratamento com LPS e INF-γ, macrófagos M1 mostraram um aumento em seus marcadores (IDO1 e IL-12p35).
Posteriormente, os exossomos derivados de macrófagos foram observados pelo TEM. Revelou-se que os exossomos derivados dos macrófagos eram ricos e de formato redondo ou oval, com estrutura membranosa, tamanho uniforme e menos poluentes (Fig. 1e). O NTA mostrou que os exossomos com um pico centralizado de curva MODE e linear suave tinham diâmetro mais concentrado e menos impurezas (Fig. 1f). A análise de Western blot relatou que em relação aos macrófagos, a expressão da proteína marcadora específica CD63 e CD181 aumentou em exossomos derivados de macrófagos (Fig. 1g). Esses resultados indicam que induzimos monócitos com sucesso a se diferenciarem em macrófagos e os polarizamos em macrófagos M1.
Exossomos derivados de macrófagos M1 Entregam miR-326 para células HCC e afetam a expressão de miR-326 em células HCC
Para verificar se os exossomos derivados de macrófagos M1 transportaram miR-326 para células HCC, as células HepG2 e SMMC-7721 foram co-cultivadas com exossomos. Pode ser reconhecido que um grande número de exossomos foram assimilados por células HepG2 e SMMC-7721 após transfecção de 4 h sob o microscópio de fluorescência (Fig. 2a, b).
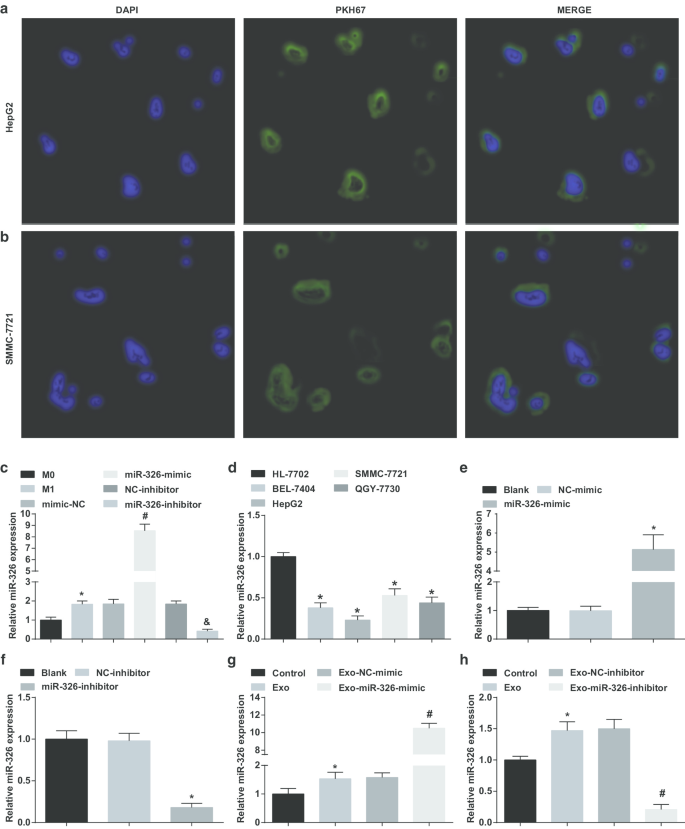
Exossomos derivados de macrófagos M1 entregam miR-326 para células HCC e afetam a expressão de miR-326 em células HCC. a Captação de exossomos derivados de macrófagos M1 por células HepG2. b Captação de exossomos derivados de macrófagos M1 por células SMMC-7721. c Comparação da expressão de miR-326 de exossomos de macrófagos em cada grupo antes e após a indução de INF-γ e LPS. d Expressão de miR-326 em linhas de células HCC (BEL-7404, HepG2, SMMC-7721, QGY-7703) e linha de células de hepatócitos humanos normais HL-7702 detectadas por RT-qPCR. e RT-qPCR detectou o efeito de miR-326 mimetizador na expressão de miR-326 em células HepG2. f RT-qPCR detectou o efeito do inibidor miR-326 na expressão de miR-326 em células SMMC-7721. g RT-qPCR detectou os efeitos de exossomos derivados de macrófagos M1 transfectados por mimetismo miR-326 na expressão de miR-326 em células HepG2. h RT-qPCR detectou os efeitos de exossomos derivados de macrófagos M1 transfectados com inibidor miR-326 na expressão de miR-326 em células SMMC-7721. No painel c, * P <0,05 versus o macrófago M0; No painel d, * P <0,05 versus células HL-7702; No painel e, * P <0,05 versus o grupo NC-mimetizador; No painel f, * P <0,05 versus o grupo de inibidor de NC; No painel g, * P <0,05 versus o grupo de controle, # P <0,05 versus o grupo Exo-NC-mimetizador; No painel h, * P <0,05 versus o grupo de controle, # P <0,05 versus o grupo de inibidores Exo-NC. Os dados de medição foram representados como média ± desvio padrão ( N =3), as comparações entre vários grupos foram conduzidas por uma análise de variância unilateral
Posteriormente, miR-326-mimetizador / inibidor foi transfectado em macrófagos e verificada a expressão de miR-326 em seus exossomos antes e após a transfecção. Foi demonstrado que em comparação com os exossomos de macrófagos M0, a expressão de miR-326 foi aumentada em exossomos derivados de macrófagos M1. A expressão elevada de miR-326 foi observada nos exossomos derivados de macrófagos M1 com tratamento mimetizador de miR-326. A expressão de MiR-326 foi reduzida em exossomos derivados de macrófagos M1 com tratamento com inibidor de miR-326 (Fig. 2c).
Em seguida, a expressão de miR-326 em linhas de células HCC foi testada. Conforme manifestado, a expressão de miR-326 foi diminuída nas células BEL-7404, HepG2, SMMC-7721 e QGY-7703 em comparação com as células HL-7702, entre as quais as células HepG2 se manifestaram com a expressão mais baixa, enquanto as células SMMC-7721 com a expressão mais alta (Fig. 2d).
Seguido por isso, miR-326-mimetizador e inibidor de miR-326 foram transfectados em células HepG2 e SSMC-7721, respectivamente, e examinaram seus efeitos na expressão de miR-326. MiR-326 imita a expressão elevada de miR-326 em células HepG2, enquanto o inibidor de miR-326 reduziu a expressão de miR-326 em células SSMC-7721 (Fig. 2e, f).
Em seguida, os exossomos de macrófagos M1 transfectados com miR-326-mimetizador e miR-326-inibidor foram co-cultivados com células HepG2 e SSMC-7721, respectivamente. Foi destacado que a co-cultura com exossomos aumentou a expressão de miR-326 em células HCC, exossomos derivados de macrófagos M1 transfectados com miR-326-mimetizador aumentaram ainda mais a expressão de miR-326 em células HepG2, enquanto macrófagos M1 transfectados com inibidor de miR-326 exossomos derivados degradaram a expressão de miR-326 em células SSMC-7721 (Fig. 2g, h). É sugerido que exossomos derivados de macrófagos M1 entreguem miR-326 para células HCC e afetem a expressão de miR-326 em células HCC.
MiR-326 exossômico derivado de macrófago M1 reduz a proliferação celular e a capacidade de formação de colônias em células HCC
Ao explorar o efeito do miR-326 exossômico na proliferação de células HCC, ensaios de MTT e de formação de colônias foram conduzidos para examinar a proliferação de células HCC. Foi sugerido que em células HepG2, a restauração de miR-326 prejudicou a proliferação celular e a capacidade de formação de colônias (Fig. 3a, e, f). Os exossomos derivados de macrófagos M1 obstruíram as células HepG2 para proliferar e formar colônias. Os exossomos derivados de macrófagos M1 transfectados por mimetização MiR-326 prejudicaram ainda mais a proliferação celular e a capacidade de formação de colônias (Fig. 3c, i, j).
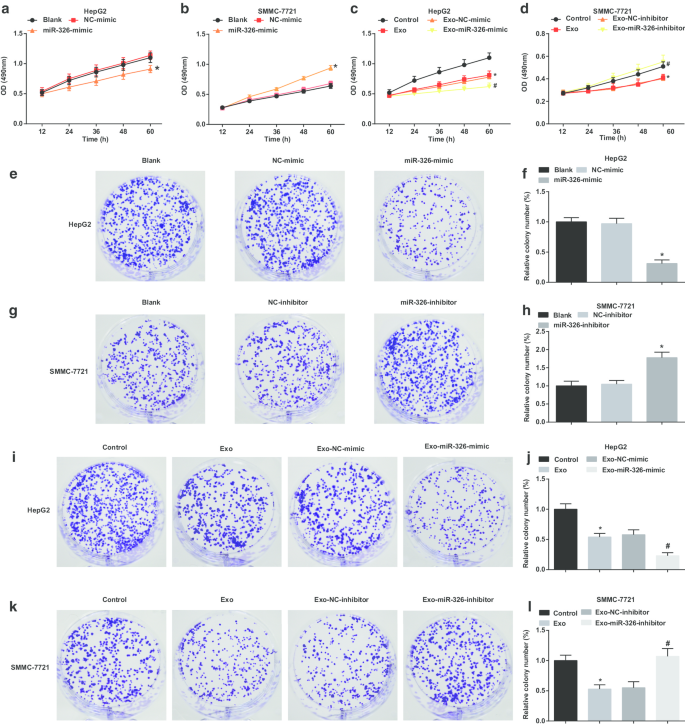
O miR-326 exossômico do macrófago M1 inibe a proliferação celular e a formação de colônias em células HCC. a O ensaio de MTT detectou o efeito da transfecção de miR-326 mimetizador na proliferação de células HepG2. b O ensaio de MTT detectou o efeito da transfecção do inibidor miR-326 na proliferação de células SMMC-7721. c O ensaio de MTT detectou o efeito da co-cultura com exossomos derivados de macrófagos M1 transfectados com miR-326 na proliferação de células HepG2. d O ensaio de MTT detectou o efeito da co-cultura com exossomos derivados de macrófagos M1 transfectados com inibidor miR-326 na proliferação de células SMMC-7721. e O ensaio de formação de colônias detectou o efeito da transfecção de miR-326 mimetizador na capacidade de formação de colônias de células HepG2. f Número de colônias de células HepG2. g O ensaio de formação de colônia detectou o efeito da transfecção do inibidor miR-326 na capacidade de formação de colônia das células SMMC-7721. h Número de colônias de células SMMC-7721. eu O ensaio de formação de colônias detectou o efeito da co-cultura com exossomos derivados de macrófagos M1 transfectados com miR-326 na capacidade de formação de colônias de células HepG2. j Número de colônias de células HepG2 tratadas com exossomos. k O ensaio de formação de colônia detectou o efeito da co-cultura de exossomos derivados de macrófagos M1 transfectados com inibidor miR-326 na capacidade de formação de colônias de células SMMC-7721. l Número de colônias de células SMMC-7721 tratadas com exossomos. No painel a e f, * P <0,05 versus o grupo NC-mimetizador; No painel b e h, * P <0,05 versus o grupo de inibidor de NC; No painel c e j, * P <0,05 versus o grupo de controle, # P <0,05 versus o grupo Exo-NC-mimetizador; No painel d e l, * P <0,05 versus o grupo de controle, # P <0,05 versus o grupo de inibidores Exo-NC. Os dados de medição foram representados como média ± desvio padrão ( N =3), as comparações entre vários grupos foram conduzidas por uma análise de variância unilateral
Em células SMMC-7721, miR-326 knockdown aumentou a proliferação celular e a capacidade de formação de colônias (Fig. 3b, g, h). Em células SMMC-7721 tratadas com exossomos derivados de macrófagos M1, a proliferação celular e a capacidade de formação de colônias foram diminuídas. Os exossomos derivados de macrófagos M1 transfectados com inibidor MiR-326 estimularam ainda mais a proliferação celular e a capacidade de formação de colônias (Fig. 3d, k, l). É sugerido que miR-326 derivado de exossomos derivados de macrófagos M1 impede a proliferação de células HCC.
M1 miR-326 exossômico derivado de macrófago suprime migração e invasão de células HCC
Em seguida, o efeito do miR-326 exossômico na invasão e migração de células HCC foi examinado. Foi demonstrado que em células HepG2, a restauração da invasão e migração do miR-326 restringiu (Fig. 4a-c). Os exossomos derivados de macrófagos M1 perturbaram as células HepG2 para invadir e migrar. A invasão e a migração degradaram-se ainda mais quando as células HepG2 foram co-cultivadas com exossomos derivados de macrófagos M1 transfectados com miR-326-mimetizador (Fig. 4g-i).
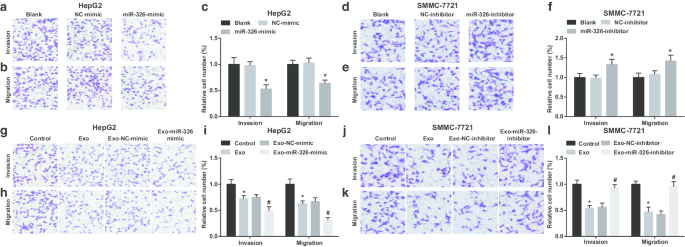
O miR-326 exossômico do macrófago M1 inibe a migração e a invasão das células HCC. a O ensaio Transwell detectou o efeito da transfecção de miR-326 mimetizador na invasão de células HepG2. b O ensaio Transwell detectou o efeito da transfecção de miR-326 imitando a migração de células HepG2. c O número de invasão e migração de células HepG2. d O ensaio Transwell detectou o efeito da transfecção do inibidor miR-326 na invasão de células SMMC-7721. e O ensaio Transwell detectou o efeito da transfecção do inibidor miR-326 na migração de células SMMC-7721. f O número de invasão e migração de células SMMC-7721. g O ensaio Transwell detectou o efeito da co-cultura com exossomos derivados de macrófagos M1 transfectados com miR-326 na invasão de células HepG2. h O ensaio Transwell detectou o efeito da co-cultura com exossomos derivados de macrófagos M1 transfectados com miR-326 na migração de células HepG2. eu O número de invasão e migração de células HepG2 co-cultivadas com exossomos derivados de macrófagos M1. j O ensaio Transwell detectou o efeito da co-cultura com exossomos derivados de macrófagos M1 transfectados com inibidor miR-326 na invasão de células SMMC-7721. k O ensaio Transwell detectou o efeito da co-cultura com exossomos derivados de macrófagos M1 transfectados com inibidor miR-326 na migração de células SMMC-7721. l O número de invasão e migração de células SMMC-7721 co-cultivadas com exossomos derivados de macrófagos M1. No painel c, * P <0,05 versus o grupo NC-mimetizador; No painel f, * P <0,05 versus o grupo de inibidor de NC; No painel i, * P <0,05 versus o grupo de controle, # P <0,05 versus o grupo Exo-NC-mimetizador; No painel l, * P <0,05 versus o grupo de controle, # P <0,05 versus o grupo de inibidores Exo-NC. Os dados de medição foram descritos como média ± desvio padrão ( N =3), as comparações entre vários grupos foram conduzidas por uma análise de variância unilateral
O knockdown do MiR-326 resultou em melhorias na invasão e migração de células SMMC-7721 (Fig. 4d-f). Quando tratadas com exossomos derivados de macrófagos M1, células SMMC-7721 foram exibidas com invasão e migração diminuídas. However, SMMC-7721 cell invasion and migration were boosted upon co-culture with miR-326-inhibitor-transfected M1 macrophages-derived exosomes (Fig. 4j–l). It is implied that miR-326 derived from M1 macrophage exosomes impedes the invasion and migration of HCC cells.
M1 Macrophage-Derived Exosomal miR-326 Promotes Apoptosis of HCC Cells
When examining the effect of exosomal miR-326 on the cell cycle and apoptosis of HCC cells, PI single staining and Annexin V-FITC/PI double staining were applied. It was illustrated that miR-326 overexpression increased cells arrested at G0/G1 phase, reduced cells arrested at S and G2/M phases and raised apoptosis in HepG2 cells (Fig. 5a–d). Co-culturing with M1 macrophages-derived exosomes increased cells arrested at G0/G1 phase, reduced cells arrested at S and G2/M phases and raised cell apoptosis of HepG2 cells. Co-cultivation with exosomes from M1 macrophages transfected with miR-326-mimic further enhanced these effects (Fig. 5i–l).
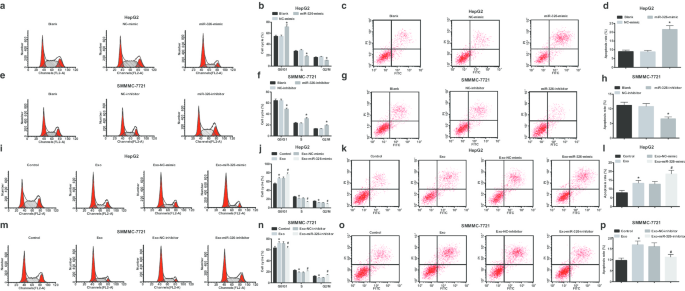
M1 macrophage exosomal miR-326 promotes apoptosis of HCC cells. a , b Flow cytometry detected the effect of transfection of miR-326 mimic on HepG2 cell cycle; c , d flow cytometry detected the effect of transfection of miR-326 mimic on HepG2 cell apoptosis; e , f flow cytometry detected the effect of transfection of miR-326 inhibitor on SMMC-7721 cell cycle; g , h flow cytometry detected the effect of transfection of miR-326 inhibitor on SMMC-7721 cell apoptosis; eu , j flow cytometry detected the effects of co-culture of with miR-326 mimic-transfected M1 macrophage-derived exosomes on HepG2 cell cycle; k , l flow cytometry detected the effect of co-culture with miR-326 mimic-transfected M1 macrophage-derived exosomes on HepG2 cell apoptosis; m , n flow cytometry detected the effect of co-culture with miR-326 inhibitor-transfected M1 macrophage-derived exosomes on SMMC-7721 cell cycle; o , p , flow cytometry detected the effect of co-culture with miR-326 inhibitor-transfected M1 macrophage-derived exosomes on SMMC-7721 cell apoptosis. In panel b and d, *P <0.05 versus the NC-mimic group; In panel f and h, *P <0.05 versus the NC-inhibitor group; In panel j and l, *P <0.05 versus the control group, #P <0.05 versus the Exo-NC-mimic group; In panel n and p, *P <0.05 versus the controlgroup, #P <0.05 versus the Exo-NC-inhibitor group. Measurement data were depicted as mean ± standard deviation (N = 3), comparisons among multiple groups were conducted by one-way analysis of variance
In SMMC-7721 cells, miR-326 down-regulation reduced cells arrested at G0/G1 phase, elevated cells arrested at S and G2/M phases, and declined cell apoptosis (Fig. 5e–h). Untransfected M1 macrophages-derived exosomes increased cells arrested at G0/G1 phase, reduced cells arrested at S and G2/M phases, and heightened cell apoptosis. MiR-326-inhibitor-transfected M1 macrophages-derived exosomes degraded cells arrested at G0/G1 phase, elevated cells arrested at S and G2/M phases, and decreased cell apoptosis (Fig. 5m–p). Briefly, it is summarized that miR-326 derived from M1 macrophage exosomes arrests cell cycle in G0/G1 phase and induces cell apoptosis in HCC.
M1 Macrophage-Derived Exosomal miR-326 Declines CD206 and NF-κB Expression in HCC Cells
Next, the potential mechanism of miR-326 derived from M1 macrophage exosomes in the biological progress of HCC cells was explored. NF-κB is the key link between inflammation and cancer. Many regulatory proteins and miRNAs could inhibit the excessively activated NF-κB signaling to suppress cancer [16]. Such a beneficial effect may include the polarization of M2 macrophages into M1 macrophages. CD206 and NF-κB expression in HepG2 and SMMC-7721 cells was tested by RT-qPCR. It was suggested that miR-326 restoration decreased CD206 and NF-κB expression in HepG2 cells, while miR-326 knockdown enhanced CD206 and NF-κB expression in SMMC-7721 cells (Fig. 6a, b). Moreover, co-culture with M1 macrophage exosomes significantly reduced CD206 and NF-κB expression in HepG2 cells, while co-culture with M1 macrophage exosomes-overexpressing miR-326 further decreased CD206 and NF-κB expression. Treated with untransfected M1 macrophages-derived exosomes, CD206 and NF-κB expression was decreased in SMMC-7721 cells. Co-cultured with miR-326-inhibitor-transfected M1 macrophages-derived exosomes, SMMC-7721 cells were featured by heightened CD206 and NF-κB expression (Fig. 6c, d). It was concluded that miR-326 from M1 macrophage exosomes played a tumor suppressor by inhibiting NF-κB in HCC cells.
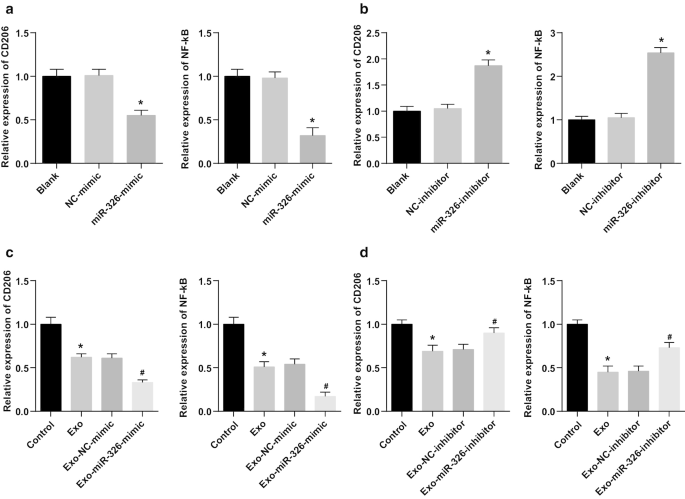
M1 macrophage exosomal miR-326 declines CD206 and NF-κB expression in HCC cells. a RT-qPCR detected the effect of transfection of miR-326 mimic on the expression of CD206 and NF-κB in HepG2 cells; b RT-qPCR detected the effect of transfection of miR-326 inhibitor on the expression of CD206 and NF-κB in SMMC-7721 cells. c RT-qPCR detected the effect of co-culture with miR-326 mimic-transfected M1 macrophage-derived exosomes on the expression of CD206 and NF-κB in HepG2 cells; d RT-qPCR detected the effect of co-culture with miR-326 inhibitor-transfected M1 macrophage-derived exosomes on the expression of CD206 and NF-κB in SMMC-7721 cells. In panel a, *P <0.05 versus the NC-mimic group; In panel b, *P <0.05 versus the NC-inhibitor group; In panel c, *P <0.05 versus the control group, #P <0.05 versus the Exo-NC-mimic group; In panel d, *P <0.05 versus the control group, #P <0.05 versus the Exo-NC-inhibitor group. Measurement data were depicted as mean ± standard deviation (N = 3), comparisons among multiple groups were conducted by one-way analysis of variance
miR-326 from M1 Macrophage Exosomes Inhibits HCC Tumor Growth In Vivo
Finally, the in vivo results were validated through tumor xenografts. As displayed, miR-326 overexpression decreased volume and weight of tumors in HepG2 cells (Fig. 7a–c). In mice transplanted with HepG2 cells co-cultured with exosomes, the treatment with M1 macrophage exosomes significantly reduced the tumor volume and weight of HepG2 cells, while treatment with M1 macrophages-overexpressing miR-326 further reduced the tumor volume and weight of HepG2 cells (Fig. 7g–i).
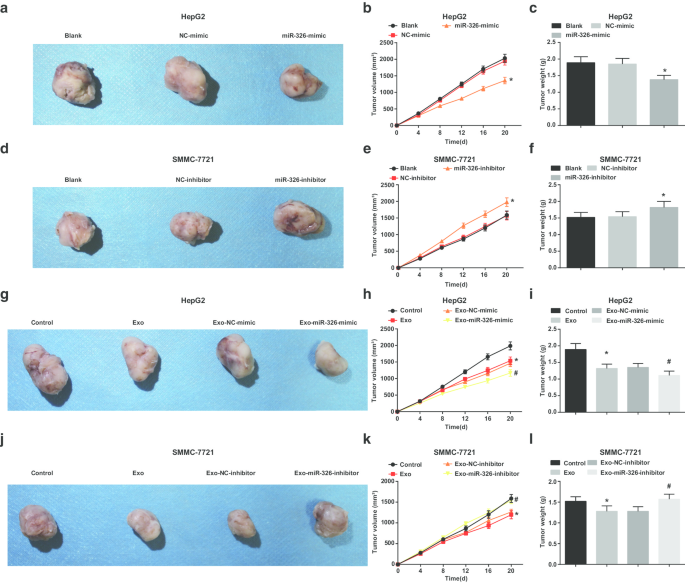
M1 macrophage exosomal miR-326 reduces the volume and weight of HCC tumor in vivo. a - c The effect of transfection of miR-326 mimic on the tumor volume and tumor of nude mice xenografted with HepG2 cells. d - f The effect of transfection of miR-326 inhibitor on the tumor volume and tumors of nude mice xenografted with SMMC-7721 cells. g - i The effect of co-culture with miR-326 mimic-transfected M1 macrophage-derived exosomes on the tumor volume and tumor of nude mice xenografted with HepG2 cells. j - l The effect of co-culture with miR-326 inhibitor-transfected M1 macrophage-derived exosomes on the tumor volume and tumor of nude mice xenografted with SMMC-7721 cells. In panel b and c, *P <0.05 versus the NC-mimic group; In panel e and f, *P <0.05 versus the NC-inhibitor group; In panel h and i, *P <0.05 versus the control group, #P <0.05 versus the Exo-NC-mimic group; In panel k and l, *P <0.05 versus the control group, #P <0.05 versus the Exo-NC-inhibitor group. Measurement data were depicted as mean ± standard deviation (n = 5), comparisons among multiple groups were conducted by one-way analysis of variance
In SMMC-7721 cells, miR-326 suppression increased volume and weight of tumors (Fig. 7d–f). Untransfected M1 macrophages-derived exosomes obstructed tumor growth in volume and weight of SMMC-7721 cells. Co-cultured with miR-326 inhibitor-transfected M1 macrophages-derived exosomes, SMMC-7721 cells were injected into mice and caused elevations in tumor volume and weight (Fig. 7j–l). Summarily, miR-326 derived from M1 macrophage exosomes depressed tumor growth of HCC in vivo.
Discussão
HCC is a common cancer that is characterized with high morbidity and mortality, difficult early diagnosis and treatment, poor prognosis and 5-year survival rate [17]. Recently, a study has highlighted that the lowly expressed lncRNA cox-2 declines the ability of M1 macrophages to suppress HCC cell growth, invasiveness, angiogenesis migration and promote apoptosis [18]. Hu et al. have discussed that miR-326 is obviously degraded in HCC tissues and cell lines, while down-regulated miR-326 is connected to the TNM stage, lymph node metastasis and differentiation of HCC patients [14]. It is customarily considered that HCC cells-derived exosomes can form a fertile environment to facilitate HCC cells growth, invasiveness and metastasis as well as development of drug resistance [19]. The current study was designed to explore the mechanism of exosomal miR-326 in regulating invasion and migration of HCC cells.
Our results indicated that miR-326 expression decreased in HCC cells but increased in exosomes. A recent study has pointed out that miR-326 expression is declined in HCC tissues [20]. Another study has presented miR-326 expression is notably reduced in HCC cell lines and tissues and its down-regulation predicts a poor prognosis in HCC [13]. It is reported that miR-326 acts a tumor-suppression role and is greatly depressed in HCC cells [21]. All these aforementioned evidences are in line with our findings. A study has purported that in comparison with the controls, miR-326 expression is raised in Tconv-derived exosomes which is observed in relapsing–remitting multiple sclerosis patients [11].
Other results emerge from our data that highly expressed exosomal miR-326 reduced cell proliferation, colony formation, migration and invasion as well as facilitated apoptosis of HCC cells in vitro and reduced the volume and weight of HCC tumor in vivo. It has been suggested previously that HCC cell growth can be suppressed via overexpression of miR-326, and HCC cell migration and invasion ability are markedly attenuated through elevating miR-326 [21]. It is reported that the up-regulated miR-326 expression suppresses HCC cell growth and invasiveness as well as stimulates cell apoptosis in vitro [14]. Besides that, a prior study has verified that overexpression of miR-326 declined tumor growth in vivo [13]. A study has revealed that ectopic expressed miR-326 markedly attenuates cell growth, and suppresses cellular migration and invasiveness in non-small cell lung cancer cell lines [22]. Moreover, it is found that miR-326 decreases profibrotic genes like MMP-9, implying its repressive function in cancer cell proliferation [23]. Also, it is presented that miR-326 represses Bcl-2 protein expression and elevates Bax expression so as to affect the apoptosis [24]. Similar to our findings, there are some miRNAs interacting with exosomes to play a role in HCC development. It is displayed that highly expressed exosomal miR-638 can repress the proliferation of HCC cells, involving the potential impact on carcinogenesis [25]. Another study also proves that HCC cells-derived exosomal miR-451a suppresses tumor angiogenesis via disrupting endothelial functions as apoptosis, tube formation, migration and permeability [26]. A prior research generally confirms that when treated with the overexpression of miR-744 exosomes, the proliferation of HCC cells is dramatically suppressed [27].
Conclusão
To briefly conclude, our study provides evidence that M1 macrophage-derived exosomal miR-326 suppresses proliferation, migration and invasion as well as advances apoptosis of HCC cells, supplying a new insight in a novel target therapy for HCC. Due to the limited sample size and limited known researches, the exact mechanism of miR-326 is not fully elucidated, and therefore, further large-scale studies are required to illustrate the underlying mechanism.
Abreviações
- HCC:
-
Hepatocellular carcinoma
- miRNAs:
-
MicroRNAs
- FBS:
-
Soro fetal bovino
- M/CSF:
-
Macrophage colony-stimulating factor
- PBS:
-
Salina tamponada com fosfato
- DMEM:
-
Dulbecco’s modified eagle medium
- PPAR:
-
Peroxisome proliferator-activated receptor
- TEM:
-
Microscopia eletrônica de transmissão
- NTA:
-
Nanoparticle tracking analysis
- NC:
-
Negative control
- RT-qPCR:
-
Reverse transcription quantitative polymerase chain reaction
- BCA:
-
Bicinchoninic acid
- BSA:
-
Albumina sérica bovina
- Exo:
-
Exosomes
- ANOVA:
-
Análise de variância unilateral
Nanomateriais
- Controle de Polarização Terahertz Completo com Largura de Banda Ampliada via Metasurfaces Dielétricos
- Estudo in vitro da influência das nanopartículas de Au nas linhas celulares HT29 e SPEV
- Efeito do eletrodo ITO pulverizado em ângulo oblíquo nas estruturas de células solares de MAPbI3 perovskita
- Fabricação e caracterização de novo composto de suporte de catalisador anódico de nanofibra de carbono Tio2 para célula de combustível de metanol direto via método de eletrofiação
- Eficácia antitumoral aprimorada e farmacocinética de Bufalin via lipossomas PEGuilados
- Ajuste do modo de movimento deslizante de nanotubos de carbono por meio de grupos hidroxila
- Avaliação da atividade do citocromo P450 3A4 inibido por nanopartículas de ouro e dos mecanismos moleculares subjacentes à sua toxicidade celular na linha celular C3A do carcinoma hepatocelular hu…
- Minúsculas nanopartículas de fluoreto de terras raras ativam o crescimento de células tumorais por meio de interações polares elétricas
- Mecanismo de falha de troca em célula de metalização programável baseada em peróxido de zinco
- Inibidor de autofagia (LY294002) e 5-fluorouracil (5-FU) Nanolipossoma baseado em combinação para eficácia aprimorada contra carcinoma de células escamosas de esôfago



