microRNA-18a de macrófagos M2 inibe TGFBR3 para promover a progressão do carcinoma nasofaríngeo e o crescimento do tumor via via de sinalização de TGF-β
Resumo
Objetivos
O carcinoma nasofaríngeo (NPC) é um tipo de doença nasofaríngea com altas propriedades de metástase e invasão. Macrófagos ativados alternativos associados a tumor (M2) são evidenciados para se conectar com NPC. Com base nisso, o objetivo deste estudo é explorar o mecanismo e a participação do microRNA-18a (miR-18a) de macrófagos M2 em NPC.
Métodos
As células mononucleares do sangue periférico foram diferenciadas em macrófagos e os macrófagos foram polarizados para o tipo M2 pela interleucina-4. As células SUNE-1 e CNE2 foram transfectadas com miR-18a restaurado ou esgotado ou receptor do fator de crescimento de transformação beta III (TGFBR3) para explorar seus papéis na progressão de NPC com o envolvimento da via de sinalização de TGF-β. Em seguida, as células SUNE-1 e CNE2 foram co-cultivadas com macrófagos M2 que foram tratados com miR-18a ou TGFBR3 restaurado ou esgotado para compreender seus papéis combinados em NPC com o envolvimento da via de sinalização de TGF-β.
Resultados
MiR-18a foi altamente expresso e TGFBR3 foi pouco expresso em células NPC. Restauração de MiR-18a, knockdown de TGFBR3 ou co-cultura com mimetizadores de miR-18a ou macrófagos M2 transfectados com si-TGFBR3 promoveram a progressão de células SUNE-1, crescimento de tumor em camundongos, diminuição de p-Smad1 / t-Smad1 e aumento de p- Smad3 / t-Smad3. A regulação negativa de miR-18a, a superexpressão de TGFBR3 ou a co-cultura com inibidores de miR-18a ou macrófagos M2 transfectados com OE-TGFBR3 deprimiram a progressão de células CNE2, crescimento de tumor em camundongos, aumento de p-Smad1 / t-Smad1 e diminuição de p-Smad3 / t-Smad3.
Conclusão
Nosso estudo elucida que miR-18a de macrófagos M2 resulta em progressão celular NPC promovida e crescimento tumoral em camundongos nus via repressão de TGFBR3, juntamente com a inativação de Smad1 e ativação de Smad3.
Introdução
O carcinoma nasofaríngeo (NPC) é um tumor maligno epitelial que tende a infiltração local e metástase à distância precoce [1]. Pacientes com NPC frequentemente se queixam de paralisia do sexto nervo e síndrome de Horner [2]. Atualmente, o tratamento aplicado consiste predominantemente em radioterapia, e radioterapia e quimioterapia integradas [3]. Infelizmente, a radioterapia e a quimioterapia são inesperadamente acompanhadas por complicações, e a resistência adquirida à radioterapia dificulta os resultados da NPC [3]. Dado que a tarefa de investigar uma terapia potencial direcionada é uma prioridade.
MicroRNAs desregulados (miRNAs) estão documentados como estando envolvidos na tumorigênese, metástase, invasão e resistência à radioterapia e quimioterapia de NPC [1]. Como uma subfamília de miRNAs, miR-18a é encontrado para facilitar a progressão de NPC através do supressor de morfogênese na inibição da genitália 1 e ativação da via mTOR [4]. Além disso, miR-18a é ainda verificado para ativar a proliferação e metástase de células NPC via regulação DICER1 [5]. Além disso, evidencia-se que a progressão das células NPC é conduzida por miR-18a por meio do comprometimento da biogênese do miRNA [6]. Além disso, foi documentado que miR-18a funciona criticamente na metástase de NPC [7]. Macrófagos ativados alternativos (M2) são componentes importantes de malignidades sólidas e hematológicas e estão relacionados com progressão, metástase e resistência à terapia [8]. Macrófagos associados a tumor polarizados com M2 estão associados a mau prognóstico de NPC [9]. É intrigantemente registrado as diferenças de macrófagos M2 em NPC negativo para o vírus Epstein-Barr e positivo para o vírus Epstein-Barr [10]. Há um estudo delineando que miR-18a inibe a metástase hepática de células de câncer de cólon induzindo macrófagos M1 [11]. O receptor do fator de crescimento beta III transformador (TGFBR3) é um co-receptor TGF-β que fornece o ligante do receptor TGF-β tipo II para estimular a sinalização e o equilíbrio da superfície celular, e o TGFBR3 solúvel é um regulador durante a progressão do câncer [12]. Foi relatado que o TGFBR3 pouco expresso induz um microambiente tumoral imunotolerante [13]. Ao contrário, a superexpressão transitória de TGFBR3 induz apoptose em células NPC humanas [14]. Até onde sabemos, a polarização de macrófagos M2 induzida por miR-181a avança a metástase de células tumorais mediadas por macrófagos M2 através do fator 6 semelhante a Kruppel e do eixo α de proteína de ligação CCAAT / potenciador [15].
Coletivamente, embora muitos estudos tenham descoberto o papel independente dos macrófagos miR-18a, TGFBR3 e M2 em NPC, as interações combinadas entre esses três fatores ainda são indescritíveis. Considerando que este estudo é lançado para decifrar os mecanismos e a participação desses fatores no NPC.
Materiais e métodos
Declaração de Ética
O experimento foi aprovado pelo comitê de ética do Third Xiangya Hospital, Central South University e atendeu aos padrões de ética médica. Este estudo foi realizado com consentimento por escrito de todos os doadores. Os experimentos com animais estavam em conformidade com os requisitos dos regulamentos de Manejo e Uso de Animais de Laboratório Nacional.
Coleção de células mononucleares do sangue periférico
Os monócitos foram obtidos de sangue periférico de doadores saudáveis por método aderente. Amostras de sangue periférico foram obtidas de doadores saudáveis do Departamento de Hematologia do Hospital Terceiro Xiangya, Central South University. Macrófagos derivados de monócitos foram obtidos por aderência plástica de células mononucleares do sangue periférico (PBMCs) previamente isoladas por centrifugação em gradiente de densidade (Ficoll-Paque, GE Healthcare) a partir de preparações de camada leucocitária de sangue de doadores saudáveis. Então, 2,0 × 10 6 PBMCs foram cultivados em meio Eagle modificado por Dulbecco (DMEM) suplementado com soro humano a 10% (Millipore, Bedford, MA, EUA) e penicilina / estreptomicina em placas de 12 poços (NalgeNunc, NY, EUA). Quando os PBMCs aderiram à parede por 2–3 h, o sobrenadante e os PBMCs suspensos foram removidos repetidamente para obter monócitos aderentes.
Polarização de macrófagos
Os monócitos foram induzidos a se diferenciar em macrófagos pelo fator estimulador de colônias de macrófagos humanos (M-CSF) e polarizados em macrófagos M2 pela interleucina (IL) -4.
Indução de macrófagos
Os monócitos foram cultivados em 20% de soro fetal bovino (FBS) -DMEM e adicionado com M-CSF (100 μg, Peprotech, NJ, EUA) até a concentração final de 100 ng / mL. O meio foi renovado pela metade a cada 3 dias e então suplementado com 100 ng / mL de M-CSF. Cultivadas até o 7º dia, parte das células foram colhidas e os marcadores de superfície de macrófagos CD68 [16], CD163 [17] e CD206 [18] foram testados por ensaio de imunofluorescência [19] para identificar macrófagos.
Polarização de macrófagos
Os macrófagos foram polarizados para macrófagos M2 através da suplementação de 20 ng / mL de IL-4 (Peprotech) ao meio de diferenciação por mais 24 h. Uma parte das amostras de macrófagos M2 foram utilizadas para detecção de citometria de fluxo. As amostras foram divididas em 3 tubos:o tubo 1 era o mesmo lote de macrófagos aderentes sem estimulação com IL-4; os tubos 2 e 3 eram os macrófagos aderentes estimulados por IL-4. As amostras foram diluídas para cerca de 10.000 células durante o carregamento e adicionadas com imunoglobulina G não específica para bloquear o receptor Fc. Em seguida, as amostras foram adicionadas com anticorpo de isotipo não específico, anticorpo CD68 marcado com PE e anticorpo CD163 marcado com PE (ambos de Biolegend, CA, EUA). Incubadas por 30 min e lavadas com 0,5% de albumina de soro bovino em solução salina tamponada com fosfato (PBS), as amostras foram centrifugadas e colocadas em suspensão celular com 500 μL de PBS para detecção.
Reação em cadeia da polimerase quantitativa com transcrição reversa
A reação em cadeia da polimerase quantitativa de transcrição reversa (RT-qPCR) foi empregada para detectar miR-18a, CCL22, receptor γ ativado por proliferador de peroxissoma (PPAR-γ) e expressão de mRNA de TGFBR3 nas células coletadas.
O RNA total foi extraído das células por Trizol (Invitrogen, Carlsbad, CA, EUA) e reversamente transcrito em RNA complementar pelo kit Mir-X miRNA First Strand Synthesis (Clontech, Mountain View, CA, EUA) para miR-18a e o PrimeScriptTM RT Master Mix Kit (Takara, Dalian, China) para CCL22, PPAR-γ e TGFBR3. U6 e gliceraldeído-3-fosfato desidrogenase (GAPDH) foram controles de carga para miR-18a, CCL22, PPAR-γ e TGFBR3. SYBR @ Premix Ex Taq ™ II (Perfect Real Time) (Takara) em um sistema LightCycler 480 II (Roche Diagnostics, Indiana, EUA) foi utilizado em PCR. O cálculo dos dados foi avaliado por 2 - △△ CT método. Os primers de PCR foram apresentados na Tabela 1.
Ensaio de Western Blot
O ensaio de Western blot foi aplicado à detecção de proteína TGFBR3, total (t) -Smad1, fosforilada (p) -Smad1, t-Smad3 e p-Smad3 nas células coletadas.
A proteína total das células foi extraída e a concentração de proteína determinada com base no kit de ácido bicinconínico. A amostra de proteína foi carregada nos poços em eletroforese em gel de poliacrilamida de dodecilsulfato de sódio e transferida para uma membrana de fluoreto de polivinilideno (PVDF). A membrana de PVDF foi bloqueada com leite desnatado e incubada com anticorpos primários TGFBR3 (1:2000, R&D Systems, Minneapolis, MN, EUA), t-Smad1 (1:1000), p-Smad1 (1:1000, Santa Cruz Biotechnology) , t-Smad3 (1:1000), p-Smad3 (1:1000) e GAPDH (1:1000, todos de Abcam, Cambridge, MA, UK) que foi seguido por incubação com o anticorpo secundário marcado com peroxidase de rábano ( 1:500, Jackson ImmunoResearch Laboratories, PA, EUA). Lavada 3 vezes por solução salina tamponada com tris com Tween 20, a membrana foi desenvolvida por quimiluminescência aprimorada. A quantificação de sinais foi concluída pelo National Institutes of Health ImageJ Imaging. Software de análise de processamento com intensidade de sinalização normalizada para GAPDH.
Cultura de células e triagem
As linhas celulares NPC humanas CNE2, TW03, C666-1 e SUNE-1 e a linha celular nasofaríngea humana normal NP96 (Shanghai Institutes for Biological Sciences, Chinese Academy of Sciences, Shanghai, China) foram cultivadas no Roswell Park Memorial Institute (RPMI) - Meio 1640 (Gibco, CA, EUA) contendo FBS (Gibco), 100 μg / mL de penicilina e 100 μg / mL de estreptomicina e passado em 80% de confluência. RT-qPCR foi utilizado para detectar a expressão de miR-18a. Entre essas linhas de células NPC, as células CNE2 e SUNE-1 mostraram a maior e a menor diferença na expressão de miR-18a a partir de células NP96, portanto, foram selecionadas para ensaios de regulação negativa ou regulação positiva de miR-18a.
Agrupamento e tratamento de células
Entre todas as linhas de células NPC, as células SUNE-1 com a menor diferença das células NP96 na expressão de miR-18a foram selecionadas. Guiado pelas especificações da Lipofectamine 2000 (Invitrogen), as células SUNE-1 foram transfectadas com mimetizadores de miR-18a, miR-18a mimetiza o controle negativo (NC), si-TGFBR3 ou si-TGFBR3 NC.
Entre todas as linhas de células NPC, as células CNE2 com a maior diferença das células NP96 na expressão de miR-18a foram selecionadas e transfectadas com inibidores de miR-18a, inibidores de miR-18a NC, superexpressão (OE) -TGFBR3 ou OE-TGFBR3 NC por Lipofectamine 2000 (Invitrogen).
Guiados pelas especificações de Lipofectamine 2000 (Invitrogen), macrófagos M2 foram transfectados com mímicos de miR-18a, mímicos de miR-18a NC, si-TGFBR3, si-TGFBR3 NC, inibidores de miR-18a, inibidores de miR-18a NC, OE-TGFBR3, ou OE-TGFBR3 NC.
Co-cultura de macrófagos M2 e células NPC
A co-cultura celular na câmara Transwell foi adotada para explorar os efeitos do miRNA do macrófago M2 nas células NPC. A câmara superior foi preenchida com macrófagos M2 com tamanho de poro de 0,4 μm, o que apenas impediu a passagem das células da câmara superior, mas não as pequenas moléculas secretadas pelas células, como vesículas, fatores de crescimento, nutrientes, etc. A câmara inferior foi espalhado com células NPC.
SUNE-1 e CNE2 foram incubados em FBS normal (Gibco). As células em fase de crescimento logarítmico foram adotadas para os experimentos.
As células SUNE-1 e CNE2 foram co-cultivadas com macrófagos M2 em meio FBS-RPMI-1640 a 10% (ambos da Gibco) em uma placa de cultura de células de inserção Transwell (Coring, Corning, NY, EUA) com um tamanho de poro de 0,4 μm .
As células SUNE-1 não foram co-cultivadas com macrófagos M2, ou co-cultivadas com macrófagos M2, macrófagos M2 transfectados mimetizando miR-18a, miR-18a mimetizando macrófagos M2 transfectados com NC, macrófagos M2 transfectados com si-TGFBR3 ou si Macrófagos M2 transfectados com TGFBR3 NC.
As células CNE2 não foram co-cultivadas com macrófagos M2 ou co-cultivadas com macrófagos M2, macrófagos M2 transfectados com inibidores miR-18a, inibidores miR-18a macrófagos M2 transfectados com NC, macrófagos M2 transfectados com OE-TGFBR3 ou OE-TGFBR3 Macrófagos M2 transfectados com NC.
Ensaio de brometo de 3- (4, 5-Dimetiltiazol-2-il) -2,5-difeniltetrazólio
A viabilidade celular foi testada pelo ensaio de 3- (4, 5-dimetiltiazol-2-il) -2, 5-difeniltetrazólio (MTT), que foi um ensaio colorimétrico aplicado para determinar a atividade da desidrogenase mitocondrial, que reduziu o MTT a formazan.
Trypsinized e semeado em placas de 96 poços em 4 × 10 4 células / poço, as células foram privadas de meio de cultura na 0, 12ª, 24ª, 36ª e 48ª h, respectivamente, e suplementadas com solução de MTT (500 μL, 0,5 g / L). Incubado por 4 h, o sobrenadante foi descartado e as células foram incubadas com 200 μL de solução de dimetilsulfóxido. Os valores de densidade óptica (DO, 490 nm) foram medidos em um leitor de microplaca (ELX808IU, BioTek, VT, EUA). Cada grupo foi configurado com 6 poços paralelos.
Ensaio de formação de colônia
A capacidade de formação de colônias das células NPC foi testada pelo ensaio de formação de colônias, que refletiu a dependência da população celular e a proliferação clonal celular.
Cultivadas por 24 he destacadas com 0,25% de tripsina, 300 células foram semeadas em um prato de 35 mm com 3 poços paralelos em cada grupo. Com o meio de cultura renovado a cada 3 dias, as células foram cultivadas por uma semana e fixadas com 5 mL de paraformaldeído a 4%. Depois disso, as células foram coradas com solução de coloração de violeta de cristal e secas ao ar. A placa foi invertida na qual uma película transparente de grades foi sobreposta, e o número de colônias (mais de 50 células) foi contado ao microscópio (Olympus, Tóquio, Japão).
Teste Scratch
A migração celular foi testada por teste de arranhão. As células foram tripsinizadas, semeadas em placas de 6 poços com 3 poços paralelos para cada grupo e cultivadas até 90% de confluência. Em seguida, as células foram incubadas no meio com FBS a 2% e arranhões verticais foram desenhados por uma ponta de 100 μL. As células foram fotografadas nas 0 e 24 h em microscópio invertido para medir a distância de migração celular.
Ensaio Transwell
A invasão e migração de células foram testadas pelo ensaio Transwell. A câmara superior da câmara Transwell foi pré-imersa e adicionada com 100 μL de Matrigel (Coring) que foi diluído em meio RPMI 1640 sem soro a 1:100. As câmaras superior e inferior foram adicionadas com 200 μL e 600 μL de meio RPMI 1640 sem soro separadamente. Posteriormente, a câmara inferior foi adicionada com 600 μL de meio RPMI 1640 contendo 10% de FBS, enquanto a câmara superior com 200 μL de suspensão de células (12,5 × 10 4 células / mL). Incubadas por 40 h, as células foram coradas com solução de coloração com violeta cristal e esfregadas com cotonete para contagem das células que passavam pelo Matrigel ao microscópio.
Citometria de fluxo
A apoptose celular e a distribuição do ciclo celular foram determinadas por citometria de fluxo.
A distribuição do ciclo celular foi avaliada por coloração com iodeto de propídio (PI). As células foram semeadas em placas de 6 poços em 4 × 10 5 células / poço e cultivadas até 70-80% de confluência. Fixadas em etanol 70% pré-resfriado durante a noite, as células foram centrifugadas (o sobrenadante foi descartado), adicionadas com RNAase (1 g / L, 200 μL) e Triton X-100 (2 μL), e coradas por solução de coloração PI para 30 minutos. Em seguida, a distribuição do ciclo celular foi detectada por citômetro de fluxo (BD Bioscience, NJ, EUA) a 488 nm de acordo com diferentes intensidades de fluorescência celular em cada frase (frase G0 / G1, frase S e frase G2 / M).
A apoptose celular foi medida por isotiocianato de fluoresceína V-anexina (FITC) e coloração dupla com PI. As células foram ressuspensas em 500 μL de tampão de ligação e coradas por 5 μL de solução de coloração de Anexina V-FITC e 10 μL de solução de PI. A apoptose celular também foi testada por um citômetro de fluxo (BD Bioscience) em 30 minutos sem exposição à luz. No gráfico de dispersão, as células vivas no quadrante esquerdo inferior (Q4) foram FITC - / PI - , as células apoptóticas no estágio inicial no quadrante inferior direito (Q3) foram FITC + / PI - , e as células necróticas e apoptóticas no estágio tardio no quadrante superior direito (Q2) foram FITC + / PI + . Taxa de apoptose =porcentagem de apoptose precoce (Q3) + porcentagem de apoptose tardia (Q2).
Xenoenxertos de tumor em camundongos nus
O crescimento do tumor foi observado através do estabelecimento de um modelo de NPC em camundongos nus. Separadas por 0,25% de tripsina, as células SUNE-1 e CNE2 na fase logarítmica foram configuradas em suspensão de célula única em 5 × 10 7 células / mL. A suspensão de células (0,2 mL) foi injetada na axila direita de camundongos por um microinjetor para estabelecer modelos de camundongos. Os camundongos modelados foram criados em um ambiente livre de patógenos específicos. Iniciado no 4º dia, o crescimento do tumor foi observado e os camundongos foram pesados a cada 4 dias. Camundongos nus foram sacrificados no dia 20 após a injeção e os tumores foram ressecados, pesados em balança eletrônica e fotografados.
Ensaio do gene Dual Luciferase Reporter
O sistema de gene repórter de luciferase dual foi adotado para confirmar os locais de ligação de miR-18a e região 3′ não traduzida (UTR) do mRNA de TGFBR3. Um site de predição biológica (http://www.microrna.org/microrna/home.do) foi usado para analisar o gene alvo do miR-18a e descobrir a existência do sítio de ligação complementar do miR-18a no 3 ' UTR de TGFBR3. O ensaio do gene repórter de luciferase dupla foi empregado para verificar se o TGFBR3 era diretamente direcionado pelo miR-18a. Um pmirGLO-TGFBR3-tipo selvagem (WT) e um pmirG-LO-TGFBR3-tipo mutante (MUT) do sítio de ligação TGFBR3 3'UTR foram construídos. TGFBR3-WT ou TGFBR3-MUT e miR-18a mimetizadores ou mimetizadores NC foram co-transfectados em células SUNE-1 e CNE2 por Lipofectamine 2000 (Invitrogen) e incubados durante 48 h. Um kit de ensaio de luciferase (Promega, Madison, WI, EUA) foi utilizado para analisar células.
Análise estatística
O software estatístico SPSS21.0 (IBM Corp. Armonk, NY, EUA) foi usado para a análise. Os dados foram expressos como média ± desvio padrão. As diferenças entre dois grupos foram analisadas por t teste enquanto as diferenças entre vários grupos por análise de variância unilateral (ANOVA), seguido pelo teste post hoc de Tukey. Uma diferença significativa foi considerada em P <0,05.
Resultados
Identificação de Macrófagos M2
Os monócitos enriquecidos pelo método aderente e os monócitos induzidos por M-CSF foram coletados de sangue periférico de doadores saudáveis. A detecção por imunofluorescência de CD68, CD206 e CD163 confirmou que PBMCs induzidos por M-CSF in vitro se transformaram em macrófagos com características moleculares típicas, que atendiam aos nossos requisitos (Fig. 1a).
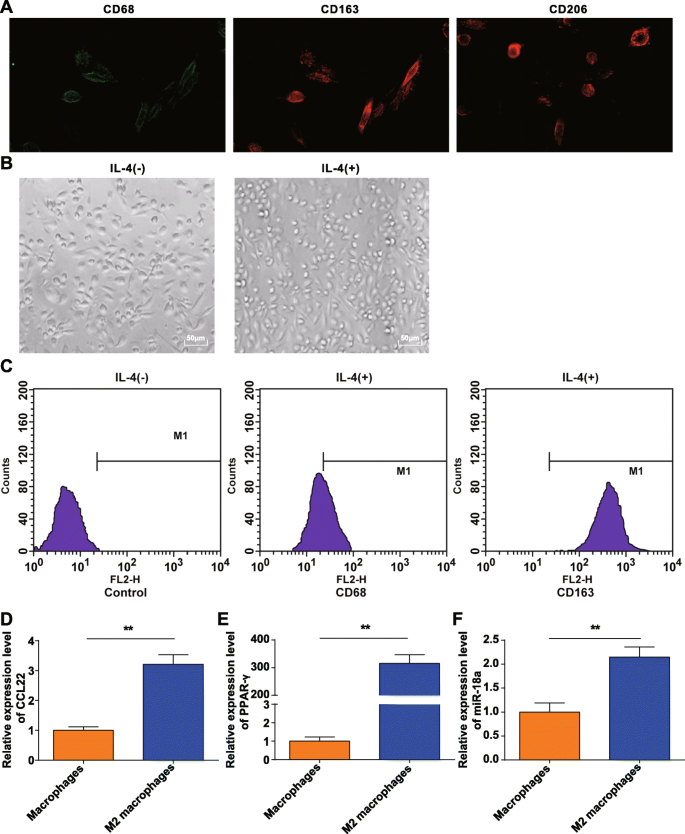
Identificação de macrófagos M2. a . CD68, CD206 e CD163 foram expressos na superfície de macrófagos obtidos após indução de monócitos in vitro. b Macrófagos polarizados por IL-4 eram macrófagos M2. c O CD163 foi altamente expresso e o CD68 foi pouco expresso em macrófagos M2 polarizados por IL-4. d CCL22 foi altamente expresso em macrófagos M2. e PPAR-γ foi altamente expresso em macrófagos M2. f miR-18a foi altamente expresso em macrófagos M2; * P <0,05; ** P <0,01. Os dados de medição foram expressos como média ± desvio padrão, N =3. As comparações entre dois grupos foram analisadas por t teste
Os macrófagos obtidos foram polarizados por IL-4 e observados ao microscópio para morfologia. Foi manifestado que os macrófagos (M0) sem estimulação com IL-4 eram diversos e irregulares, apresentando formato redondo, oval ou fusiforme. Estimulados pela IL-4, os macrófagos M2 tornaram-se maiores e predominantemente redondos, estando em conformidade com as características morfológicas dos macrófagos M2 descritas anteriormente [20] (Fig. 1b).
A citometria de fluxo testou os antígenos de superfície das células aderentes estimuladas por 20 ng / mL de IL-4 por 24 horas e descobriu que CD68 foi expresso a 21,16%, enquanto CD163 estava a 98,69% do número total de células (Fig. 1c), confirmando as células aderentes sendo macrófagos M2. RT-qPCR revelou que com células M0, ao contrário, CCL22 e PPAR-γ (moléculas de polarização típicas) aumentaram em macrófagos M2 (Fig. 1d, e), indicando a indução bem-sucedida de macrófagos M2.
RT-qPCR também manifestou que a expressão de miR-18a aumentou em macrófagos M2 em contraste com os macrófagos M0 ( P ˂ 0,05) (Fig. 1f).
MiR-18a é altamente expresso e TGFBR3 é mal expresso em células NPC
O site de bioinformática (miRanda) previu os alvos potenciais de miR-18a e TGFBR3 foi considerado um alvo de miR-18a (Fig. 2a). O ensaio do gene repórter de luciferase dupla foi implementado para verificar se o miR-18a tinha como alvo 3′UTR de TGFBR3. TGFBR3-WT ou TGFBR3-MUT foram clonados no vetor pmirGLO e co-transfectados com miméticos de miR-18a ou NC em células SUNE-1 e CNE2. Os mimetizadores de miR-18a não tiveram influência na atividade de luciferase de TGFBR3 3′UTR-MUT, mas prejudicaram a de TGFBR3 3′UTR-WT em células SUNE-1 e CNE2, sugerindo que TGFBR3 era um gene alvo regulado por miR-18a (Fig. . 2b).
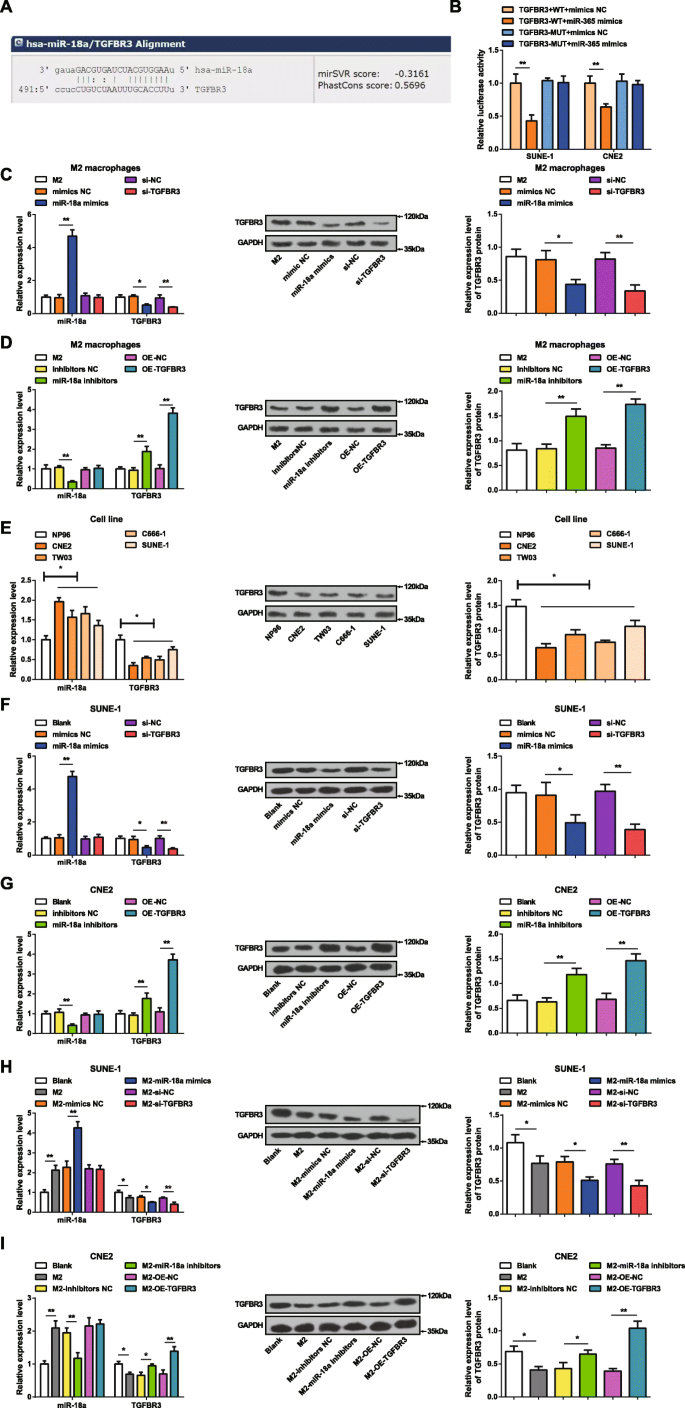
MiR-18a é altamente expresso e TGFBR3 é pouco expresso em células NPC. a miRanda previu miR-18a tendo como alvo TGFBR3. b O ensaio do gene repórter de luciferase duplo verificou miR-18a direcionado a TGFBR3. c A expressão de miR-18a foi elevada e a expressão de TGFBR3 diminuiu em macrófagos M2 transfectados com mimetizadores de miR-18a. d A expressão de miR-18a foi diminuída e a expressão de TGFBR3 foi elevada em macrófagos M2 transfectados com inibidores de miR-18a. e A expressão de miR-18a foi elevada e a expressão de TGFBR3 diminuiu em linhas de células NPC versus células NP96. f A expressão de miR-18a foi elevada e a expressão de TGFBR3 diminuiu em células SUNE-1 transfectadas com mimetizadores de miR-18a. g A expressão de miR-18a foi diminuída e a expressão de TGFBR3 foi elevada em células CNE2 transfectadas com inibidores de miR-18a. h A expressão de miR-18a foi elevada e a expressão de TGFBR3 diminuiu em células SUNE-1 co-cultivadas com macrófagos M2 transfectados com mimetizadores de miR-18a. eu A expressão de miR-18a foi diminuída e a expressão de TGFBR3 foi elevada em células CNE2 co-cultivadas com macrófagos M2 transfectados com inibidores de miR-18a; * P <0,05; ** P <0,01. Os dados de medição foram expressos como média ± desvio padrão, N =3. As comparações entre dois grupos foram analisadas por t teste. As comparações entre vários grupos foram analisadas por ANOVA de uma via, seguida pelo teste post hoc de Tukey
A eficiência de transfecção de mimetizadores de miR-18a ou inibidores de miR-18a em macrófagos M2 foi manifestada pela determinação da expressão de miR-18a e TGFBR3 em macrófagos M2 por RT-qPCR e ensaio de Western blot. Era óbvio que (Fig. 2c, d) a superexpressão de miR-18a elevou a expressão de miR-18a e reduziu a expressão de TGFBR3 em macrófagos M2. Ao contrário, a inibição de miR-18a reduziu a expressão de miR-18a e aumentou a expressão de TGFBR em macrófagos M2. Nenhuma diferença foi testemunhada na expressão de miR-18a enquanto a expressão de TGFBR3 diminuiu quando macrófagos M2 transfectados com si-TGFBR3. Nenhuma diferença foi testemunhada na expressão de miR-18a enquanto a expressão de TGFBR3 cresceu após a transfecção de OE-TGFBR3 para macrófagos M2.
A expressão de miR-18a e TGFBR3 em linhas celulares CNE2, TW03, C666-1, SUNE-1 e NP96 foi testada por RT-qPCR e ensaio de Western blot. Em células NPC, em comparação com células NP96, a expressão de miR-18a aumentou e a expressão de TGFBR3 diminuiu (Fig. 2e). Devido às células CNE2 e células SUNE-1 manifestarem-se com as maiores e menores diferenças na expressão de miR-18a de células NP96, elas foram selecionadas para os ensaios de regulação negativa e regulação positiva de miRNA, respectivamente.
Para identificar os efeitos de miR-18a e TGFBR3 nas células NPC, as células SUNE-1 foram transfectadas com mimetizadores de miR-18a ou si-TGFBR3, enquanto as células CNE2 com inibidores de miR-18a ou OE-TGFBR3. O ensaio de RT-qPCR e Western blot demonstrou que miR-18a imita a expressão elevada de miR-18a e a expressão reduzida de TGFBR3 em células SUNE-1. A transfecção de si-TGFBR3 não teve efeito na expressão de miR-18a enquanto reduziu a expressão de TGFBR3 em células SUNE-1. Os inibidores de miR-18a diminuíram a expressão de miR-18a e elevaram a expressão de TGFBR3 em células CNE2. A transfecção de OE-TGFBR3 em células CNE2 não afetou a expressão de miR-18a, mas elevou a expressão de TGFBR3 (Fig. 2f, g).
Para explorar os efeitos de miR-18a de macrófagos M2 em células NPC, miR-18a mimetiza ou macrófagos M2 transfectados com si-TGFBR3 ou inibidores de miR-18a ou macrófagos M2 transfectados com OE-TGFBR3 foram co-cultivados com SUNE-1 ou células CNE2 na câmara Transwell, respectivamente. O ensaio de RT-qPCR e Western blot testou a expressão de miR-18a e TGFBR3 em células SUNE-1 ou CNE2. Células SUNE-1 co-cultivadas com macrófagos M2 não transfectados ou mimetizadores de miR-18a transfectados demonstrados com expressão aumentada de miR-18a e expressão de TGFBR3 degradada. Nenhuma diferença foi observada na expressão de miR-18a e expressão de TGFBR3 reduzida em células SUNE-1 que foram co-cultivadas com macrófagos M2 transfectados com si-TGFBR3 (Fig. 2h). Após a co-cultura com macrófagos M2 não transfectados, as células CNE2 foram caracterizadas por aumento da expressão de miR-18a e diminuição da expressão de TGFBR3. No entanto, expressão reduzida de miR-18a e expressão elevada de TGFBR3 apresentadas em células CNE2 que foram previamente co-cultivadas com macrófagos M2 transfectados com inibidores de miR-18a. Nenhuma diferença foi reconhecida na expressão de miR-18a, e a expressão de TGFBR3 aumentou em células CNE2 co-cultivadas com macrófagos M2 transfectados com OE-TGFBR3 (Fig. 2i).
miR-18a de Macrófagos M2 promove a viabilidade celular NPC e capacidade de formação de colônias
O ensaio MTT e o ensaio de formação de colônia foram aplicados para identificar os impactos de miR-18a e TGFBR3 na viabilidade e capacidade de formação de colônias de células SUNE-1 e células CNE2. mimetizadores de miR-18a, inibidores de miR-18a, si-TGFBR3 ou OE-TGFBR3 foram transfectados em células SUNE-1 ou células CNE2. Foi indicado que em células SUNE-1, a regulação positiva de miR-18a ou a regulação negativa de TGFBR3 aumentaram a viabilidade celular e o número de colônias elevado (Fig. 3a, e). Em células CNE2, a viabilidade celular foi prejudicada e o número de colônias foi reduzido pela inibição de miR-18a ou superexpressão de TGFBR3 (Fig. 3b, f).
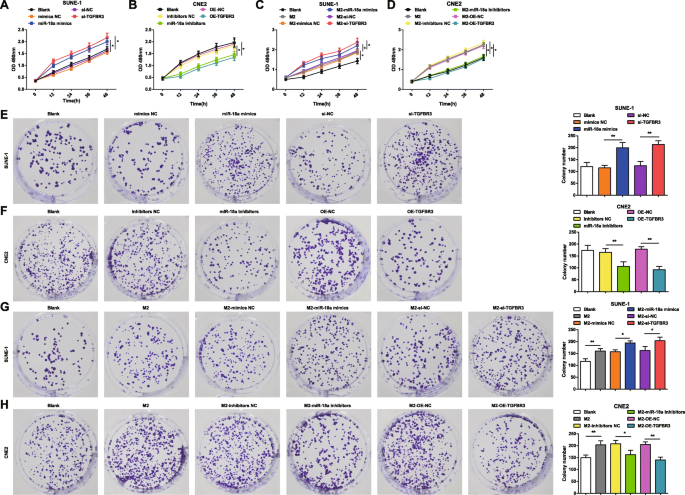
miR-18a de macrófagos M2 induz a viabilidade das células NPC e a capacidade de formação de colônias. a miR-18a mimetiza ou si-TGFBR3 aumentou a viabilidade de células SUNE-1. b Os inibidores miR-18a ou OE-TGFBR3 diminuíram a viabilidade das células CNE2. c A co-cultura com macrófagos M2 transfectados com mimetizadores de miR-18a ou si-TGFBR3 aumentou a viabilidade de células SUNE-1. d A co-cultura com inibidores de miR-18a ou macrófagos M2 transfectados com OE-TGFBR3 diminuiu a viabilidade das células CNE2. e miR-18a imita ou si-TGFBR3 aumentou o número de colônias de células SUNE-1. f Os inibidores miR-18a ou OE-TGFBR3 diminuíram o número de colônias de células CNE2. g A co-cultura com macrófagos M2 transfectados com mimetizadores de miR-18a ou si-TGFBR3 aumentou o número de colônias de células SUNE-1. h A co-cultura com inibidores de miR-18a ou macrófagos M2 transfectados com OE-TGFBR3 diminuiu o número de colônias de células CNE2; * P <0,05; ** P <0,01. Os dados de medição foram expressos como média ± desvio padrão, N =3. As comparações entre vários grupos foram analisadas por ANOVA de um fator, seguido pelo teste post hoc de Tukey
Para examinar os efeitos de miR-18a de macrófagos M2 na viabilidade e capacidade de formação de colônias de células NPC, miR-18a mimetiza, inibidores de miR-18a, si-TGFBR3-, ou macrófagos M2 transfectados com OE-TGFBR3 foram co-cultivados com células SUNE-1 ou células CNE2 na câmara Transwell. O ensaio de formação de colônias e MTT exibiu que células SUNE-1 co-cultivadas com macrófagos M2 não transfectados ou transfectados por mimetizadores de miR-18a ou transfectados com si-TGFBR3 foram apresentados com viabilidade celular reforçada e número aumentado de colônias (Fig. 3c, g) .
Células CNE2 co-cultivadas com macrófagos M2 não transfectados foram destacadas com viabilidade celular fortalecida e colônias aumentadas. No entanto, células CNE2 co-cultivadas com macrófagos M2 transfectados com inibidores miR-18a ou transfectados com OE-TGFBR foram exibidas com viabilidade celular diminuída e colônias diminuídas (Fig. 3d, h).
miR-18a de Macrófagos M2 promove invasão de células NPC e habilidades de migração
Para uma melhor compreensão de como miR-18a e TGFBR3 afetaram a migração e invasão de células NPC, o teste de raspagem e o ensaio Transwell foram implementados. Os resultados revelaram que as células SUNE-1 transfectadas com mimetizadores de miR-18a ou si-TGFBR3 foram caracterizadas por aumento da distância de migração celular e células de invasão (Figs. 4a e 5a).
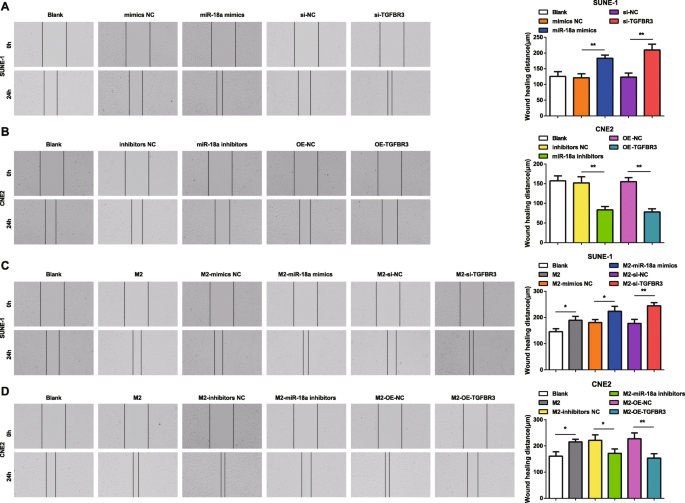
miR-18a de macrófagos M2 promove a capacidade de migração de células NPC. a miR-18a imita ou si-TGFBR3 aumentou a migração de células SUNE-1. b miR-18a inhibitors or OE-TGFBR3 decreased migration of CNE2 cells. c Co-culture with miR-18a mimics- or si-TGFBR3-transfected M2 macrophages increased migration of SUNE-1 cells. d Co-culture with miR-18a inhibitors or OE-TGFBR3-transfected M2 macrophages decreased migration of CNE2 cells; *P <0.05; **P <0.01. Measurement data were expressed as mean ± standard deviation, N =3. Comparisons among multiple groups were analyzed by one-way ANOVA, followed by Tukey’s post hoc test
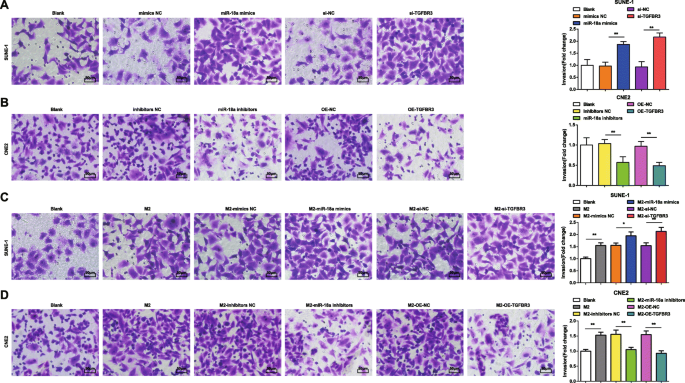
miR-18a from M2 macrophages promotes NPC cell invasion ability. a miR-18a mimics or si-TGFBR3 increased invasion of SUNE-1 cells. b miR-18a inhibitors or OE-TGFBR3 decreased invasion of CNE2 cells. c Co-culture with miR-18a mimics- or si-TGFBR3-transfected M2 macrophages increased invasion of SUNE-1 cells. b Co-culture with miR-18a inhibitors or OE-TGFBR3-transfected M2 macrophages decreased invasion of CNE2 cells; *P <0.05; **P <0.01. Measurement data were expressed as mean ± standard deviation, N =3. Comparisons among multiple groups were analyzed by one-way ANOVA, followed by Tukey’s post hoc test
In CNE2 cells transfected with miR-18a inhibitors or OE-TGFBR3, the reductions appeared in cell migration distance and invasion cells (Figs. 4b and 5b).
How miR-18a from M2 macrophages influenced invasion and migration abilities of NPC cells were deciphered by miR-18a mimics-, miR-18a inhibitors-, si-TGFBR3-, or OE-TGFBR3-transfected M2 macrophages co-culturing with SUNE-1 or CNE2 cells. The results highlighted that SUNE-1 cells co-cultured with untransfected, or miR-18a mimics-transfected or si-TGFBR3-transfected M2 macrophages were manifested with increased cell migration distance and invasion cells (Figs. 4c and 5c).
Both cell migration distance and invasion cells increased in CNE2 cells co-cultured with untransfected M2 macrophages. Upon co-culture with miR-18a inhibitors-transfected or OE-TGFBR3-transfected M2 macrophages, CNE2 cells were showed with reduced migration distance and invasion cells (Figs. 4d and 5d).
miR-18a from M2 Macrophages Arrests Fewer NPC Cell at G0/G1 Phase and Suppresses Apoptosis
Cell cycle distribution and apoptosis were tested by flow cytometry to stratify the effects of miR-18a and TGFBR3 on NPC cells. It was indicated that transfection of miR-18a mimics or si-TGFBR3 reduced SUNE-1 cells arrested in the G0/G1 phase, increased cells in the S and G2/M phases, and reduced cell apoptosis rate (Figs. 6a and 7a).
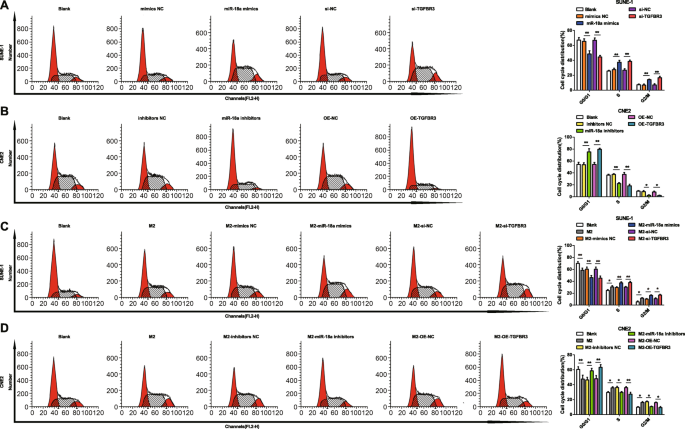
miR-18a from M2 macrophages arrests fewer NPC cell at G0/G1 phase. a miR-18a mimics or si-TGFBR3 decreased SUNE-1 cells in the G0/G1 phase, and increased SUNE-1 cells in the S and G2/M phases. b miR-18a inhibitors or OE-TGFBR3 increased CNE2 cells in the G0/G1 phase, and decreased CNE2 cells in the S and G2/M phases. c Co-culture with miR-18a mimics- or si-TGFBR3-transfected M2 macrophages decreased SUNE-1 cells in the G0/G1 phase, and increased SUNE-1 cells in the S and G2/M phases. d Co-culture with miR-18a inhibitors or OE-TGFBR3-transfected M2 macrophages increased CNE2 cells in the G0/G1 phase, and decreased CNE2 cells in the S and G2/M phases; *P <0.05; **P <0.01. Measurement data were expressed as mean ± standard deviation, N =3. Comparisons among multiple groups were analyzed by one-way ANOVA, followed by Tukey’s post hoc test
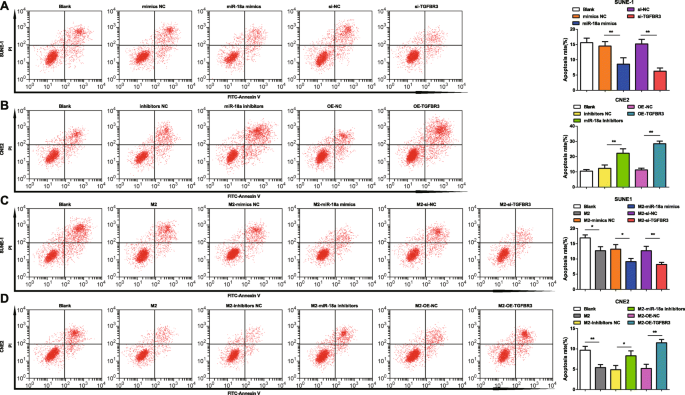
miR-18a from M2 macrophages inhibits NPC cell apoptosis. a miR-18a mimics or si-TGFBR3 decreased apoptosis of SUNE-1 cells. b miR-18a inhibitors or OE-TGFBR3 increased apoptosis of CNE2 cells. c Co-culture with miR-18a mimics- or si-TGFBR3-transfected M2 macrophages decreased apoptosis of SUNE-1 cells. d Co-culture with miR-18a inhibitors or OE-TGFBR3-transfected M2 macrophages increased apoptosis of CNE2 cells; *P <0.05; **P <0.01. Measurement data were expressed as mean ± standard deviation, N =3. Comparisons among multiple groups were analyzed by one-way ANOVA, followed by Tukey’s post hoc test
Upon transfection with miR-18a inhibitors or OE-TGFBR3, CNE2 cells in the G0/G1 phase trended toward an elevation while those in the S and G2/M phases toward a reduction, and cell apoptosis rate raised (Figs. 6b and 7b).
With the purpose to decode the mechanism of miR-18a from M2 macrophages in NPC cell cycle distribution and apoptosis, M2 macrophages transfected with miR-18a mimics, miR-18a inhibitors, si-TGFBR3, or OE-TGFBR3 were co-cultured with SUNE-1 cells or CNE2 cells in the Transwell chamber. Co-cultured with untransfected, or miR-18a mimics-transfected or si-TGFBR3-transfected M2 macrophages, reduced SUNE-1 cells were displayed in the G0/G1 phase and increased cells in the S and G2/M phases, and SUNE-1 cell apoptosis rate decreased (Figs. 6c and 7c).
Co-cultured with untransfected M2 macrophages, CNE2 cells in the G0/G1 phase reduced, cells in the S and G2/M phases increased, and apoptosis rate declined. On the contrary, co-cultured with miR-18a inhibitors-transfected or OE-TGFBR3-transfected M2 macrophages, CNE2 cells in the G0/G1 phase elevated while those in the S and G2/M phases decreased, and apoptosis rate elevated (Figs. 6d and 7d).
miR-18a from M2 Macrophages Reduces p-Smad1/t-Smad1 and Elevates p-Smad3/t-Smad3 in NPC Cells
Western blot assay detected TGF signaling pathway-related proteins in NPC cells to further explain the effects of miR-18a and TGFBR3 on TGF signaling pathway.
It was explained that transfection of miR-18a mimics or si-TGFBR3 reduced p-Smad1/t-Smad1 and elevated p-Smad3/t-Smad3 in SUNE-1 cells (Fig. 8a).
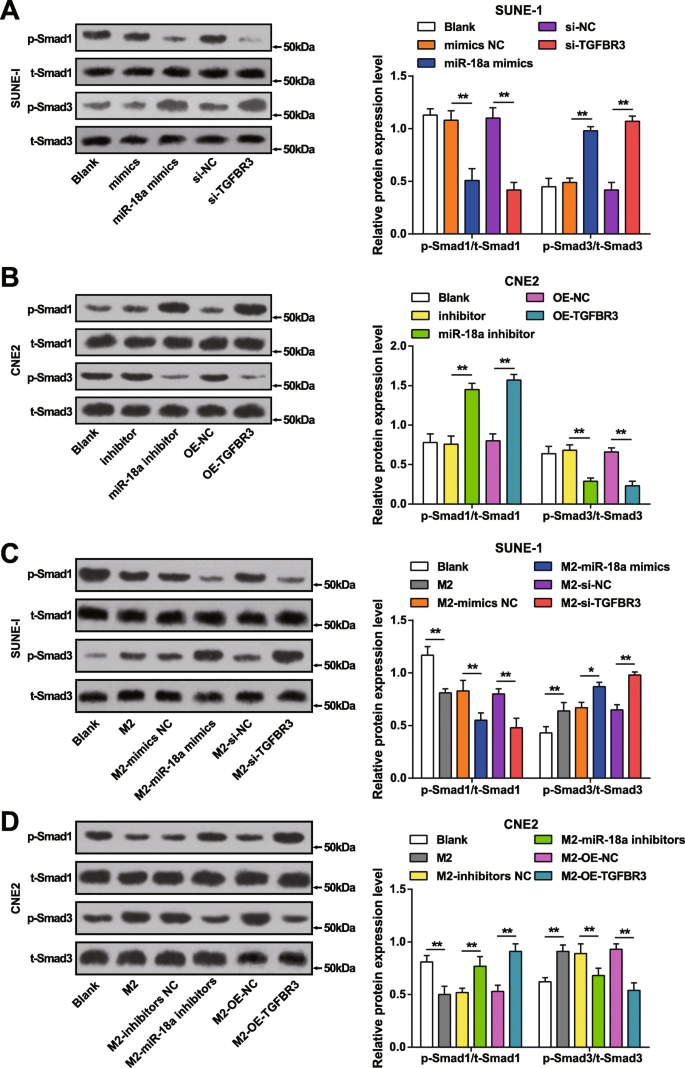
miR-18a from M2 macrophages decreases p-Smad1/t-Smad1 and increases p-Smad3/t-Smad3 in NPC cells. a miR-18a mimics or si-TGFBR3 in SUNE-1 cells decreased p-Smad1/t-Smad1 and elevated p-Smad3/t-Smad3. b miR-18a inhibitors or OE-TGFBR3 in CNE2 cells increased p-Smad1/t-Smad1 and decreased p-Smad3/t-Smad3. c Co-culture with miR-18a mimics- or si-TGFBR3-transfected M2 macrophages decreased p-Smad1/t-Smad1 and elevated p-Smad3/t-Smad3. d Co-culture with miR-18a inhibitors or OE-TGFBR3-transfected M2 macrophages increased p-Smad1/t-Smad1 and decreased p-Smad3/t-Smad3; *P <0.05; **P <0.01. Measurement data were expressed as mean ± standard deviation, N =3. Comparisons among multiple groups were analyzed by one-way ANOVA, followed by Tukey’s post hoc test
Transfection with miR-18a inhibitors or OE-TGFBR3 led to declined p-Smad3/t-Smad3 and increased p-Smad1/t-Smad1 in CNE2 cells (Fig. 8b).
miR-18a from M2 macrophages influencing TGF signaling pathway in NPC cells was determined by Western blot assay through testing TGF signaling pathway-related proteins in SUNE-1 cells and CNE2 cells which had co-cultured with miR-18a mimics-, miR-18a inhibitors-, si-TGFBR3-, or OE-TGFBR3-transfected M2 macrophages in the Transwell chamber.
SUNE-1 cells co-cultured with untransfected, miR-18a mimics-transfected, or si-TGFBR3-transfected M2 macrophages were manifested with reduced p-Smad1/t-Smad1 and incremental p-Smad3/t-Smad3 (Fig. 8c).
Co-cultured with untransfected M2 macrophages, CNE2 cells trended toward declined p-Smad1/t-Smad1 and elevated p-Smad3/t-Smad3. In an opposite way, CNE2 cells were demonstrated with increased p-Smad1/t-Smad1 and decreased p-Smad3/t-Smad3 when co-cultured with miR-18a inhibitors-transfected or OE-TGFBR3-transfected M2 macrophages (Fig. 8d).
miR-18a from M2 Macrophages Induces Tumor Growth in Nude Mice with NPC
Tumor xenografts were conducted on nude mice to further elucidate the impacts of miR-18a and TGFBR3 on tumor growth of NPC.
It was indicated that injected with miR-18a mimics-transfected or si-TGFBR3-transfected SUNE-1 cells, mice were manifested with enlarged tumor volume and heavier tumor weight (Fig. 9a).
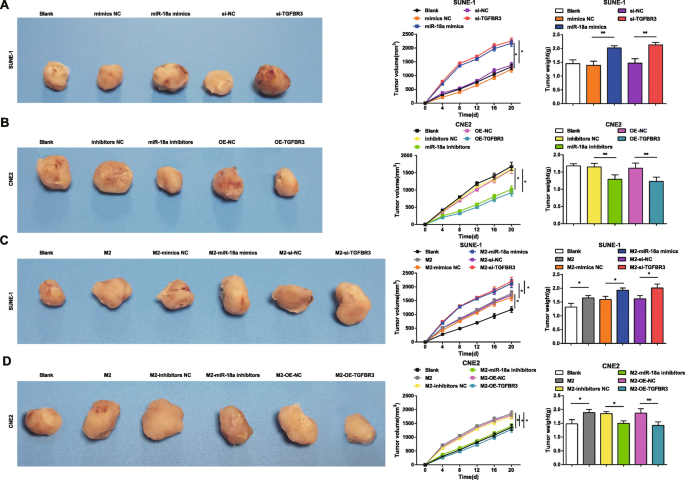
miR-18a from M2 macrophages promotes tumor growth in nude mice with NPC. a miR-18a mimics or si-TGFBR3 in SUNE-1 cells increased tumor volume and weight. b miR-18a inhibitors or OE-TGFBR3 in CNE2 cells decreased tumor volume and weight. c Co-culture with miR-18a mimics- or si-TGFBR3-transfected M2 macrophages increased tumor volume and weight. d Co-culture with miR-18a inhibitors or OE-TGFBR3-transfected M2 macrophages decreased tumor volume and weight; *P <0.05; **P <0.01. Measurement data were expressed as mean ± standard deviation, three nude mice in each group. Comparisons among multiple groups were analyzed by one-way ANOVA, followed by Tukey’s post hoc test
Reduced tumor volume and weight were presented in mice with injection of miR-18a inhibitors-transfected or OE-TGFBR3-transfected CNE2 cells (Fig. 9b).
Tumor growth was observed in mice which had injected with miR-18a mimics-, miR-18a inhibitors-, si-TGFBR3-, or OE-TGFBR3-transfected M2 macrophages to illustrate the mechanism of miR-18a from M2 macrophages in NPC.
After co-culture with untransfected, miR-18a mimics-transfected or si-TGFBR3-transfected M2 macrophages, SUNE-1 cells were injected into mice and mice were observed with larger tumor volume and heavier tumor weight (Fig. 9c).
CNE2 cells were co-cultured with M2 macrophages and injected into mice with the results suggesting growing tumor volume and weight. Both tumor volume and weight were inclined to reduce when mice were injected with CNE2 cells which had co-cultured with miR-18a inhibitors-transfected or OE-TGFBR3-transfected M2 macrophages (Fig. 9d).
Discussion
NPC refers to a polygenic disease threatened by a wide range of factors [21]. MiRNAs are previously implied to participate in the pathogenesis of NPC via regulation of their target genes which are indicators of cellular processes and pathways [22]. Concretely, miR-18a advances NPC progression by miRNA biogenesis impairing [6]. Given that this study goes forward to decipher the combined interactions of miR-18a from M2 macrophages and TGFBR3 in NPC with the conclusion elucidating that miR-18a from M2 macrophages stimulates NPC progression via TGFBR3 inhibition (Fig. 10).
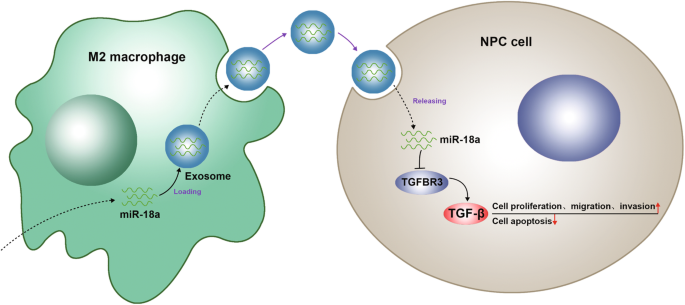
Schematic representation of macrophage-derived exosomal miR-18a in NPC and the involvement of TGFBR3-mediated TGF-β signaling pathway
At the start of this study, macrophages are stimulated by IL-4 to differentiate to M2 macrophages which are found to enrich the expression of miR-18a. As we know, during macrophage polarization, miRNA’s expression was altered [23]. In addition, M2 polarization enriches genes which are involved in the cell cycle and metabolic processes, and the M2 phenotype is conducive to tumor growth and angiogenesis in neoplastic tissues [24]. Based on the M2 macrophages-enriched miR-18a, a series of experiments were successfully conducted.
Initially, our study has uncovered that miR-18a is highly expressed while TGFBR3 is poorly expressed in NPC cells. Drawn from a previous study, it is concluded that miR-18a is overexpressed in NPC tissues with its association with lymph node metastasis and clinical stage [5]. Besides that, miR-17-92 cluster members including miR-18a are documented to be overexpressed in NPC tissues [25]. Furthermore, upregulated miR-18a is reported to demonstrate in NPC tissues which is connected with tumor node metastasis stage and tumor size [4]. Experimentally, except for the downregulated TGFBR3 in tongue squamous cell carcinoma [26], there has been another study depicting reduced TGFBR3 in clear-cell renal cell carcinomas accompanied by unwanted prognosis [27]. Anyhow, the results in this study are consistent with these study findings to some extent.
In order to explore the roles of miR-18a and TGFBR3 in NPC cell progression, we have conducted a series of assays with the results indicating that upregulated miR-18a or downregulated TGFBR3 triggers NPC cell progression while miR-18a repression or TGFBR3 elevation has the opposite effects on NPC cells. Widely, suppression of miR-18a is evidenced to hamper cell progression in malignancies including ovarian cancer, colitis-associated colorectal cancer, and hepatocellular carcinoma [28,29,30]. Narrowly, an existed study has pronounced that upregulated miR-18a promotes NPC cell progression via mediation of DICER1 [6]. In addition, it is noticed that overexpressed miR-18a in NPC is believed to connect with NPC metastasis and repressed miR-18a partially contributes to better prognosis of NPC patients [31]. Lately, it is surveyed that downregulation of miR-18a is capable of discouraging NPC proliferation, invasion, and migration [4]. Additionally, a decrease in TGFBR3 expression is regarded to link with laryngeal squamous cell carcinoma (LSCC) invasion and miR-223/TGFBR3 axis regulation takes part in LSCC progression inhibition [32]. TGFBR3 elevation is documented to restrict NPC cell viability, induce apoptosis, and activate pro-apoptosis signaling pathways [14]. Previously, a study has indicated that upregulation of TGFBR3 promotes apoptosis and cells arrested in the G2/M phase, resulting in impaired cell viability and migration in salivary gland adenoid cystic carcinoma [33]. Intriguingly, it is formerly described that induction of TGFBR3 contributes to disrupt intrahepatic cholangiocarcinoma progression [34].
Despite the protective role of lowly expressed miR-18a and overexpressed TGFBR3 in NPC cell in vitro, we have performed tumor xenografts in nude mince in vivo for further verification with the results explaining that miR-18a knockdown or TGFBR3 elevation restrains tumor growth in nude mice. As demonstrated in a prior study, miR-18a-injected nude mice show with enhanced tumor growth [5] and conversely, the miR-18a antagomir-injected nude mice are displayed with suppressed tumor growth in NPC [4]. In the light of the TGFBR3 reduction in tumor growth, it is suggested that poorly expressed TGFBR3 provokes tumor formation in clear-cell renal cell carcinoma [27]. In the opposite way, an increase in TGFBR3 is recognized to hinder tumor growth in lung cancer with the presence of long non-coding RNA ADAM metallopeptidase with thrombospondin type 1 motif, 9 antisense RNA 2 elevation, and miR-223-3p suppression [35]. This study has also predicted and verified that TGFBR3 is a target gene of miR-18a. But, more studies still need to be conducted for further verification.
Conclusion
Generally speaking, this study elaborates the concrete mechanisms that miR-18a from M2 macrophages inhibits TGFBR3 expression to exacerbate the progression of NPC via TGF-β signaling pathway, the results of which is abrogated by miR-18a knockdown or TGFBR3 elevation. This study updates the therapeutic target for NPC. However, a large cohort researches are still in need for in-depth explorations.
Disponibilidade de dados e materiais
Não aplicável.
Nanomateriais
- Composto Híbrido de Sílica Nanoestruturada / Ouro-Celulose Ligado Amino-POSS via Processo Sol-Gel e Suas Propriedades
- Eficácia antitumoral aprimorada e farmacocinética de Bufalin via lipossomas PEGuilados
- Preparação de nanoesferas poliméricas impressas com íons de paládio (II) e sua remoção de paládio (II) da solução aquosa
- Nanomontagens de ácido 5-aminolevulínico-esqualeno para fotodetecção e terapia de tumor:estudos in vitro
- Promoção do crescimento celular SH-SY5Y por nanopartículas de ouro modificadas com 6-mercaptopurina e um peptídeo penetrador de neurônio
- Nanopartículas de maghemita atuam como nanozimas, melhorando o crescimento e a tolerância ao estresse abiótico em Brassica napus
- Síntese e caracterização de BiOCl modificado e sua aplicação na adsorção de corantes de baixa concentração de solução aquosa
- Material e propriedades ópticas de pontos quânticos de carbono fluorescente fabricados a partir de suco de limão via reação hidrotérmica
- Crescimento auto-propagado de MOCVD e fotoluminescência dramaticamente aprimorada de nanofios InGaAs / InP Core – Shell
- A entrega de nanopartículas magnéticas de Fe3O4 modificada de CpG inibe o crescimento do tumor e as metástases pulmonares espontâneas para aumentar a imunoterapia



